요 약
차세대염기서열분석법을 이용한 IG, TCR 유전자 재배열검사는 ALL의 MRD 측정에 매우 예민하고 특이적인 방법이다. 검사실은 백혈병 클론을 잘 대표하는 index sequence를 검출, 선정하고, MRD 검체에서 이를 검출, 정량하여 검출한계, 최저정량한계를 고려하여 해석, 보고하여야 한다. 동시에 클론 진화에 의한 새로운 클론성 재배열의 출현도 검출, 보고할 수 있어야 한다. 본 권고안은 국내 임상검사실에서 시행하고 있는 ALL 환자에서의 차세대염기서열분석 IG, TCR 유전자 재배열 검사를 이용한 MRD 검사의 결과 해석 및 보고에 있어서 고려하고 갖추어야 할 사항을 제시하여, 진료에 유용한 정보를 제공하고자 작성되었다.
Abstract
Next-generation sequencing (NGS) for detecting clonal immunoglobulin (IG) and T-cell receptor (TCR) gene rearrangements is a highly sensitive and specific method for monitoring minimal residual disease (MRD) in patients with acute lymphoblastic leukemia (ALL). Although a standardized testing method for ALL MRD monitoring using NGS IG and TCR gene rearrangement assays has been proposed, no guidelines or recommendations for interpreting and reporting these results have been established thus far. Thus, in the present study, we propose a set of guidelines for interpreting and reporting MRD-associated data using NGS IG and TCR gene rearrangement assays in patients with ALL. These guidelines include the selection and reporting of representative index sequences and interpretation of MRD analysis results based on the limit of detection and lower limit of quantitation.
미세잔존질환(minimal residual disease, MRD)이란 현미경학적 관찰과 같은 기존의 방법으로는 검출할 수 없는, 즉 5% 미만의 잔존질환을 일컫는다. 이들은 재발의 원인이 되는 것으로 생각되며, 다른 예후 인자와 비교하여도 독립적이고 가장 강력한 예후인자이다. 현미경학적 관찰보다 더욱 예민한 검사 기법을 이용해야만 검출 및 정량이 가능하여, 급성림프모구백혈병(acute lymphoblastic leukemia, ALL)에서는 10-4 혹은 0.01% (10,000개의 전체 세포 중 1개의 잔존 백혈병세포를 검출할 수 있는 수준) 또는 그보다 더 예민도가 높은 기법이 이용될 것이 추천된다. “측정 가능한 잔존질환(measurable residual disease)”이라는 의미로 사용되기도 한다.
이를 검출하는 방법 중 하나는 클론성 면역글로불린(immunoglobulin, IG) 또는 T-세포 수용체(T-cell receptor, TCR) 유전자 재배열을 이용한 것이다. 림프구증식성질환의 클론성세포는 클론성 IG 또는 TCR 유전자 재배열을 가지며, 이것은 정상 세포와 구별하는 일종의 DNA 지문으로서의 역할을 한다[1, 2]. 차세대염기서열분석법(next-generation sequencing, NGS)을 이용하여 IG, TCR 유전자 재배열 분석을 시행하여 클론성 재배열(clonal rearrangement)의 존재 여부를 확인하고, 이를 정량화하는 것은 ALL을 비롯한 림프구증식성질환 환자의 미세잔존질환 측정에 매우 민감도와 특이도가 높은 방법이며[3-9], 최근 국내 임상검사실에서도 시행되고 있다.
차세대염기서열분석법 이전부터 사용되었던 EuroClonality/BIOMED-2의 IG, TCR 유전자 재배열에 대한 fragment PCR 또는 heteroduplex analysis에 대해서는 클론성 해석에 대한 가이드라인이 있었고, MRD 모니터링에 대한 가이드라인도 제시되었다[2, 10, 11]. 그러나, EuroClonality-NGS에서 ALL의 IG, TCR 유전자 재배열 MRD 검사의 시행에 대한 표준화된 검사법을 제시하였지만[4, 5, 12-14], 결과의 해석과 보고에 관한 가이드라인은 아직 제시되지 않았다.
본 권고안은 국내 임상검사실에서 시행하고 있는 ALL 환자에서의 차세대염기서열분석 IG, TCR 유전자 재배열 검사를 이용한 MRD 측정에 대한 결과해석 및 보고에 대한 방향을 제시하고자 작성되었다. 권고안을 활용할 주요 대상은 차세대염기서열분석 IG, TCR 유전자 재배열 검사를 시행하고 보고하는 의료인이며, 이 검사를 처방하고 결과를 수령하는 의료인에게도 도움이 될 수 있다. 위에서 기술한 바와 같이 이 검사의 해석과 보고에 대한 국외, 국내 문헌은 없으므로, 다음과 같은 간접적인 관련 문헌을 토대로 작성하였다. EuroClonality/BIOMED-2의 검사 표준화와 결과 해석 및 보고에 대한 가이드라인[10, 11], EuroClonality/BIOMED-2의 MRD해석에 대한 가이드라인[2], EuroClonality-NGS에서 제시한 MRD 검사에 대한 표준화와 이에 관련된 문헌[4, 5, 12, 13], 국내 검사실의 실제 검사 운용 경험[15-18], 그 외의 주요 문헌[1, 3, 6-9, 14, 19-26] 등을 참고하였다. 최종적으로 국내에서 실제로 검사를 시행하는 5개 기관 6명의 전문의의 자문을 거쳐 아래와 같이 권고안을 제시하였다. 그러나, 이미 가이드라인으로 인정된 것을 종합한 내용이 아니기 때문에, 활용 시에는 이를 염두에 두고, 각 검사실의 상황에 맞게 적용하여야 한다.
분자유전학 정량검사에 해당되는 모든 내용이 차세대염기서열분석 IG, TCR 유전자 재배열 검사를 이용한 ALL의 MRD 검출과 정량에도 동일하게 적용된다. 그중 검출한계와 최저정량한계의 설정은 performance validation의 단계이지만, 정량 결과의 해석과 보고 시 검출한계와 최저정량한계를 고려하여야 하므로 중요하다. 다만, 본 권고안은 결과의 해석 및 보고에 관한 내용을 주안점으로 하고 있으므로, 이에 대한 검증 방법 등은 각 기관에서 정해야 한다는 것만 제시하고자 한다[2, 15, 19]. 정량 PCR에서 검증된 검출한계, 최저정량한계를 적용할 수 있는 housekeeping 유전자의 복제 수(copy number)를 확인하고, 해당 검체의 housekeeping 유전자의 복제수에 따라 이 검체에서 적용 가능한 검출한계, 최저정량한계를 제시했던 것을 고려하여[2, 20], 검증된 검출한계, 최저정량한계를 적용할 수 있는 total read count를 비롯한 타 정도관리 지표와 그 목표치를 설정하고[18], 이를 만족할 경우 (i) 검출한계 미만, (ii) 검출한계 이상이나 최저정량한계 미만, (iii) 최저정량한계 이상인 경우 결과를 어떻게 해석하고 보고할 것인가를 정해야 한다.
각 검사실에서는 차세대염기서열분석 IG, TCR 유전자 재배열 검사를 이용한 ALL의 MRD 검사를 위한 index sequence 선정 및 보고에 대해 다음과 같은 사항을 정해야 한다.
Index sequence란 림프구계 클론성증식질환의 진단 또는 재발시 IG 또는 TCR 유전자의 클론성 재배열 염기서열로 IG, TCR 유전자에 대한 NGS 검사를 이용한 MRD 검사에서 검출 및 정량의 대상이 된다. 실제 환자 검사의 workflow는 ALL로 진단받은 초진 또는 재발한 환자의 검체(골수 또는 말초혈액)에서 백혈병 클론의 크기를 잘 반영하는(대표성이 있는) 클론성 재배열을 검출하는 것에서 시작하며, 이 클론성 재배열의 염기서열을 index sequence 또는 clonotype, clonosequence라고 한다.
Index sequence 선정을 위해서 검사를 시행할 대상 유전자와 primer set는 EuroClonality/BIOMED-2 consortium에서 제시한 workflow를 참고해 정할 수 있다[11].
B-lineage가 의심되는 경우 IGH 유전자만 또는 IGH와 IGK 유전자를 우선 검사하는 것을 권하고 있으며, 이들 두 유전자에서 index sequence로 사용할 만한 대표성 있는 클론성 재배열이 검출 되지 않을 경우, IGL을 검사할 것을 권하고 있다[11]. 그러나, IGK의 재배열이 있은 후에야 IGL의 재배열이 일어남을 고려할 때에[10, 11, 21], IGL 검사의 유용성은 높지 않은 것으로 알려져 있다. IGH에 대한 검사를 먼저 시행하고 index sequence의 기준에 맞는 대표성 있는 클론성 재배열을 검출하지 못할 경우 IGK에 대한 검사를 시행할 것인지, 동시에 시행할 것인지는 각 기관에서 정할 수 있다. 비용 및 시간 효과성을 고려해야 하며, 이와 함께 클론 진화(clonal evolution) 시 기존 클론(original clone) 전체 또는 일부가 사라지는 경우를 대비하여 가능하면 2개 이상의 대표성이 있는 index sequence를 이용해 모니터링하는 것이 추천되는 점도 고려해야 한다[1, 8, 9]. 따라서, 최대한 많은 수의 대표성 있는 index sequence를 얻기 위해서는 IGH에서 index sequence가 검출되는지 여부와 관계없이 IGK에 대해서도 동시에 검사하는 방안을 고려할 수도 있다.
IGH에 대해서 IGH FR1 primer set를 우선적으로 사용하며, 이를 이용해 대표성이 있는 index sequence를 얻지 못할 경우에 IGH FR2 → IGH FR3 → IGK 순으로 시행할 수도 있고[15, 22], IGH FR1 primer set를 이용해 대표성이 있는 index sequence를 얻지 못할 경우에 바로 IGK에 대해 시행할 수도 있다. 고려할 점은 IGH FR2, IGH FR3 primer set는 IGH 유전자 variable region (IGHV)의 somatic hypermutation에 의해 IGH FR1 primer set가 결합하지 못하여 클론성 재배열을 검출해내지 못할 때에 유용한데, 이 IGHV somatic hypermutation은 leukemic blast와 같은 precursor cell보다는 mature B-lymphoid neoplasm의 clonal lymphoid cell 단계에 서 주로 발생하므로[10], IGH FR1으로 대표성 있는 index sequence가 검출되지 않은 ALL 환자에서 IGH FR2, IGH FR3 primer set가 크게 유용하지 않을 수도 있다.
T-lineage가 의심되는 경우 TCRB를 먼저, 또는 TCRB와 TCRG를 같이 시행할 것을 권하고 있으며[11], 이들 유전자에서 대표성 있는 index sequence가 검출되지 않는 경우 TCRD에 대해 검사할 것을 권하고 있다. 그러나, 대부분의 T-ALL을 포함한 clonal T-cell proliferation에서 TCRB 또는 TCRG 의 클론성 재배열이 관찰되므로, TCRD 검사의 유용성은 크지 않은 것으로 예상되어[11], 실제로 잘 사용되지 않는다. TCR에서의 검사 또한 TCRB를 먼저 시행하고, index sequence가 얻어지는지 여부에 따라 TCRG를 더 검사하거나 동시에 둘 다 시행할 수도 있다[11]. IG에서와 마찬가지로 비용 및 시간효과성과 동시에 가능한 많은 index sequence를 얻는 목적 등을 적절히 고려하여 각 기관에서 검사 workflow를 정해야 한다.
마지막으로 위와 같은 workflow로 충분한 개수의 대표성 있는 index sequence를 얻지 못한 경우는 primer 부착 부위(binding site)의 돌연변이이나, primer 부착 부위 외 부위의 재배열에 의한 것일 수 있다. 이때에 cross-lineage 재배열 이용을 고려할 수 있으며[11], 이를 결과보고서에 제시할 수 있다. B-ALL의 40–90%에서 TCR의 클론성 재배열이 관찰되며, T-ALL의 20%에서 IG의 클론성 재배열이 관찰되므로[8, 11], B-ALL에서 TCR을, T-ALL에서 IG를 검사하여 대표성 있는 index sequence를 얻는 시도를 할 수 있다[16].
재배열들 중에서 상대적으로 높은 비율로 관찰되는 클론성 재배열이 index sequence인지 여부는 각 검사실의 선정 기준에 따라 판단할 수 있다. Total read count와 유전자의 종류, 다른 클론성 재배열의 조합과 상대적인 비율에 따라[15, 18, 23], 전체 read의 5% 이상을 기준으로 할 수도 있고[4, 15, 17], 3% [21], 2.5% [18]를 기준으로 하기도 한다. 이 index sequence가 백혈병 클론(leukemic clone)에 대해 얼마나 대표성이 있는 index sequence인지도 고려해야 하는데, 대표성이 높지 않다면 MRD 검사에서 환자의 클론 크기를 과소평가할 수 있기 때문이다. B-ALL에서 IGH의 클론성 재배열을 예로 들면, total read 중 clonal read의 비율이 IGH 재배열이 있는 세포 중 백혈병 클론의 비율에 얼마나 근접하는지가 해당 클론성 재배열이 백혈병 클론에 대해 얼마나 대표성이 있는 index sequence인지를 나타낸다. 가능하다면 각 검사실에서 이 근접도를 평가하기 위한 방법(예: 유세포분석을 통한 전체 세포 중 CD19-양성 세포의 비율과 비교) [18]과 기준을 설정하고, 대표성이 부족한 경우 다른 유전자(예를 들면 IGK)에 대한 검사를 시행할 것인지와 치료 모니터링 시 해당 클론성 재배열을 index sequence로 이용하였을 때에 MRD의 양을 과소평가할 수 있음을 명시할지 여부 등을 정해야 한다. 기준을 만족하는 index sequence가 여러 개 있는 경우가 있는데, IGH의 경우는 1개의 index sequence만 관찰되는 경우도 있지만, 2개의 index sequence가 관찰되는 경우도 흔하다[17]. IGK도 single clone 내에서 다수의 재배열이 있을 수 있음이 알려져 있으며, 실제로 기준을 만족하는 2개 이상의 index sequence가 흔히 관찰된다[24, 25]. IGH, IGK 모두에서 후자의 경우 index sequence들의 비율을 합하면 대표성이 높아질 수 있어, 이와 같은 경우에는 MRD 모니터링 시 두 index sequence의 양을 합산할 것을 권할 수도 있으나, 아직 이에 대한 학술적인 근거는 확립되어 있지 않다. 이는 TCRB, TCRB에도 모두 동일하게 적용할 수 있다.
Index sequence 선정 시 각 검사실에서 정하여야 할 또 한 가지는 클론성 재배열의 비율을 index sequence로 생각되는 재배열만을 할 것인지, sequencing error를 고려하여 몇 개의 mismatch가 있는 재배열을 포함한 merged sequence를 할 것인지 여부이며, 이것은 해당 sequence에 대한 위에서 언급한 대표성 여부와도 관련된다. IG의 경우는 1–2개의 mismatch는 sequencing error에 의한 것으로 판단하여 이 범위 내의 mismatch가 있는 merged sequence를 사용하기도 한다[15, 21]. TCR의 경우는 IG에 비해 다양성(diversity)이 낮기 때문에[10] 완벽한 match만을 index sequence로 판단하기도 한다[26]. 또한 IGK의 경우, 특정 유전자(또는 부위)간의 재배열(Intron-Kdel 재배열, V3D-20와 어떤 J 또는 Kdel, V3-11와 어떤 J 또는 Kdel)은 다클론(polyclonal) 재배열에서 흔히 발견되어 index sequence로 적절하지 않음이 제시되기도 하였는데[26], 그러나 이와 동시에 조합뿐 아니라 해당 sequence 자체의 특이성도 고려하여 해석하여야 한다.
또한, 만약 해당 검사실의 선정 기준을 모두 충족하지는 않으나, 백혈병 클론에서 유래되었으며, index sequence로 사용될 수 있을 것으로 추측되는 클론성 재배열을 보고할 것인지 여부도 각 검사실에서 기준과 지침을 마련하여야 한다. 예를 들면 이와 같은 클론성 재배열을 suspected index sequence로 보고하고, 추적관찰하는 것을 고려할 수도 있다.
마지막으로 다클론 재배열 중 특정 sequence가 상대적인 비율이 높아 백혈병 클론의 클론성 재배열로 오인되었을 가능성[11]과 전체 세포 중의 비율을 계산하기 위해 spiking한 양성 대조 세포(positive control cell)의 재배열[4]을 모두 배제하고, index sequence를 확정해야 한다.
Fig. 1에 index sequence 선정을 위한 검사에 대한 workflow 및 결과 해석 순서도를 제시하였다.
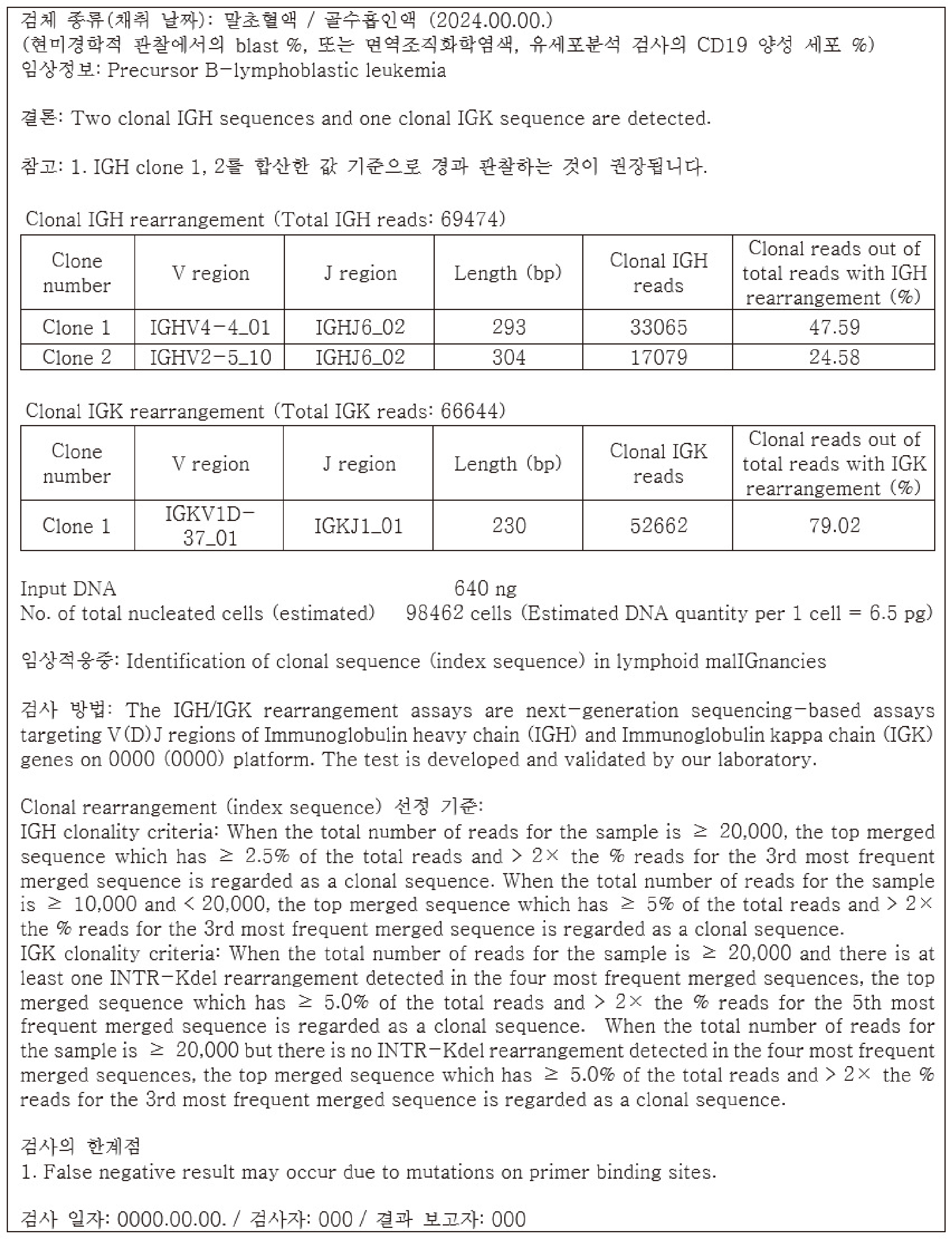
결과 보고서에는 기본 항목으로 환자의 임상 정보 및 검체 정보(종류 및 채취 시점), 넣어준 DNA (input DNA)의 양과 이를 통해 추정되는 전체 유핵 세포(total nucleated cell)의 개수, 각 유전자의 read count, index sequence로 사용될 수 있는 클론성 재배열이 보고되었는지 여부와 그 read count 및 total read에 대한 비율(%)이 포함될 수 있다. 특히, 조혈모세포이식 후를 포함하여 재발한 경우라면, 전체 IG, TCR repertoire의 변화가 있을 수 있어 임상정보의 확인이 매우 중요하다. 이에 더해 사용한 index sequence를 검출하는 데에 사용된 primer set의 종류, 각 index sequence의 V 유전자, J 유전자의 조합 및 길이(base pairs), index sequence 서열정보, 검체의 현미경학적 관찰 또는 유세포분석이나 면역조직화학염색을 통해 예측한 백혈병 세포의 비율이 추가될 수 있다. 전체 유핵 세포 중에서 클론성 재배열이 있는 세포의 비율을 환산하여 보고할 수도 있는데, 이때에 환산에 이용되는 spiking한 내부 대조(internal control)가 초진 또는 재발 환자의 검체와 같이 클론성 재배열이 있는 백혈병 클론의 비율이 높을 때에는 계산의 오류를 야기할 수도 있으므로 이를 고려하여 보고 여부를 결정하여야 한다. 만약 한 유전자에서 여러 개의 index sequence가 선정되었으며, 이들을 합산한 것이 더 대표성이 있다고 생각되면 이에 대해 언급 할 수 있다.
만약, index sequence가 하나인데 대표성이 검사실의 기준에 도달하지 못하거나, 여러 개인데도 합산하였을 때 대표성이 검사실의 기준에 도달하지 못하면, 이 index sequence로 MRD를 추적관찰할 경우 잔존질환(residual disease)을 과소평가할 수 있음을 명시할 수 있다. 마지막으로 검사 방법, index sequence를 선정하는 기준, 검사의 한계 등을 추가할 수 있다. Fig. 2에 index sequence 검출에 대한 결과보고서 예시를 제시하였다.
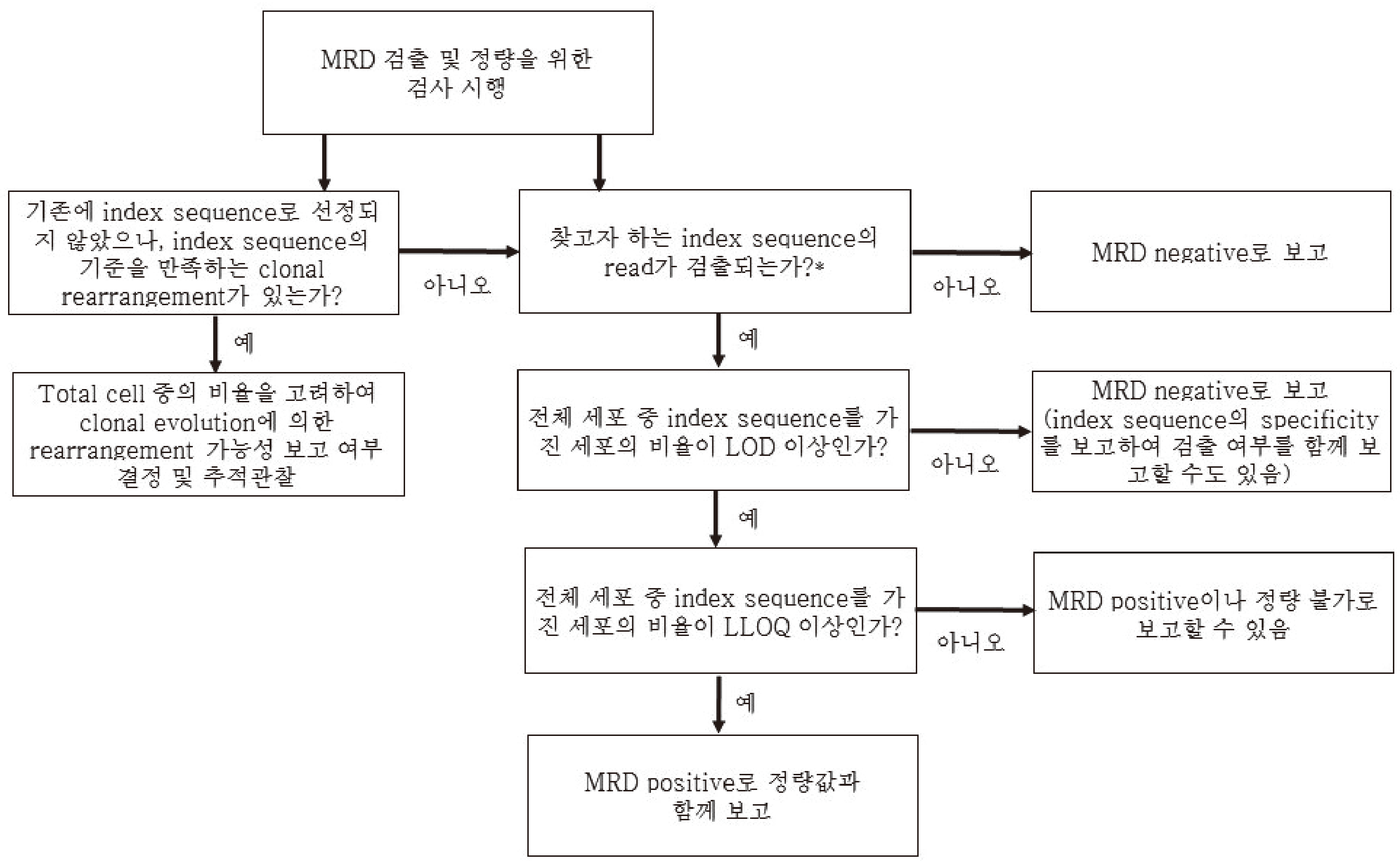
MRD의 검출과 정량은 초진 또는 재발 시 관찰된 index sequence와 동일한(또는 허용되는 mismatch 범위 내) 재배열을 찾고, 이들의 전체 유핵 세포 중에서의 분율을 보고하는 것이다. 이때에 위에서 언급한 검출한계, 최저정량한계를 고려하여야 한다. 예를 들면, 어떤 검사실에서 10-4의 예민도를 얻기 위해서는 최소 50만 read 이상이 읽어져야 함을 확인하였다고 할 때에[18], 50만 read 이상이 얻어진 환자에서 확인하고자 하는 index sequence의 read count가 0인 경우는 ‘MRD 음성’으로 보고하면 된다. 확인하고자 하는 클론성 재배열의 read 수가 ‘0’은 아니나 검출한계 미만인 경우 MRD 음성으로 보고할 것인지, 아니면 검출한계 미만으로 보고할 것인지, 검출한계 미만으로 보고하면서 sequence의 특이성을 고려하여 참고로 클론의 read count를 함께 보고할 것인지에 대해서 제시된 바는 없어 각 검사실에서 정해야 하는데, 정량 PCR의 가이드라인을 참고할 수 있다[2]. 검출한계 이상이나 최저정량한계 미만인 경우 ‘MRD 양성이나, 그 양은 최저정량한계 미만으로 정량적 수치는 제시할 수 없다’고 할 수 있는데, 역시 정량 PCR의 가이드라인을 참고할 수 있다[2]. 이때에도 참고로 클론의 read count를 함께 보고할 것인지 각 검사실에서 정해야 한다. 최저정량한계 이상인 경우에는 MRD 양성으로 보고하고, 정량 결과를 함께 보고한다. 이때에 index sequence 선정 시 두 개 이상의 index sequence 를 합산하는 것이 클론의 크기에 대한 대표성이 있다고 제시하였으면, MRD 보고서에도 이를 명시하면, 임상의가 참고할 수 있다. 만약 목표로 하는 예민도를 얻을 수 있는 충분한 total read가 확보되지 않은 경우는 목표로 하는 예민도가 보장되지 않을 수 있음을 제시하거나 또는 확보된 total read에 따라 예상되는 예민도를 제시하고, 검사의 한계를 기술할 수 있다.
클론 진화에 의해 초진 또는 재발 검체에서 클론성으로 관찰되지 않았으나 새로이 출현한 클론성 재배열이 존재할 수 있으므로, MRD를 위해 의뢰된 검체에서도 이미 보고된 index sequence의 검출 정량뿐 아니라 임상적으로 의미가 있을 수 있는 새로운 클론성 재배열을 검출, 정량, 보고하는 것이 필요하다. 초진 또는 재발검체에서의 index sequence를 찾는 것과 같은 방법으로 새로이 출현한 클론성 재배열로 의심되는 sequence를 찾아야 하는데, 주의할 점은 이들이 index sequence의 기준을 만족한다고 하더라도, 실제로는 치료에 의해 잔존 백혈병 세포 외의 해당 lineage의 세포(예를 들면 B-ALL 환자의 경우 정상 B림프구)들이 함께 감소함에 따라, 상대적으로 양이 많은 다클론 sequence 중 일부가 index sequence의 기준을 만족시켜, 이들이 새로이 출현한 클론성 재배열로 오인될 수 있다. 따라서, 초진 또는 재발 당시 확인된 다클론성 재배열들 중 새로이 출현한 클론성 재배열의 염기서열과 100% 일치하는 것이 있는지 확인하는 과정이 필요하다. 이후 이와 같은 재배열에 대해 어떤 의의를 부여할 것인지, 추후 모니터링에 포함할 것인지 여부 등을 각 검사실에서 정해야 한다. 예를 들면 해당 클론성 재배열이 total read count에서 차지하는 비율만을 고려할 것이 아니라, 전체 유핵 세포에서 차지하는 비율도 함께 고려하는 방법을 사용하기도 한다[21]. 또는, 해당 클론성 재배열이 새로운 클론성 재배열의 가능성이 있음을 제시하는 동시에, 전체 유핵 세포에서의 비율을 함께 보고하여 그 의의를 시사하는 방법이 가능하다. 위의 index sequence 선정 시와 마찬가지로 이들이 전체 유핵세포 중에 클론성 세포의 비율을 계산하기 위해 spiking한 내부대조에서 유래한 재배열 여부를 배제해야 한다. Fig. 3에 MRD 검출 및 정량을 위한 검사 결과 해석 순서도를 제시하였다.
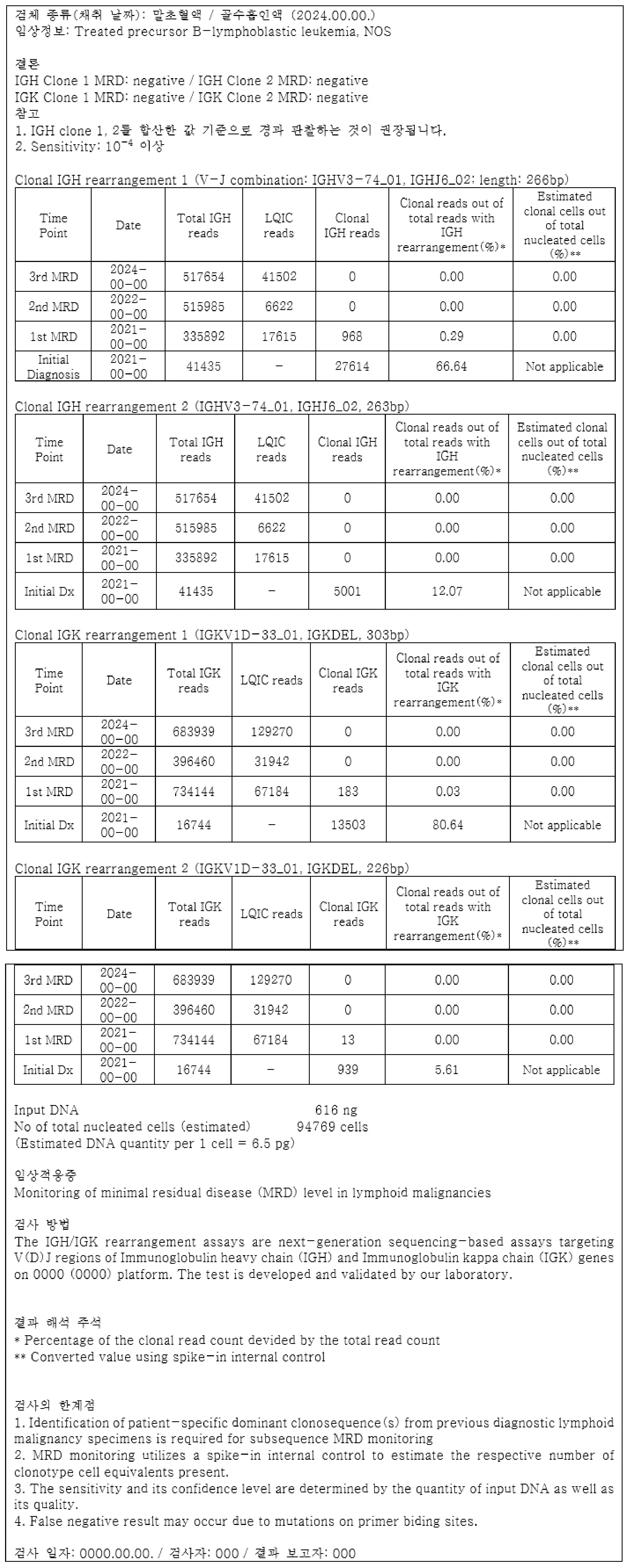
결과 보고서에는 기본 항목으로 환자의 임상 정보 및 검체 정보(종류 및 채취 시점), 넣어준 DNA의 양과 이를 통해 추정되는 total nucleated cell의 개수, 각 유전자의 read count와 각 index sequence의 read count 및 total read에 대한 비율(%) 및 전체 유핵 세포에 대한 비율(%)과 이를 통한 MRD 음성, 양성 여부가 포함될 수 있다. 이때에도 index sequence 선정 시와 마찬가지로, 조혈모세포이식 후를 포함하여 재발한 경우라면, 전체 IG, TCR repertoire의 변화가 있을 수 있어 임상정보의 확인이 매우 중요하다. 또한 검출한계 이상이나 최저정량한계 미만인 값이 얻어진 경우 ‘양성이나 정량 불가능’과 같이 보고할 수 있다. 또한 각 유전자에서 얻어진 total read에 따라 예상되는 그에 대한 예민도를 기록할 수 있으며, 특히 total read가 목표치 미만일 때에는 결과 해석을 위해 필요하다. 이에 더해 index sequence를 처음 얻었을 당시에 보고했던 정보 중 사용한 index sequence를 검출하는 데에 사용된 primer set 의 종류, 각 index sequence의 V 유전자, J 유전자의 조합 및 길이, index sequence 서열 정보를 다시 한 번 제시할 수 있다. 만약 index sequence를 처음 얻었을 때에 한 유전자에서 여러 개의 index sequence가 보고되었으며, 이들을 합산한 것이 더 대표성이 있다고 생각되면 이에 대해 언급할 수 있다. Index sequence를 처음 얻은 시점과 현재 MRD를 측정한 시점 사이에 MRD를 측정한 적이 있다면 이 결과도 표 또는 그래프 양식으로 함께 보고하여 시점에 따른 MRD의 변화를 보여줄 수 있다. 검체의 현미경학적 관찰 또는 유세포분석이나 면역조직화학염색을 통해 예측한 백혈병 세포의 비율이 추가될 수 있으나, MRD가 현미경학적 관찰 미만의 잔존질환을 의미하므로, MRD 검출을 위해 의뢰된 검사에서 이와 같은 정보의 유용성은 크지 않다. 또한 처음 index sequence를 얻었을 때 관찰되지 않았으나, MRD 검체에서 index sequence의 기준을 만족하는 클론성 재배열이 관찰될 경우 함께 보고하며, 예상되는 의의를 제시한다. 검사 방법, MRD 판정의 기준, 검사의 한계 등의 정보도 제공할 수 있다. Fig. 4에 MRD 검출과 정량에 대한 결과보고서 예시를 제시하였다.
감사의 글
대한진단유전학회의 2023년 임상지침/권고안 개발사업 연구비 지원에 감사드립니다. 또한 자문 의견을 주신 공동 연구자와 연세의대 신새암, 가톨릭의대 안아리, 서울의대 이지수, 순천향의대 김지은 교수님께 감사드립니다.
REFERENCES
1. de Haas V, Verhagen OJ, von dem Borne AE, Kroes W, van den Berg H, van der Schoot CE. 2001; Quantification of minimal residual disease in children with oligoclonal B-precursor acute lymphoblastic leukemia indicates that the clones that grow out during relapse already have the slowest rate of reduction during induction therapy. Leukemia. 15:134–40. DOI: 10.1038/sj.leu.2401970.
2. van der Velden VH, Cazzaniga G, Schrauder A, Hancock J, Bader P, Panzer-Grumayer ER, et al. European Study Group on MRD detection in ALL (ESG-MRD-ALL). 2007; Analysis of minimal residual disease by Ig/TCR gene rearrangements: guidelines for interpretation of real-time quantitative PCR data. Leukemia. 21:604–11. DOI: 10.1038/sj.leu.2404586. PMID: 17287850.
3. Kim IS. 2020; Minimal residual disease in acute lymphoblastic leukemia: technical aspects and implications for clinical interpretation. Blood Res. 55(S1):S19–26. DOI: 10.5045/br.2020.S004. PMID: 32719172. PMCID: PMC7386891.
4. Brüggemann M, Kotrová M, Knecht H, Bartram J, Boudjogrha M, Bystry V, et al. EuroClonality-NGS working group. 2019; Standardized nextgeneration sequencing of immunoglobulin and T-cell receptor gene recombinations for MRD marker identification in acute lymphoblastic leukaemia; a EuroClonality-NGS validation study. Leukemia. 33:2241–53. DOI: 10.1038/s41375-019-0496-7. PMID: 31243313. PMCID: PMC6756028.
5. Scheijen B, Meijers RWJ, Rijntjes J, van der Klift MY, Möbs M, Steinhilber J, et al. EuroClonality-NGS Working Group. 2019; Next-generation sequencing of immunoglobulin gene rearrangements for clonality assessment: a technical feasibility study by EuroClonality-NGS. Leukemia. 33:2227–40. DOI: 10.1038/s41375-019-0508-7. PMID: 31197258. PMCID: PMC6756030.
6. Giraud M, Salson M, Duez M, Villenet C, Quief S, Caillault A, et al. 2014; Fast multiclonal clusterization of V(D)J recombinations from high-throughput sequencing. BMC Genomics. 15:409. DOI: 10.1186/1471-2164-15-409. PMID: 24885090. PMCID: PMC4070559.
7. Ho C, Rothberg PG. 2021; Next-generation sequencing-based antigenreceptor gene clonality assays: will they become the clinical standard? J Mol Diagn. 23:1043–6. DOI: 10.1016/j.jmoldx.2021.07.002. PMID: 34293488.
8. Della Starza I, Chiaretti S, De Propris MS, Elia L, Cavalli M, De Novi LA, et al. 2019; Minimal residual disease in acute lymphoblastic leukemia: technical and clinical advances. Front Oncol. 9:726. DOI: 10.3389/fonc.2019.00726. PMID: 31448230. PMCID: PMC6692455. PMID: 21657571b4ce4cab9b5e2eabb05bf5e4.
9. Kim M, Park CJ. 2020; Minimal residual disease detection in pediatric acute lymphoblastic leukemia. Clin Pediatr Hematol Oncol. 27:87–100. DOI: 10.15264/cpho.2020.27.2.87. PMID: 0516a71f29084c3ba8d4bd97e95b7d63.
10. van Dongen JJ, Langerak AW, Brüggemann M, Evans PA, Hummel M, Lavender FL, et al. 2003; Design and standardization of PCR primers and protocols for detection of clonal immunoglobulin and T-cell receptor gene recombinations in suspect lymphoproliferations: report of the BIOMED-2 Concerted Action BMH4-CT98-3936. Leukemia. 17:2257–317. DOI: 10.1038/sj.leu.2403202. PMID: 14671650.
11. Langerak AW, Groenen PJ, Brüggemann M, Beldjord K, Bellan C, Bonello L, et al. 2012; EuroClonality/BIOMED-2 guidelines for interpretation and reporting of Ig/TCR clonality testing in suspected lymphoproliferations. Leukemia. 26:2159–71. DOI: 10.1038/leu.2012.246. PMID: 22918122. PMCID: PMC3469789.
12. Knecht H, Reigl T, Kotrová M, Appelt F, Stewart P, Bystry V, et al. EuroClonality-NGS Working Group. 2019; Quality control and quantification in IG/TR next-generation sequencing marker identification: protocols and bioinformatic functionalities by EuroClonality-NGS. Leukemia. 33:2254–65. DOI: 10.1038/s41375-019-0499-4. PMID: 31227779. PMCID: PMC6756032.
13. Langerak AW, Brüggemann M, Davi F, Darzentas N, van Dongen JJM, Gonzalez D, et al. EuroClonality-NGS Consortium. 2017; High-throughput immunogenetics for clinical and research applications in immunohematology: potential and challenges. J Immunol. 198:3765–74. DOI: 10.4049/jimmunol.1602050. PMID: 28416603.
14. Bystry V, Reigl T, Krejci A, Demko M, Hanakova B, Grioni A, et al. EuroClonality-NGS. 2017; ARResT/Interrogate: an interactive immunoprofiler for IG/TR NGS data. Bioinformatics. 33:435–7. DOI: 10.1093/bioinformatics/btw634.
15. Jo I, Chung NG, Lee S, Kwon A, Kim J, Choi H, et al. 2019; Considerations for monitoring minimal residual disease using immunoglobulin clonality in patients with precursor B-cell lymphoblastic leukemia. Clin Chim Acta. 488:81–9. DOI: 10.1016/j.cca.2018.10.037. PMID: 30389459.
16. Chu D, Kim M, Cho YU, Hwang SH, Jang S, Seo EJ, et al. 2023; Immunoglobulin gene rearrangement in Korean patients with B-lymphoblastic leukemia- an immunophenotype and repertoire analysis. Hemasphere. 7:e58008b2. DOI: 10.1097/01.HS9.0000968524.58008.b2. PMID: 64e7fc9adf834610b682cf90e167c3f9.
17. Shin S, Hwang IS, Kim J, Lee KA, Lee ST, Choi JR. 2017; Detection of immunoglobulin heavy chain gene clonality by next-generation sequencing for minimal residual disease monitoring in B-lymphoblastic leukemia. Ann Lab Med. 37:331–5. DOI: 10.3343/alm.2017.37.4.331. PMID: 28445014. PMCID: PMC5409014.
18. Lee JW, Kim Y, Ahn A, Lee JM, Yoo JW, Kim S, et al. 2022; Clinical implication of minimal residual disease assessment by next-generation sequencing-based immunoglobulin clonality assay in pediatric B-acute lymphoblastic leukemia. Front Oncol. 12:957743. DOI: 10.3389/fonc.2022.957743. PMID: 36185293. PMCID: PMC9521036. PMID: 980933830448449e973d16bb75c2529b.
19. Kim JJ, Park S, Cho Y, Ha J, Shin S, Lee ST. 2022; Recommendations for the verification of quantitative molecular hemato-oncology tests. Lab Med Online. 12:227–34. DOI: 10.47429/lmo.2022.12.4.227.
20. Cross NC, White HE, Colomer D, Ehrencrona H, Foroni L, Gottardi E, et al. 2015; Laboratory recommendations for scoring deep molecular responses following treatment for chronic myeloid leukemia. Leukemia. 29:999–1003. DOI: 10.1038/leu.2015.29.
21. Liu Y, Ho C, Yu W, Huang Y, Miller J, Gao Q, et al. 2024; Quantification of measurable residual disease detection by next-generation sequencingbased clonality testing in B-cell and plasma cell neoplasms. J Mol Diagn. 26:168–78. DOI: 10.1016/j.jmoldx.2023.11.009.
22. Arcila ME, Yu W, Syed M, Kim H, Maciag L, Yao J, et al. 2019; Establishment of immunoglobulin heavy (IGH) chain clonality testing by next-generation sequencing for routine characterization of B-cell and plasma cell neoplasms. J Mol Diagn. 21:330–42. DOI: 10.1016/j.jmoldx.2018.10.008. PMID: 30590126. PMCID: PMC6436112.
23. Arcila M. NGS-based clonality testing assessing clonality status, somatic hypermutation and monitoring minimum residual disease (MRD). https://www.amp.org/AMP/assets/File/education/Handout_Invivoscribe_MRD%20Final2_Arcila%20Oct102017.pdf?pass=20. Last accessed on July 2024.
24. Leenders AM, Kroeze LI, Rijntjes J, Luijks J, Hebeda KM, Darzentas N, et al. 2021; Multiple immunoglobulin κ gene rearrangements within a single clone unraveled by next-generation sequencing-based clonality assessment. J Mol Diagn. 23:1097–104. DOI: 10.1016/j.jmoldx.2021.05.002. PMID: 34020040.
25. Langerak AW, Nadel B, De Torbal A, Wolvers-Tettero IL, van Gastel-Mol EJ, Verhaaf B, et al. 2004; Unraveling the consecutive recombination events in the human IGK locus. J Immunol. 173:3878–88. DOI: 10.4049/jimmunol.173.6.3878. PMID: 15356136.
26. Invivoscribe, Inc. Instructions for use: LymphoTrack MRD software v2.0.2. Document 280364, Rev. F. March 2021.
Fig. 1
A flowchart guiding index sequence selection using the next-generation sequencing results from patients with acute lymphoblastic leukemia.
*Combining multiple index sequences can improve representativeness. If an index sequence fails to meet laboratory criteria, it may underestimate disease burden.
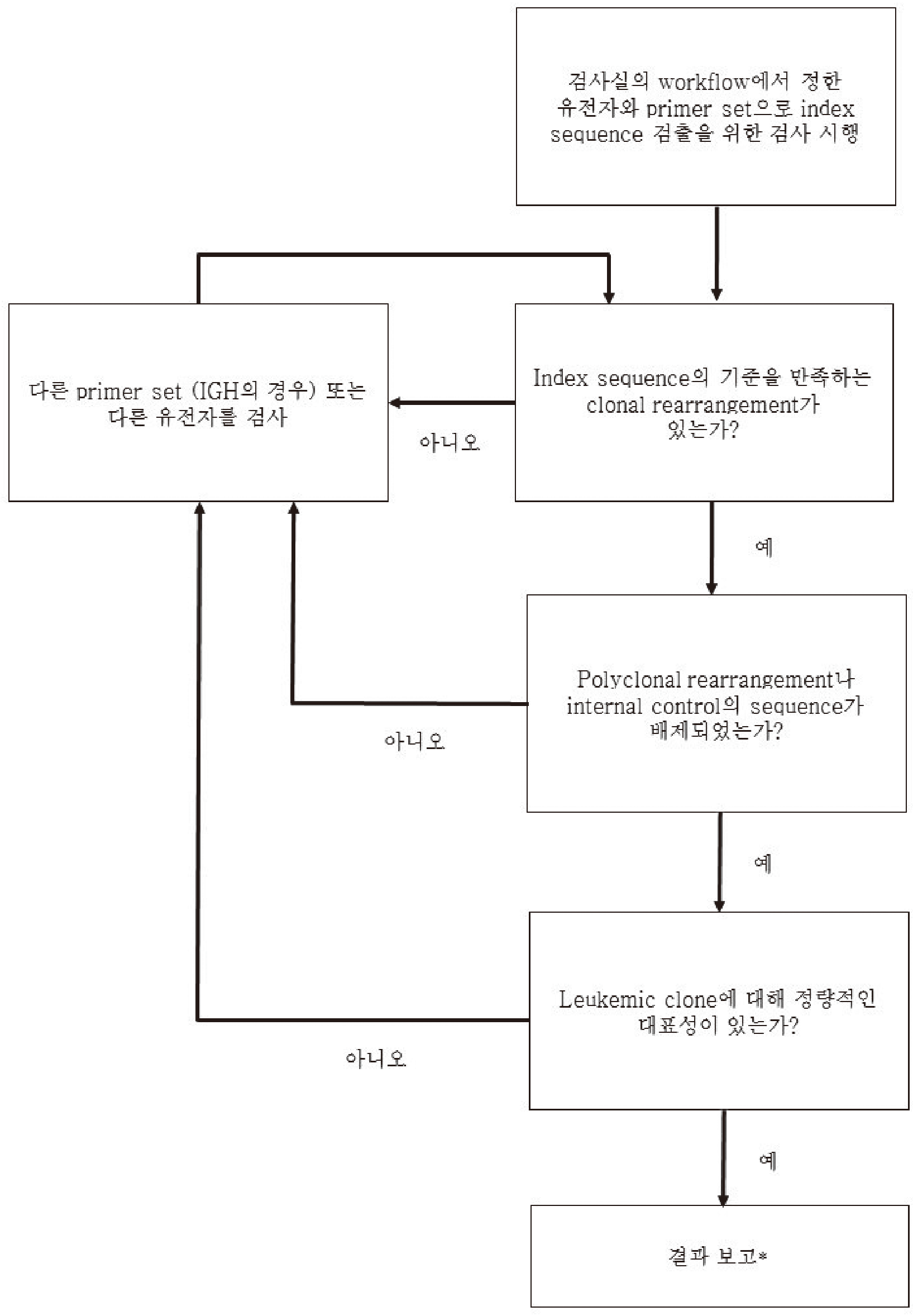
Fig. 2
A sample report of index sequence selection using the data obtained from the next-generation sequencing of immunoglobulin genes in patients with B-acute lymphocytic leukemia.




 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download