Abstract
Urinary tract infection (UTI) is a common and costly disease that affects millions of people worldwide each year. Accurate laboratory diagnosis of UTI is crucial to reduce antimicrobial resistance in UTI pathogens because of unnecessary antimicrobial use. Routine urinalysis with urine dipstick analysis and Gram staining can be used to screen for UTI. The conventional ‘gold standard’ for diagnosing UTIs involves culture-based tests. This method entails culturing the urine sample to amplify the bacteria to detectable levels, followed by biochemical and serological tests, as well as antimicrobial susceptibility tests. However, culture-based tests have the disadvantage of requiring 48-72 hours to report results owing to the time needed for bacterial growth. Therefore, various methods have been developed and are used to diagnose UTI to replace time-consuming culture tests. These methods include flow cytometry, mass spectrometry, and nucleic-acid-based diagnostic tests. This review introduces various laboratory methods used to diagnose UTI in clinical microbiology laboratories and discusses their principles and interpretation methods.
Go to : 
요로감염(Urinary Tract Infection, UTI)은 전 세계적으로 매년 수백만 명의 사람들에게 영향을 미치는 흔하고 비용이 많이 드는 질병이다[1,2]. UTI는 일반적으로 숙주의 정상 미생물총(normal flora)에 속하는 세균인 Escherichia coli, Klebsiella pneumoniae, Proteus spp., Enterobacter spp.에 의해 많이 발생한다. 특히 E. coli는 UTI를 일으키는 가장 흔한 원인균으로 전체 UTI 사례의 약 65%-75%를 차지 한다. K. pneumoniae는 두 번째로 흔한 원인균으로 전체 UTI 사례의 약 6%-8%를 차지한다[2,3]. UTI는 해부학적 감염부위에 따라 요도염(urethritis), 방광염(cystitis), 신우신염(pyelonephritis)으로 구분할 수 있으며, 감염부위에 따라 증상이 다르게 나타날 수 있다. 하부 UTI인 방광염은 일반적으로 국소 증상인 배뇨곤란, 빈뇨, 절박뇨, 통증, 그리고/또는 방광 압통을 동반한다. 한편 상부 UTI인 신우신염은 옆구리 통증과 발열, 메스꺼움 및 구토 또는 요늑각 압통과 같은 전신 감염 증상과 관련이 있으며, 방광염 증상이 있거나 없을 수 있다[4]. 증상이 없이 소변에서 세균이 검출되는 것을 무증상 세균뇨(asymptomatic bacteriuria)라고 하는데 요로 점막을 손상시키는 비뇨기과적 수술 또는 시술을 받거나 임신 중인 특별한 경우를 제외하고는 항생제 치료가 필요 없다[5].
항생제는 UTI 치료의 주요 방법이지만 예방적 또는 경험적 치료목적으로 사용한 항생제의 과용 및 오용으로 인해 내성 세균이 점점 증가하고 있다[2]. 선행연구들에 따르면 UTI 치료에서 항생제 남용이 널리 퍼져 있으며, 권장 기간을 초과하거나 권장하지 않는 항생제를 사용하는 경우가 많은 것으로 조사되었다[6,7]. 이러한 부적절한 사용은 일반적으로 사용되는 항생제에 대한 세균의 내성을 유발하여 치료 효과를 떨어뜨리게 된다[2,8]. 따라서 이러한 불필요한 항생제의 사용으로 인한 UTI 원인균에서의 항생제 내성을 줄이기 위해서 조기에 UTI에 대한 정확한 검사실적 진단이 매우 중요하다.
UTI 진단을 위한 전통적인 ‘gold standard’ 검사방법은 세균의 배양을 이용하는 것으로 요검체를 배양을 통해 검사가 가능한 만큼의 수로 균을 증식시킨 후 생화학적 및 혈청학적 검사와 항생제 감수성 검사를 시행하게 된다[9]. 하지만 배양기반 검사는 균의 증식을 위한 배양 시간이 필요하여 일반적으로 검사결과가 보고되는 데 약 48-72시간이 소요되는 단점이 있다[10]. 따라서 종종 임상 현장에서는 배양검사 결과가 나오기 전에 경험적인 항생제 치료를 시작하게 된다. 이러한 검사시간을 단축하기 위해 다양한 방법을 이용한 검사법의 개발이 활발히 이루어지고 있다. 최근에는 배양을 통한 세균의 증식을 기다리지 않고 분자생물학적 기술을 활용하여 수 시간 내에 결과를 보고하는 검사방법이나 병원의 중앙검사실이 아닌 환자의 치료현장에서 검사를 시행하여 바로 검사결과를 알 수 있는 다양한 형태의 현장검사(Point of care test, POCT)가 개발되어 평가되고 있다.
항생제 내성 세균문제가 보건의학적인 큰 이슈가 됨에 따라 UTI 같은 감염질환 의심시에 항생제 오남용을 막고 꼭 필요한 경우에만 적절한 항생제를 사용하도록 하는 항생제 올바로 쓰기(Antimicrobial stewardship, AMS)가 필수가 되었다. 이러한 AMS의 올바른 실행과 정착을 위해서 원인 감염미생물을 동정하고 항균제 감수성 분석을 시행하는 임상미생물 검사의 중요성이 점점 커지고 있다. 따라서 이번 리뷰에서는 UTI 진단을 위한 임상미생물 검사 중 전통적인 배양기반 검사 방법들뿐만 아니라 최근에 개발된 검사 방법을 소개하여 임상현장 의료진들의 검사에 대한 이해도와 활용도를 보다 높이고자 한다.
Go to : 
미생물 검사를 위해서는 적절한 요검체를 채취하는 것이 매우 중요하다. 요검체를 얻기 위해서 질, 회음부, 요도 상재균의 오염을 방지할 필요가 있다. 이를 위하여 흔히 청결채취중간뇨(clean-catch midstream urine), 직접채취카테터뇨(straight catheterized urine), 치골상부흡인뇨(suprapubic bladder aspiration) 등이 이용된다. 청결채취중간뇨는 가장 흔히 간편하게 이용되는 요검체로, 첫 소변을 조금 배출 한 뒤 중간뇨를 무균용기에 받아 채취한다. 이때 여성인 경우는 검체가 오염되지 않도록 조심해서 피부를 벌리고 요를 채취해야 한다. 직접채취카테터뇨는 카테터를 이용해 직접 방광에서 오염되지 않은 요검체를 채취할 수 있으나 반대로 요도의 미생물이 카테터를 통해 방광으로 들어갈 수 있는 위험성이 있다. 치골상부흡인뇨는 요검체를 받기 어려운 어린 소아환자나 외상환자 등에서 주로 시행되며 오염이 안된 요검체를 얻을 수 있다. 요검체는 채취 후 가급적 바로 미생물 검사실로 보내서 2시간 이내에 검사하는 것이 가장 좋다. 만일 바로 보낼 수 없을 경우에는 즉시 냉장보관하면 약 24시간 동안은 보관이 가능하다[11-13]. 냉장이 불가능한 경우에는 방부제인 boric acid가 들어있는 특별한 수송용기인 BD Vacutainer Plus Urine C&S Preservative Tube (Becton Dickinson, Franklin Lakes, NJ, USA)에 채뇨한 요검체는 냉장효과와 비슷하게 세균증식 억제 효과를 가질 수 있으며 최장 48시간까지 실온에서 보관이 가능하다[14].
기본 요분석검사는 신장과 요로계의 다양한 질환을 찾아내고 신질환의 변화를 모니터하기 위해 사용되는 간단하며 유용한 검사이다. 요분석검사는 크게 검체뇨 평가, 육안적 검사, dipstick 요분석, 요침사 현미경분석 등의 네 부분으로 구성된다. 검체뇨 평가에서는 요검체가 검사에 적절한지 평가하는 것으로 육안적으로 소변색, 투명도 및 냄새의 변화를 검사한다. Dipstick 요분석은 시약띠(reagent strip)의 화학 발색반응을 이용하여 검사를 하는 간편하고 신속한 검사이며 요침사 현미경분석은 요검체를 원침한 후 침전물 내의 세포, 원주 및 결정체를 현미경으로 관찰하는 검사이다. 이중 Dipstick 요분석은 요검체의 pH, 비중, 잠혈, 단백, 백혈구 에스테라제(Leukocyte esterase, LE), 아질산염(nitrite), 당, 케톤체, 유로빌리노겐(urobilinogen), 빌리루빈(bilirubin) 등을 검사하는데 UTI는 이들 중 Leukocyte esterase, 아질산염과 연관 되어있다[11].
LE 검사는 소변의 중성구에서 유래하는 esterase 효소를 측정하는 것으로 시약띠에 포함된 ester 화합물이 esterase에 의해 가수분해되면 보라색으로 변하게 되어 소변 내 백혈구의 존재를 간접적으로 나타내게 된다. LE 검사는 높은 민감도(97% 이상)를 보이지만, 특이도는 낮아 LE 검사가 양성이더라도 반드시 UTI를 의미하지는 않는다. LE 검사는 요로감염 초기에는 음성 결과를 보일 수 있으며, 또한 소변에 비타민 C 등의 환원제 존재 시 위음성의 가능성이 있어 해석에 주의하여야 한다[15,16].
아질산염 검사는 소변 내 아질산염의 존재를 확인하여 UTI의 가능성을 평가한다. 아질산염은 음식물을 통하여 체내로 들어온 질산염(nitrate)이 요로감염균, 주로 그람음성간균에 의해 산화되어 형성된 물질로 만일 소변 내 아질산염(nitrite)이 존재하면 시약띠의 p-arsenilic acid와 결합하여 핑크색으로 발색반응을 나타내게 된다. Dipstick 요분석검사에서 핑크색 반응이 나타나면 소변 내 의미있는 요로감염균(≥105 colony-forming units (CFU)/mL)의 존재를 의미하고 음성 반응은 무균성 소변을 의미한다. 세균이 4시간 이상 방광에 저류되어야 질산염이 아질산염으로 변화하기 때문에 저류시간이 긴 아침 첫 소변에서 양성일 확률이 높고(민감도 70%-90%), 빈뇨가 있는 UTI 초기에는 저류 시간이 짧아 음성일 수도 있다. UTI의 80%-90%에서 양성을 보이지만 회음부나 요도구 주위에 존재하는 적은 양의 세균에 의해서도 양성이 될 수 있다. 또한 실온에 수시간 방치 후 검사하면 외부 세균이 오염되어 위양성일 수 있고 소변색을 붉게 만드는 색소나 약물이 있어도 위양성을 보일 수 있다. 한편 질산염 환원 효소가 없는 세균인 그람양성구균, 녹농균, 진균 감염일 때 또는 식이에 질산염이 없을 때, 환원제인 비타민 C가 고농도로 있을 때에는 위음성을 보일 수 있다[15,16].
Dipstick 요분석의 LE 검사와 아질산염 검사는 UTI의 선별검사로서 사용되며, LE 검사와 아질산염 검사 모두 음성인 경우 UTI의 가능성이 매우 낮지만, LE 검사 또는 아질산염 검사 중 하나라도 양성인 경우 UTI의 가능성이 있어 요침사 검사, 요배양 검사 등의 추가 검사를 통해 UTI를 최종 진단하게 된다.
그람염색은 현미경으로 세균과 백혈구의 존재를 확인하기 위해 사용할 수 있는 방법으로 쉽고 간편하게 감염균에 대한 빠른 정보를 제공해준다. 요검체의 그람염색은 원침하지 않은 요 한방울을 슬라이드에 놓고 펴지 않고 말려서 염색을 시행한다. 유침렌즈(1,000×)의 배율로 관찰하여 시야당 1개 이상의 세균이 있으면 ≥105 CFU/mL의 세균뇨와 연관되며, 1개 이상의 백혈구가 존재하면 농뇨(pyuria)를 시사한다[11]. 일반적으로 그람염색을 통해 UTI의 원인균을 확인할 수 있는 민감도는 원인균마다 차이는 있지만 대략 11-80% 정도로 알려져 있다[17]. 한편 요검체가 배양검사에 적합한 검체인지 선별하는데 그람염색과 요분석(urinalysis)이 활용되기도 한다(Table 1). 요분석에서 감염을 시사하는 소견은 LE가 양성, 다형핵 백혈구(polymorphonuclear leukocyte)가 >10/μL이고 상피세포가 많지 않으며, 그람염색상에서 1-2 종류의 세균이 1,000× 시야당 1개 이상인 경우이다[12].
Criteria for determining the suitability of specimen culture tests
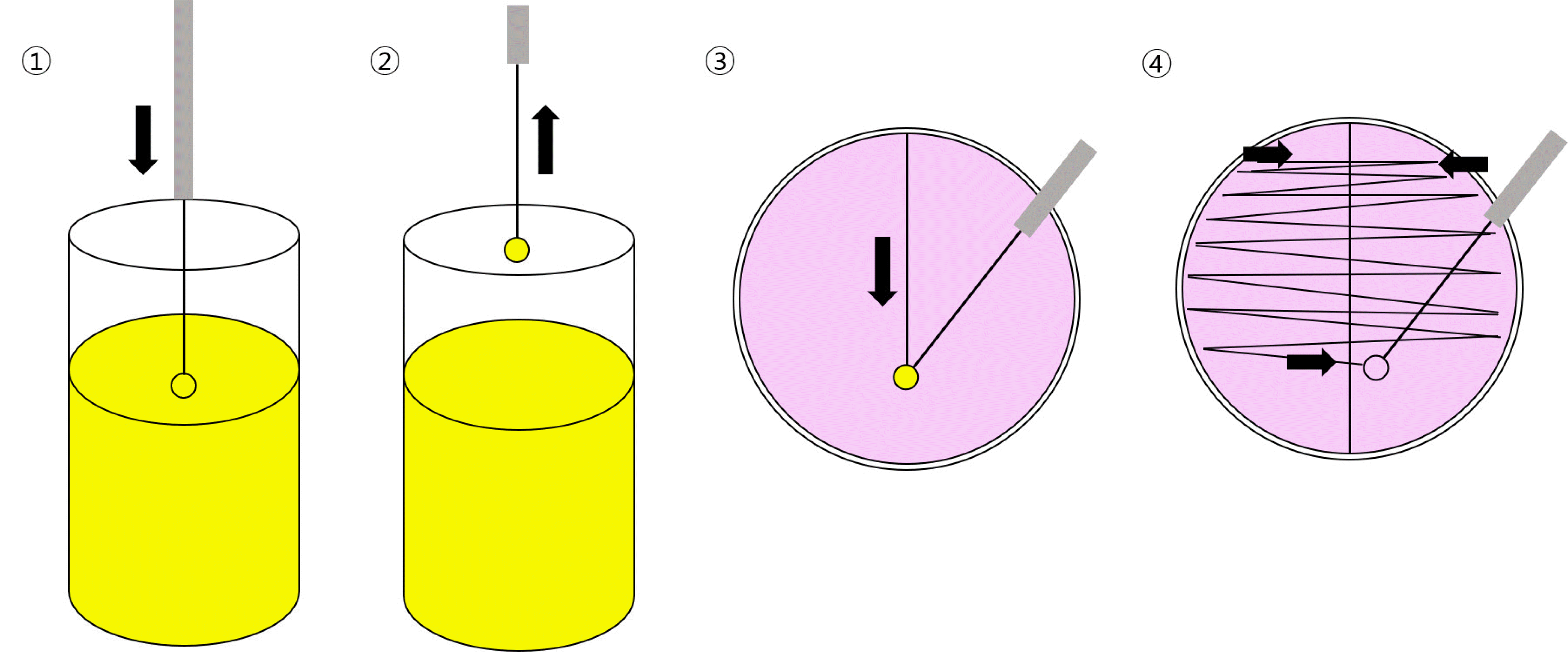
요배양검사는 해석에 있어 CFU/mL 단위의 세균수가 중요하기 때문에 정량방법으로 배양한다. 일반적으로 사용이 간편한 0.001 mL 백금이를 이용한 정량 백금이(calibrated loop)법을 사용한다(Fig. 1). 세균의 수가 적은 직접 채취 카테터뇨 검체나 치골상부흡인뇨의 경우에 0.01 mL 백금이를 이용하여 보다 많은 양의 검체 접종이 추천된다. 요배양은 5% 면양혈액배지(blood agar plate)와 MacConkey 배지에 접종하며 장내세균이 많이 자라는 경우에는 그람양성 알균을 쉽게 분리하기 위해 그람양성균의 선택배지로 Columbia colistin nalidixic acid (Columbia CNA) agar에 접종하기도 하기도 한다[18]. 접종한 배지는 35°C에서 하룻밤 배양을 하여 세균 집락수(CFU)를 확인한다. 만일 0.001 mL 백금이를 사용하여 접종한 경우 관찰되는 집락수에 1,000을 곱하여 보고를 하고, 관찰되는 집락이 없으면 ‘Less than 1,000 CFU/mL’로 보고한다.
이러한 표준 요배양검사 이외에 더 많은 양의 요검체를 사용하여 저농도의 세균을 감지하는 향상된 소변배양검사(Enhanced quantitative urine culture, EQUC)도 일부 검사실에서 사용되고 있다. EQUC는 더 많은 요검체를 사용할 뿐만 아니라 검사실에 따라서는 더 다양한 배지와 더 긴 배양시간을 이용하는 경우도 있다(Table 2). EQUC는 요배양 음성결과의 비율을 크게 줄이는 데 효과적이며, 표준 요배양 검사보다 더 민감하고 특이적이라고 보고한 연구들이 있으며 이는 특히 소아, 여성, 면역력이 약한 환자에게 중요한 의미를 가질 수 있다[19-21].
Comparison of urine cultivation protocols
또한 현장검사(POCT)로 agar paddle (dip-slide)법을 사용하여 배양할 수 있다. Agar paddle에 요에 담가서 배지표면에 요가 묻게 한 뒤 배양한 후 집락수(CFU)를 표준그림과 비교하여 요 1 mL 당의 세균 수를 추정한다. 시장에 출시된 agar paddle로는 Urotube (Becton Dickinson, Franklin Lakes, NJ, USA)와 Uriline (BioMeriux, Marcy-l’Étoile, France)가 있다. 이 후 미생물 검사실로 보내서 균동정과 항생제 감수성검사를 시행하게 된다. 하지만 이 방법은 전통적인 배양검사방법과 비교하여 상대적으로 집락수를 적게 추정하며, 두 가지 이상의 세균이 증식했을 경우 동정이 어려운 단점을 가지고 있다. 전통적인 배양검사기준으로 민감도와 특이도를 산정하였을 때 민감도는 73%, 특이도는 94%로 보고되었다[4].
요검체에서 병원체 분리 및 동정 후에는 추가적으로 24-48시간이 소요되는 항균제 감수성 검사(Antimicrobial susceptibility testing, AST)를 시행한다. AST는 일반적으로 특정 항균제 존재 하에서 세균 증식을 측정하는 표현형 검사이다. 각 병원체-항균제 조합에 대한 AST 결과는 표준화된 지침인 Clinical & Laboratory Standards Institute (CLSI), The European Committee on Antimicrobial Susceptibility Testing (EUCAST)의 가이드라인에 따라 민감성(susceptible), 중간내성(intermediate), 내성(resistant)으로 해석하여 보고한다. 세균에 대한 항균제 감수성검사에 대한 해석기준이 정리된 CLSI M100 ED34:2024에서 몇몇 항균제인 경우는 항균제명 다음에 U로 표시해 놓은 것이 있는데 이는 세균이 요검체에서 분리되었을 때에만 보고하는 경우이다. 같은 항균제라도 요검체에서 분리된 경우에는 그 기준이 달라질 수 있어 주의해서 해석하여야 한다[22]. 희석 배양, 디스크 확산법, 기울기 확산법 등과 같은 수작업으로 진행하는 AST 방법은 검체준비 단계와 16-24시간의 배양 시간을 필요로 한다[11,12].
최근에는 민감도 향상과 검사소요시간(turnaround time, TAT)을 약 10-16시간 정도로 단축시킬 수 있는 자동화 AST 장비를 이용하여 동정 및 항균제 감수성검사를 동시에 진행하고 있다. 이러한 자동화된 동정 및 자동화 검사시스템으로는 Vitek 2 (BioMeriux, Marcy-l’Étoile, France), Microscan Walkaway (Beckman Coulter, Brea, CA, USA), Phoenix Automated Microbiology System (Becton Dickinson, Franklin Lakes, NJ, USA) 등이 있다[11,12].
유세포 분석은 샘플을 작은 관을 통과시키면서 레이저로 조사하며 산란광을 측정하여 액체 내 세포를 검출하는 방법이다. 유세포 분석기는 다양한 세포 집단의 수를 계산할 수 있어 자동화장비로의 개발이 용이하고 소변 내 백혈구, 적혈구 및 세균을 빠르게 측정이 가능하여 UTI의 스크리닝 도구로서 관심이 높아지고 있다[4]. 유세포분석기반 UF-5000 분석기(Sysmex, Kobe, Japan)는 세균의 형태를 구분하는 특수 채널과 전용 소프트웨어를 탑재하여 세균을 포함한 세포의 형태를 판별할 수 있고 Gram flag이라는 기능을 통해 그람음성균과 그람양성균을 구분할 수 있다[23]. 이러한 유세포분석기반 UF-5000 분석기를 이용하여 UTI 스크리닝 검사방법으로써의 유용성을 평가한 선행연구에서 총 671건의 소변 샘플을 분석한 결과 Gram flag 기능을 통해 UTI 주요 원인 균인 그람음성균을 판별하는 양성예측도가 93%를 보여 UTI를 효과적으로 스크리닝 할 수 있었다[24]. 또한 344건의 요검체를 이용하여 UF-5000 분석기와 배양검사 결과를 비교한 연구에서는 UF-5000으로 측정한 μL 당 세균수 와 WBC 수로 배양 양성결과를 예측할 수 있었다. 기준값을 135 세균수/μL, 23 WBC/μL 로 설정하였을 때 가장 좋은 결과를 보여 양성예측도, 음성예측도가 각각 71%, 96%를 보였으며 이 결과를 활용하면 불필요한 요배양 검사를 줄일 수 있을 것이라고 하였다[25].
배양된 세균의 동정에 있어 다양한 기술을 이용한 방법이 시도되고 있다. Matrix-assisted laser desorption/ionization time of flight mass spectrometry (MALDI-TOF MS)는 세균의 단백질에 레이저를 조사하여 이온화시켜 진공관 안을 비행하는 시간 패턴을 데이터베이스에 보관된 세균의 패턴과 비교하여 세균을 동정하는 방법이다[26]. MALDI-TOF MS의 검사과정은 배양된 세균의 집락을 검체 플레이트에 바르고 그 위에 매트릭스 시약을 떨어뜨려 반응시킨 후 장비에 장착을 하면 수 분 이내에 세균의 동정이 가능하다. 최근에는 검사의 정확도가 향상됨에 따라 국내 미생물검사실에도 많이 보급이 되어 일상적인 세균 동정검사에 많이 활용되고 있다[26].
이러한 빠른 동정이 가능한 MALDI-TOF MS를 활용하여 배양 없이 직접 소변으로부터 세균을 동정하려는 연구가 진행되고 있다. 전향적인 연구에서는, 소변 샘플을 투석 여과(diafiltration) 및 원심분리(centrifugation)를 통해 농축 후 MALDI-TOF MS 분석에 적용하는 프로토콜을 제시하였는데, 전통적인 요배양 결과와 비교했을 때 민감도 67%, 특이도 100%를 보였다[27]. 요로감염 의심 환자로부터 1,000건의 요검체를 무작위 추출하여 그람염색에서 단일 형태의 세균만 관찰되는 샘플에 대해서만 MALDI-TOF 분석을 시행한 연구에서는 96.1%가 요배양검사와 일치하는 결과를 보였다[28]. 관련 연구들이 좀더 보완된다면 MALDI-TOF MS를 이용하여 세균배양 없이 요검체로 부터 직접 요로감염균의 동정이 가능할 것으로 기대된다.
분자진단기법인 PCR을 이용하여 세균을 동정하는 방법은 특정 타겟이 되는 유전자를 PCR 방법으로 검출하거나 모든 세균에 공통적으로 존재하는 16S rRNA 유전자의 DNA 염기서열 분석을 통해 종수준까지 확인하는 것이다[29]. 일반적인 배양검사는 급성 UTI를 진단하는 민감도가 약 60% 정도이며 세균의 수가 105 CFU/mL 이상이어야 임상적인 의미가 있다[19,30]. 요배양검사는 이전 항생제 투약에 의해 영향을 받으며, 요배양검사 시 주로 사용하는 Blood agar plate (BAP)나 MacConkey agar plate에서 잘 자라는 호기성 세균인 E. coli, Enterococcus, Staphylococcus species 등만 주로 검출되었으며 상기 배지에서 자라지 않는 Mycoplasma, Ureaplasma, Chlamydia trachomatis, Treponema pallidum 등은 검출이 어려웠다[19,31]. 하지만 핵산기반 검사를 이용하면 검사실 환경에서 배양이 잘되지 않는 까다로운 세균이나 일반적인 검사방법으로는 잘 동정되지 않는 세균의 검출도 가능하다[32,33].
최근에는 다중 실시간 PCR (multiplex real-time PCR)을 이용하여 UTI를 일으키는 주요 세균들을 요검체에서 동시에 검출하는 검사법이 개발되고 있다. 7개의 주요 UTI 원인균을 요검체에서 직접 검출할 수 있는 다중 실시간 PCR 겁사법의 임상적인 유용성 평가를 위한 연구에서 요배양검사와의 일치율이 98%이고 음성예측도(NPV)가 94%로 보고되어 다중 실시간 PCR 검사를 이용한 세균의 진단이 유용할 것이라고 보고하였다[34]. 또한 UTI 증상을 가진 환자들의 요검체에서 핵산을 추출하여 26종의 세균을 타겟으로 하는 Multiplex PCR로 검사를 진행하여 이를 배양검사결과와 비교하였을 때 90%의 일치율을 보였다[35].
핵산 기반 검출 방법은 다양한 장점을 가지고 있어 UTI의 진단에 유용하게 활용될 수 있다. 첫째, 다양한 유전자를 타겟으로 하여 검출을 할 수 있다. 이는 특정 유전자를 가진 세균만을 선택적으로 검출하여 진단의 정확도를 높일 수 있다는 장점을 제공한다. 둘째, 핵산 기반 검출 방법은 PCR과 같은 증폭 기술을 활용하여 아주 적은 양의 핵산도 검출할 수 있어 매우 높은 민감도로 감염 초기 단계에서도 정확한 진단을 가능하게 한다. 셋째, 핵산 기반 검출 방법은 기존의 배양 기반 검출 방법에 비해 훨씬 빠르게 결과를 제공한다. 이를 통해 환자의 치료 시작시점을 앞당기고, 적절한 항생제 선택에 도움을 줄 수 있다. 넷째, 항생제 내성과 관련된 유전자 또는 유전자변이 등을 검출하여 항생제 내성을 예측하는 데도 활용될 수 있다[4,32]. 하지만 이러한 특정 타겟을 정하여 증폭 검출하는 PCR 기반 방법들은 타겟 이외의 유전자는 검출을 할 수 없는 단점이 존재한다.
최근 들어 많은 발전을 하였고 여러 영역에서 활용되는 차세대염기서열 분석법(Next generation sequencing, NGS)을 활용하면 이런 PCR 기반 검사 방법들의 단점들을 어느 정도 극복할 수 있다. NGS방법은 검사하고자 하는 목적에 따라 특정 타겟 부위만을 증폭하여 분석하는 Amplicon sequencing과 전체 유전체를 분석하는 shotgun sequencing이 있다[36,37]. Amplicon sequencing인 경우 주로 세균에 공통적으로 존재하는 16s rRNA 유전자를 증폭하여 데이터베이스의 염기서열과 비교 분석하여 세균을 동정하게 된다[38]. 반면 shotgun sequencing은 미생물을 포함하는 검체의 모든 genome을 읽는 방법이다[39]. shotgun sequencing 방법은 primer set으로 한정하는 특정 타겟 부위만 증폭하여 분석하는 것이 아닌 전체 genome을 분석하는 것이어서, 이론적으로는 드물게 발견되는 세균, 진균 및 바이러스를 포함한 검체 내의 host genome과 함께 모든 미생물의 검출이 가능하다. 또한 데이터베이스와 비교하여 다양한 내성유전자 또는 내성과 관련된 유전자의 돌연변이의 검출도 가능하다[39]. NGS는 검사 후에 데이터의 복잡한 생물정보학적인 분석과정이 필수이고 아직 NGS 결과를 해석하기 위한 표준화된 기준이 마련되지 않은 단점이 존재한다[40]. 하지만 이러한 단점에도 불구하고 앞으로 NGS 분석법은 UTI를 포함한 다양한 감염성 질환의 진단에 매우 유용하게 활용될 것으로 전망된다.
Go to : 
UTI의 검사실적 진단은 다른 감염질환과 마찬가지로 배양검사가 ‘gold standard’ 방법이다. 하지만 배양방법은 세균을 증식시키는 과정이 필요하기에 결과를 얻기까지 많은 시간이 필요하다. 배양검사 이전에 UTI를 스크리닝하는 목적으로 요분석과 그람염색방법이 사용되고 있으며 또한 요배양에 적합한 검체인지를 판단하는 기준으로 쓰이기도 한다. 최근에는 유세포분석이나 질량분석기를 이용하여 배양과정없이 요검체에서 직접 세균을 검출하거나, 핵산기반 진단검사를 이용하여 민감하고 빠르게 원인균을 동정하는 방법이 개발되어 평가되고 있다. 이러한 검사방법들은 UTI를 조기에 진단하고 원인균에 대한 정보를 제시하여 적절한 항생제 치료가 가능케 할 것으로 기대된다.
Go to : 
REFERENCES
1. Flores-Mireles AL, Walker JN, Caparon M, Hultgren SJ. 2015; Urinary tract infections: epidemiology, mechanisms of infection and treatment options. Nat Rev Microbiol. 13:269–84. DOI: 10.1038/nrmicro3432. PMID: 25853778. PMCID: PMC4457377.
2. Hrbacek J, Cermak P, Zachoval R. 2020; Current antibiotic resistance trends of uropathogens in Central Europe: survey from a tertiary hospital urology department 2011-2019. Antibiotics (Basel). 9:630. DOI: 10.3390/antibiotics9090630. PMID: 32971752. PMCID: PMC7559630.
3. Chandra H, Singh C, Kumari P, Yadav S, Mishra AP, Laishevtcev A, et al. 2020; Promising roles of alternative medicine and plant-based nanotechnology as remedies for urinary tract infections. Molecules. 25:5593. DOI: 10.3390/molecules25235593. PMID: 33260701. PMCID: PMC7731396.
4. Fritzenwanker M, Imirzalioglu C, Chakraborty T, Wagenlehner FM. 2016; Modern diagnostic methods for urinary tract infections. Expert Rev Anti Infect Ther. 14:1047–63. DOI: 10.1080/14787210.2016.1236685. PMID: 27624932.
5. Ipe DS, Sundac L, Benjamin WH Jr, Moore KH, Ulett GC. 2013; Asymptomatic bacteriuria: prevalence rates of causal microorganisms, etiology of infection in different patient populations, and recent advances in molecular detection. FEMS Microbiol Lett. 346:1–10. DOI: 10.1111/1574-6968.12204. PMID: 23808987.
6. Durkin MJ, Keller M, Butler AM, Kwon JH, Dubberke ER, Miller AC, et al. 2018; An assessment of inappropriate antibiotic use and guideline adherence for uncomplicated urinary tract infections. Open Forum Infect Dis. 5:ofy198. DOI: 10.1093/ofid/ofy198. PMID: 30191156. PMCID: PMC6121225.
7. Fleming-Dutra KE, Hersh AL, Shapiro DJ, Bartoces M, Enns EA, File TM Jr, et al. 2016; Prevalence of inappropriate antibiotic prescriptions among US ambulatory care visits, 2010-2011. JAMA. 315:1864–73. DOI: 10.1001/jama.2016.4151. PMID: 27139059.
8. Sanchez GV, Master RN, Karlowsky JA, Bordon JM. 2012; In vitro antimicrobial resistance of urinary Escherichia coli isolates among U.S. outpatients from 2000 to 2010. Antimicrob Agents Chemother. 56:2181–3. DOI: 10.1128/AAC.06060-11. PMID: 22252813. PMCID: PMC3318377.
9. Franco-Duarte R, Černáková L, Kadam S, Kaushik KS, Salehi B, Bevilacqua A, et al. 2019; Advances in chemical and biological methods to identify microorganisms-from past to present. Microorganisms. 7:130. DOI: 10.3390/microorganisms7050130. PMID: 31086084. PMCID: PMC6560418.
10. Davenport M, Mach KE, Shortliffe LMD, Banaei N, Wang TH, Liao JC. 2017; New and developing diagnostic technologies for urinary tract infections. Nat Rev Urol. 14:296–310. DOI: 10.1038/nrurol.2017.20. PMID: 28248946. PMCID: PMC5473291.
11. Korean Society for Laboratory Medicine. 2021. Laboratory medicine. 6th ed. Panmun Education;Seoul: p. 687–9.
12. Chong Y, Lee K, Kim HS, Shin JH, Jeong SH, Yong D, et al. 2022. Diagnostic microbiology. 7th ed. Seo Heung Publishing Company;Seoul: p. 114–7.
13. Carroll KC, Pfaller MA, Landry ML, McAdam AJ, Patel R, Richter SS, et al. 2019. Manual of clinical microbiology. 12th ed. ASM Press;Washington, DC: p. 302–30. DOI: 10.1128/9781555819842.
14. Eisinger SW, Schwartz M, Dam L, Riedel S. 2013; Evaluation of the BD Vacutainer Plus Urine C&S Preservative Tubes compared with nonpreservative urine samples stored at 4°C and room temperature. Am J Clin Pathol. 140:306–13. DOI: 10.1309/AJCP5ON9JHXVNQOD. PMID: 23955448.
15. McTaggart SJ. 2005; Childhood urinary conditions. Aust Fam Physician. 34:937–41.
16. Patel HP. 2006; The abnormal urinalysis. Pediatr Clin North Am. 53:325–37. v. DOI: 10.1016/j.pcl.2006.02.004. PMID: 16716783.
17. Orellana MA, Gómez-Lus ML, Lora D. 2012; Sensitivity of Gram stain in the diagnosis of urethritis in men. Sex Transm Infect. 88:284–7. DOI: 10.1136/sextrans-2011-050150. PMID: 22308534.
18. Karah N, Rafei R, Elamin W, Ghazy A, Abbara A, Hamze M, et al. 2020; Guideline for urine culture and biochemical identification of bacterial urinary pathogens in low-resource settings. Diagnostics (Basel). 10:832. DOI: 10.3390/diagnostics10100832. PMID: 33081114. PMCID: PMC7602787.
19. Price TK, Dune T, Hilt EE, Thomas-White KJ, Kliethermes S, Brincat C, et al. 2016; The clinical urine culture: enhanced techniques improve detection of clinically relevant microorganisms. J Clin Microbiol. 54:1216–22. DOI: 10.1128/JCM.00044-16. PMID: 26962083. PMCID: PMC4844725.
20. Deen NS, Ahmed A, Tasnim NT, Khan N. 2023; Clinical relevance of expanded quantitative urine culture in health and disease. Front Cell Infect Microbiol. 13:1210161. DOI: 10.3389/fcimb.2023.1210161. PMID: 37593764. PMCID: PMC10428011.
21. Thapaliya J, Khadka P, Thapa S, Gongal C. 2020; Enhanced quantitative urine culture technique, a slight modification, in detecting under-diagnosed pediatric urinary tract infection. BMC Res Notes. 13:5. DOI: 10.1186/s13104-019-4875-y. PMID: 31900212. PMCID: PMC6942300.
22. Lewis II JS, Mathers AJ, Bobenchik AM, Bryson AL, Campeau S, Cullen SK, et al. Clinical and Laboratory Standards Institute (CLSI). 2024. CLSI M100: performance standards for antimicrobial susceptibility testing. 34th ed. CLSI;Berwyn:
23. Previtali G, Ravasio R, Seghezzi M, Buoro S, Alessio MG. 2017; Performance evaluation of the new fully automated urine particle analyser UF-5000 compared to the reference method of the Fuchs-Rosenthal chamber. Clin Chim Acta. 472:123–30. DOI: 10.1016/j.cca.2017.07.028. PMID: 28760666.
24. Wang H, Han FF, Wen JX, Yan Z, Han YQ, Hu ZD, et al. 2023; Accuracy of the Sysmex UF-5000 analyzer for urinary tract infection screening and pathogen classification. PLoS One. 18:e0281118. DOI: 10.1371/journal.pone.0281118. PMID: 36724192. PMCID: PMC9891513.
25. Enko D, Stelzer I, Böckl M, Schnedl WJ, Meinitzer A, Herrmann M, et al. 2021; Comparison of the reliability of Gram-negative and Gram-positive flags of the Sysmex UF-5000 with manual Gram stain and urine culture results. Clin Chem Lab Med. 59:619–24. DOI: 10.1515/cclm-2020-1263. PMID: 33068381.
26. Ferreira L, Sánchez-Juanes F, García-Fraile P, Rivas R, Mateos PF, Martínez-Molina E, et al. 2011; MALDI-TOF mass spectrometry is a fast and reliable platform for identification and ecological studies of species from family Rhizobiaceae. PLoS One. 6:e20223. DOI: 10.1371/journal.pone.0020223. PMID: 21655291. PMCID: PMC3105015.
27. Demarco ML, Burnham CA. 2014; Diafiltration MALDI-TOF mass spectrometry method for culture-independent detection and identification of pathogens directly from urine specimens. Am J Clin Pathol. 141:204–12. DOI: 10.1309/AJCPQYW3B6JLKILC. PMID: 24436267.
28. Burillo A, Rodríguez-Sánchez B, Ramiro A, Cercenado E, Rodríguez-Créixems M, Bouza E. 2014; Gram-stain plus MALDI-TOF MS (matrix-assisted laser desorption ionization-time of flight mass spectrometry) for a rapid diagnosis of urinary tract infection. PLoS One. 9:e86915. DOI: 10.1371/journal.pone.0086915. PMID: 24466289. PMCID: PMC3899310.
29. Chakravorty S, Helb D, Burday M, Connell N, Alland D. 2007; A detailed analysis of 16S ribosomal RNA gene segments for the diagnosis of pathogenic bacteria. J Microbiol Methods. 69:330–9. DOI: 10.1016/j.mimet.2007.02.005. PMID: 17391789. PMCID: PMC2562909.
30. Leber AL, Burnham CAD. 2023. Clinical microbiology procedures handbook. 5th ed. ASM Press;Washington, DC: p. 1–12.
31. Imirzalioglu C, Hain T, Chakraborty T, Domann E. 2008; Hidden pathogens uncovered: metagenomic analysis of urinary tract infections. Andrologia. 40:66–71. DOI: 10.1111/j.1439-0272.2007.00830.x. PMID: 18336452.
32. Aguilera-Arreola MG, Martínez-Peña MD, Hernández-Martínez F, Juárez Enriques SR, Rico Verdín B, Majalca-Martínez C, et al. 2016; Cultivation-independent approach for the direct detection of bacteria in human clinical specimens as a tool for analysing culture-negative samples: a prospective study. Springerplus. 5:332. DOI: 10.1186/s40064-016-1949-3. PMID: 27065040. PMCID: PMC4792836.
33. Domann E, Hong G, Imirzalioglu C, Turschner S, Kühle J, Watzel C, et al. 2003; Culture-independent identification of pathogenic bacteria and polymicrobial infections in the genitourinary tract of renal transplant recipients. J Clin Microbiol. 41:5500–10. DOI: 10.1128/JCM.41.12.5500-5510.2003. PMID: 14662931. PMCID: PMC309025.
34. van der Zee A, Roorda L, Bosman G, Ossewaarde JM. 2016; Molecular diagnosis of urinary tract infections by semi-quantitative detection of uropathogens in a routine clinical hospital setting. PLoS One. 11:e0150755. DOI: 10.1371/journal.pone.0150755. PMID: 26954694. PMCID: PMC4783162.
35. Wojno KJ, Baunoch D, Luke N, Opel M, Korman H, Kelly C, et al. 2020; Multiplex PCR based urinary tract infection (UTI) analysis compared to traditional urine culture in identifying significant pathogens in symptomatic patients. Urology. 136:119–26. DOI: 10.1016/j.urology.2019.10.018. PMID: 31715272.
36. Perez-Carrasco V, Soriano-Lerma A, Soriano M, Gutiérrez-Fernández J, Garcia-Salcedo JA. 2021; Urinary microbiome: yin and yang of the urinary tract. Front Cell Infect Microbiol. 11:617002. DOI: 10.3389/fcimb.2021.617002. PMID: 34084752. PMCID: PMC8167034.
37. Moustafa A, Li W, Singh H, Moncera KJ, Torralba MG, Yu Y, et al. 2018; Microbial metagenome of urinary tract infection. Sci Rep. 8:4333. DOI: 10.1038/s41598-018-22660-8. PMID: 29531289. PMCID: PMC5847550.
38. Szlachta-McGinn A, Douglass KM, Chung UYR, Jackson NJ, Nickel JC, Ackerman AL. 2022; Molecular diagnostic methods versus conventional urine culture for diagnosis and treatment of urinary tract infection: a systematic review and meta-analysis. Eur Urol Open Sci. 44:113–24. DOI: 10.1016/j.euros.2022.08.009. PMID: 36093322. PMCID: PMC9459428.
39. Almas S, Carpenter RE, Rowan C, Tamrakar VK, Bishop J, Sharma R. 2023; Advantage of precision metagenomics for urinary tract infection diagnostics. Front Cell Infect Microbiol. 13:1221289. DOI: 10.3389/fcimb.2023.1221289. PMID: 37469596. PMCID: PMC10352793.
40. Hasman H, Saputra D, Sicheritz-Ponten T, Lund O, Svendsen CA, Frimodt-Møller N, et al. 2014; Rapid whole-genome sequencing for detection and characterization of microorganisms directly from clinical samples. J Clin Microbiol. 52:139–46. DOI: 10.1128/JCM.02452-13. PMID: 24172157. PMCID: PMC3911411.
Go to : 




 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download