Abstract
Gastric cancer remains a significant disease burden in Korea, with Helicobacter pylori infections being the most crucial risk factor. With the advent of next-generation sequencing, the role of gastric microbiota in gastric cancer has attracted increasing attention. Studies have shown that the gastric microbiota of patients with gastric cancer differs in composition from that of the controls, with reduced microbial diversity. Lactic acid bacteria and oral microflora are often enriched in gastric cancer and are believed to induce chronic inflammation or promote the production of nitroso compounds. This review focuses on recent studies comparing the gastric microbiome in gastric cancer patients and controls.
위암은 전세계 다섯 번째로 많이 발생하는 암이며 세 번째로 많은 암 사망 원인이다.1 2020년 전 세계에서 백만 명이 넘은 새로운 위암 환자가 발생하였고, 769,000명이 위암으로 사망하였다. 우리나라에서는 2020년 26,662명의 위암 환자가 새로 진단받았으며, 2021년 7,249명의 환자가 위암으로 사망하였다.2 헬리코박터 파일로리는 위암의 가장 중요한 위험 인자이다. 전 세계 50% 이상의 인구가 헬리코박터 파일로리에 감염되어 있으며,3 대다수의 위암이 헬리코박터 파일로리 감염과 연관되어 있다.4,5 헬리코박터 파일로리 감염은 위 점막의 만성 위염을 유발하고, 위선을 파괴하여, 위축성 위염을 유발한다. 위암의 대다수를 차지하는 비분문부 장형 위암은 헬리코박터 파일로리 감염으로 인한 위축성 위염 및 장상피화생과 같은 전암성 병변을 거쳐 발생한다.6 그러나 위암은 헬리코박터 파일로리 감염 환자의 1-3%에서만 발생하여, 헬리코박터 파일로리 감염만으로는 위암의 발생 과정을 모두 설명하기 어렵다.7 또한, 헬리코박터 파일로리 제균 치료를 시행한 경우나 혹은 진행한 위축성 위염과 장상피화생에서 헬리코박터 파일로리 감염이 자연 소실된 경우에도 일부 환자에서는 여전히 위암이 발생한다.8 따라서 헬리코박터 파일로리 감염 이외의 다른 원인이 위암 발생에 기여할 것으로 생각되었다. 다른 위험 요인으로는 고령, 남성, 흡연, 음주, 및 염분과 질소 화합물의 과다 섭취 등이 제안되었다.9-11 헬리코박터 파일로리 감염에서 독성인자로 작용하는 CagA 양성 균주의 감염이나, 감염에 의해서 유발되는 만성 염증 및 위 점막의 후성유전체 변화가 위암 발생의 원인으로 작용한다는 보고도 있었다.12,13 최근에는 헬리코박터 파일로리 이외의 위 미생물무리 (microbiota)의 역할이 주목받고 있다.14,15 이 글에서는 최근에 보고되고 있는 위암 환자의 위 미생물무리에 관한 연구들을 살펴보고자 한다.
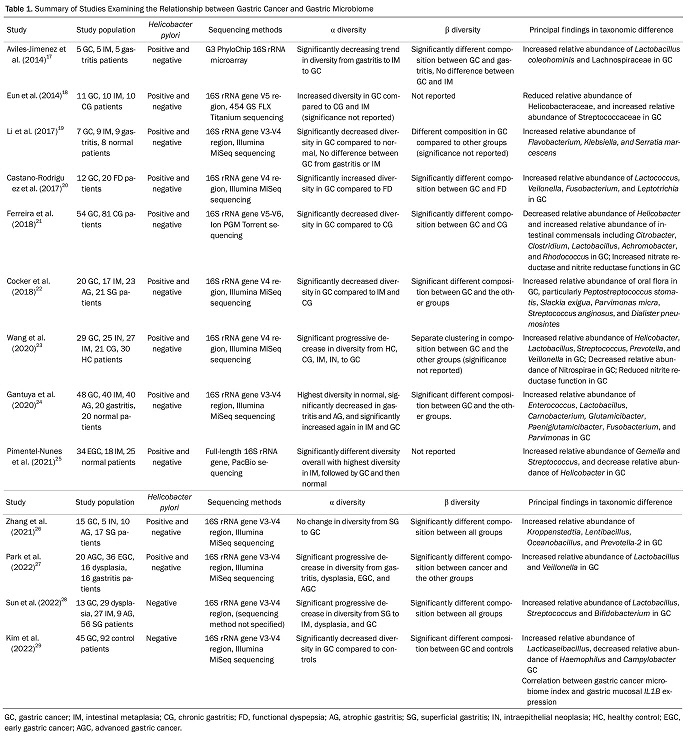
헬리코박터 파일로리가 발견되기 이전에도 위에서 미생물이 발견되었다는 보고는 있었다. 한 예로 1895년에도 위암환자의 위에서 세균과 함께 젖산이 발견되었는 보고가 있었다.6 하지만, 헬리코박터 파일로리가 위에서 관찰된 이후에도,위의 낮은 산도와 펩신의 단백질 분해 효과, 담즙산의 역류등으로 인하여 헬리코박터 파일로리 이외의 다른 균들이 실제위에서 번식할 수 있는 가능성은 거의 없다고 간주되었다. 하지만 차세대 염기서열 분석 기술이 발전하고 생물정보학과 결합하며 위장관 미생물무리에 관한 이해가 깊어지고 있다.16 주로 분변 마이크로바이옴(microbiome) 분석을 통해 시작된 연구들은, 이제 위에서도 위 미생물무리가 존재한다는 것을 명확하게 보여주고 있다.15 특히 최근 몇 년간은 위암과 위 미생물무리의 관계를 살펴보는 연구들이 활발하게 진행되었다.14-29 그러나, 연구마다 서로 다른 결과를 보고하고 있어 이들을 종합적으로 살펴볼 필요가 있다. 위암 관련 위 미생물무리에 관한 연구는 크게 세 가지 형태의 연구가 있는데, 가장많은 형태의 연구는 위암 환자와 대조군의 위 점막 조직(일부는 위액)에서 위 미생물무리를 분석하고 비교하는 환자 대조군 형태의 연구이다. 둘째는 위암 환자에서 위암 조직과 주변위 점막 조직의 위 미생물무리를 비교 분석하는 연구가 있다.셋째로 위암 환자의 위 미생물무리를 동물 실험 모델이 이식하여 분석하는 연구가 있다. 이 글에서는 위암 환자와 대조군에서 위 미생물무리를 비교한 연구 결과들을 소개하고자 한다. 이러한 연구들의 주요 내용은 Table 1에 요약되어 있다.이후 일부 동물 실험 연구에 관해서도 간략하게 살펴보고자한다.
차세대 염기서열분석의 발전으로 위 마이크로바이옴의 분석이 활성화되었는데, 이들 연구는 대부분 위 미생물무리의 전체 유전자가 아닌 16S 리보솜 RNA (rRNA) 유전자의 과변이 부위(hypervariable region)를 염기서열 분석으로 시행한연구가 대부분이다. 16S rRNA 유전자는 거의 모든 세균에존재하면서도 종별로 잘 보존되어 있기 때문에 계통발생학 연구에 사용되어 왔다.30 여기에 16S rRNA 유전자의 여러 과변이 부위가 차세대 염기서열 분석에 적합한 크기로 염기서열을 설계할 수 있다는 장점 때문에 마이크로바이옴 연구에 널리 사용되게 되었다. 따라서, 연구의 일차적인 관심은 개별 미생물과 위암의 관계보다는 위암 환자에서 위 미생물무리의 다양성이 위암이 없는 위염 환자 혹은 정상 대조군에서 위 미생물무리의 다양성과 어떻게 다른가에 있다. 미생물무리의 다양성지표는 알파 다양성과 베타 다양성 두 가지로 나눌 수 있는데, 알파 다양성에는 위암 환자에서 위 미생물무리 자체의 다양성으로 얼마나 많은 수의 미생물이 있는가(richness)와 다양한 미생물들이 미생물무리 내에서 얼마나 고르게 분포하고 있느냐(evenness) 등이 있다. 베타 다양성은 위암 환자의 위 미생물무리 구성이 대조군의 위 미생물무리 구성과 어떻게 다른가 하는 것이다. 대부분의 연구들은 위암 환자의 미생물무리가 만성 위염이나 만성 위염 및 장상피화생이 있는 대조군의 위 미생물무리와 베타 다양성에 있어서 유의하게 다른 구성을 보여주는 것으로 보고하고 있다.17,20-22,24,26-29 하지만, 위암 환자에서 위 미생물무리의 알파 다양성이 대조군에 비해서 감소하는지 증가하는지에 관해서는 연구 결과가 엇갈린다. 다수의 연구들은 위암 환자에서 위 미생물무리의 알파 다양성이 감소한다고 보고하였다.17,19,21-23,27-29 한편, 일부 연구들은 위암 환자에서 위 미생물무리의 알파 다양성이 증가한다고 보고하였다.18,20 심지어, 위암 발생의 여러 단계에 걸쳐 위 미생물무리의 알파 다양성이 감소했다가 증가하거나,24 증가했다가 감소하거나,25 차이가 없다26고 보고한 연구들도 있다. 이러한 차이는 대조군이 만성 위염 환자만 포함하는지 장상피화생 환자도 포함하는지 또는 헬리코박터 파일로리 양성 환자를 얼마나 포함하는지에 따라 영향을 받을 가능성이 있다. 헬리코박터 파일로리 감염은 위 미생물무리의 다양성을 감소시키고, 헬리코박터 파일로리 제균 치료는 위 미생물무리의 다양성을 증가시키기 때문이다.31 위암의 발생 과정에서는 위 미생물무리의 다양성이 증가하는데 영향을 미치는 인자와 감소하는데 영향을 미치는 인자가 혼재되어 있다. 위축성 위염이 진행하여 위산 분비가 감소하고 헬리코박터 파일로리 감염이 소실되는 경우에는 위 미생물무리의 다양성이 증가한다. 한편, 헬리코박터 파일로리 감염을 포함한 다양한 원인에 의한 위 점막의 만성 염증은 대부분의 미생물들에게 생존에 부적합한 환경을 제공하기 때문에 다양성이 감소하는 데 기여하게 된다.14 따라서 이러한 인자들을 구분하여 위 미생물무리 다양성의 변화를 살펴볼 필요가 있다. 예를 들면, 헬리코박터 파일로리 음성 위암 환자와 헬리코박터 파일로리 음성 만성 위염 및 장상피화생 대조군에서 위 미생물무리를 비교하였을 때는 위암 환자에서 위 미생물무리의 다양성이 대조군과 비교하여 감소하였다.29 또한, 환자의 인종, 식이, 16S rRNA 유전자 중 염기서열분석 부위와 같은 기술적인 인자들도 연구들 사이에 엇갈리는 결과의 원인이 된다. 따라서, 이러한 인자들이 위 미생물무리에 미치는 영향에 관한 연구 결과가 더 축적되어야 하며, 대규모의 다인종 환자를 포함한 전향적 연구가 위암 환자에서 위 미생물무리의 다양성에 관한 좀 더 명확한 근거를 제시해 줄 수 있을 것으로 생각된다.
위암과 관련된 위 미생물무리 연구에서 주목받은 내용 중하나는 신속 요소호기 검사나 조직 검사와 같이 확립된 진단 방법에서 헬리코박터 파일로리 음성으로 생각되었던 환자 중 상당수의 위 미생물무리에서 헬리코박터 파일로리가 발견되었다는 것이다.22,32 사실은 거의 모든 환자에서 헬리코박터 파일로리의 DNA가 발견되어, 헬리코박터 파일로리 감염 음성을 판단하는 기준을 상대 풍부도(relative abundance) 1%로 제시되기도 하였다.32 이는 전통적인 검사에서 헬리코박터 파일로리 감염 여부가 과소평가 되어 왔다는 것을 의미한다. 또한, 위축성 위염 및 장상피화생이 진행하면서 헬리코박터 파일로리 감염이 자연 소실되었다고 생각한 경우도 실제로는 헬리코박터 파일로리의 숫자가 감소할 뿐 감염이 지속되어 위암 발생 위험에 기여하였던 것으로 생각할 수 있다. 위암의 발생 과정에서 헬리코박터 파일로리의 상대 풍부도가 감소한다는 연구들이 이를 뒷받침한다.18,21,25
위암 환자의 위 미생물무리는 헬리코박터 파일로리 감염 유무에 따라 구성에 큰 차이를 보인다.33 그러나 헬리코박터 파일로리 감염 유무에 따라 다른 위 미생물들이 어떤 영향을 받는지 혹은 서로 어떤 상호작용을 하는지에 관한 근거는 부족하다. 헬리코박터 파일로리 감염 여부를 제외하면 나머지 위 미생물무리의 구성 및 다양성은 유의한 차이를 보여주지 않았다는 보고도 있다.22 생물학적 측면에서 헬리코박터 파일로리 양성 위암 환자와 음성 위암 환자에서 위 미생물무리의 역할은 다르게 해석될 여지가 있다. 헬리코박터 파일로리 양성 위암 환자에서 위 미생물무리는 헬리코박터 파일로리와 상호작용 혹은 상승작용을 통해 위암 발생에 기여하는 것으로 이해되고 있다.34 한편, 헬리코박터 파일로리 음성 위암 환자에서는 과거 헬리코박터 파일로리 감염에 의해 진행된 위축성 위염, 장상피화생, 혹은 분자생물학적 차원에서 DNA 메틸화 등 위 점막에 일어난 변화 위에 위 미생물무리가 추가로 만성 염증 등을 유발하여 위암 발생에 기여할 가능성이 있다.15,29 헬리코박터 파일로리 음성 위암에서 대조군과 차이를 보이는 위 미생물무리의 변화가 위 점막의 IL1B 발현 증가와 유의한 상관관계가 있다는 보고가 있었다.29 IL1B는 염증성 사이토카인으로 헬리코박터 파일로리 감염이 위 점막에서 IL1B의 발현을 증가시키며, 위암에서도 IL1B의 발현이 증가되어, 헬리코박터 파일로리 감염과 위암 발생을 연결하는 기전으로 이해되어 왔다.35 이를 종합하면, 헬리코박터 파일로리와 나머지 위 미생물무리는 서로 상호작용하며 혹은 순차적으로 위암 발병에 기여할 가능성이 있다.
위 미생물무리를 구성하는 개별 미생물 중 헬리코박터 파일로리처럼 위암과의 강한 연관성을 보여준 미생물에 관한 연구 결과는 없었다. 하지만, 몇몇 연구들은 특정 미생물과 위암의 관계를 보고하였다.
유산균(lactic acid bacteria)은 위암 환자의 위 미생물무리에서 상대 풍부도가 증가해 있는 것으로 일관되게 보고되는 미생물군이다. 대부분의 연구들은 위암 환자의 위 미생물무리에서 Lactobacillus의 상대 풍부도가 증가한 것으로 보고하였다.17,18,20-23,27,28 다른 유산균 중에는 Streptococcus,18,22,23,25 Bifidobacterium,20,28 Lactococcus,27 및 Lacticaseibacillus29의 상대 풍부도가 증가한 것으로 보고한 연구도 있었다. 일부 유산균은 세포 실험에서 염증 물질의 발생을 줄이거나 위암 세포의 증식을 억제하는 것으로 보고된 바 있다.36,37 그러나 젖산은 암세포의 에너지원으로 사용될 수 있다.20 또한, 유산균의 과증식은 질산염(nitrate)이 아질산염(nitrate)으로 환원되는 과정을 촉진하여, 질소 화합물의 형성을 유도할 수 있다.38 Ferreira 등21의 연구에서는 위암의 위 미생물무리에서 Lactobacillus가 증가해 있었고, 생물정보학적으로 예측한 위 미생물무리의 기능에서 질산화(nitrosating) 기능이 증가해 있어 이러한 가설을 뒷받침하였다. 또한, 유산균은 잠재적으로 활성산소(reactive oxygen species)의 생성을 유도할 수 있으며, 이는 위암의 발생에 관계되어 있다.39 따라서, 유산균의 과도한 증식은 헬리코박터 파일로리와 별도로 위암 발생에 기여할 가능성이 있다. 다만 이러한 결과가 프로바이오틱스로서 일부 유산균을 섭취하는 것이 위암 발생에 기여할 수 있다는 의미는 아니며, 이에 관해서는 별도의 연구가 필요하다.
구강 상재균도 유산균만큼이나 위암 환자의 위 미생물무리에서 증가한 것으로 보고되고 있는 균들이다. Coker 등22의 연구에서는 위암에서 증가하는 위 미생물 21개 중 14개가 구강 세균으로 보고하였다. Zhang 등26의 연구에서는 4개의 구강 미생물이 위암의 미생물무리에서 증가하였다고 보고하였고, Gantuya 등24은 위암에서 구강 상재균 중 Fusobacterium의 증가를, Park 등27의 연구와 Castaño-Rodríguez 등20의 연구에서는 Veillonella의 증가를 보고하였다. 구강 상재균은 위암의 발생 과정 중 위축성 위염이나 장상피화생 단계에서 위산 분비가 저하되면서 위 점막에서 상대 풍부도가 증가한다.16 따라서 위암 환자의 위 미생물무리에서 이 균들의 상대 풍부도가 증가하는 것은 위암 자체보다는 위축성 위염이나 장상피화생으로 인한 것일 수도 있다. 그러나, 구강 상재균은 식도암, 대장암, 췌장암, 및 염증성 장질환 등 여러 질환과 연관되어 있다.40-42 식도암 중에서는 편평세포암과 구강 상재균의 연관성이 잘 알려져 있는데, 식도편평세포암에서 구강 미생물무리의 다양성이 감소해 있고, Prevotella와 Streptococcus 등의 상대 풍부도가 증가한 것으로 보고되었다.42 염증을 유발하는 특성으로는 Fusobacterium이 잘 알려져 있다.43 따라서 구강 상재균은 위산 분비가 감소하는 위축성 위염이나 장상피화생 단계부터 위 점막에서 만성 염증을 유발하여 위암 발생에 기여할 가능성이 있다.
이외에도 Nitrospirae에 속하는 많은 균들은 아질산염을 산화하여 질소 화합물의 생성을 줄이는데, Wang 등23은 이러한 Nitrospirae가 위암에서 감소해 있다고 보고한 바 있다. 아울러 이 연구에서는 위암에서 질산염 및 아질산염 환원 효소 기능이 위암에서 감소하였다고 보고하여, Nitrospirae의 감소가 위내 질소화합물의 증가에 따른 위암 발병에 기여할 가능성을 제시하였다. 이는 위에서 언급한 Ferreira 등21의 연구에서도 위암의 전체 미생물무리에서 질산화 기능이 증가해 있다고 보고한 것과 일치하는 결과이다. 한편, 피부 상재균으로 잘 알려져 있는 Propionibacterium acnes가 위암 환자의 위 미생물무리에서 증가한 것으로 보고한 연구들도 있다.44 이 균은 short chain fatty acid (SCFA)를 생산하며 위염 발생에 기여하는 것으로 알려져 있다.45
지금까지 언급된 연구들은 위암과 연관된 위 미생물무리에관한 다량의 새로운 정보를 제공하였다. 그러나 이들 연구는 뚜렷한 한계를 가지고 있다. 첫째, 대부분의 연구가 환자 대조군 설계로 위암 환자와 대조군 사이의 위 미생물무리의 차이를 보여주었지, 엄밀하게 위암 발생 과정에서 위 미생물무리가 변화하는 것을 보여주었다고 말하기 어렵다. 위암 발생 과정을 전향적으로 추적하는 연구가 위암과 연관된 위 미생물의변화를 보여줄 수 있다.46 둘째, 위암 환자의 위 미생물무리에서 상대 풍부도가 증가하는 미생물들이 연구마다 일부 일관되게 보고되기도 하지만, 여전히 많은 미생물들이 연구마다 서로 다르게 보고된다. 이것은 위에서도 언급한 바와 같이 인종이나 식이를 포함한 여러 인자의 차이 때문일 수도 있지만,근본적으로 16S rRNA 유전자의 과변이 부위 일부에 대해서염기서열 분석을 시행하여 종(species)이 아닌 속(genus) 수준까지만 구분이 가능한 현재 기술의 한계라고 볼 수도 있다.이는 16S rRNA 유전자 전장 염기서열 분석을 통해 종 수준까지 구분이 가능한 해상도를 구현하여 해결할 수 있는 가능성이 있다.47 셋째, 16S rRNA 유전자 염기서열 분석만으로는위 미생물무리의 계통학적 구성을 보여줄 뿐 실제 기능을 보여주지는 못한다. 현재 연구들에서 제시하고 있는 위암 관련위 미생물무리의 기능은 PICRUSt 등 생물정보학적 도구를 이용하여 예측한 기능이며, 이는 샷건 메타지노믹스(shotgun metagenomics)로 분석한 유전자 기능의 약 80% 정도를 예측할 수 있는 것으로 알려져 있다.48 따라서, 위암 관련 위 미생물무리의 기능을 알기 위해서 현재로서 가장 이상적인 방법은 샷건 염기서열분석을 통해 미생물무리의 메타지놈 분석(metagenomic analysis)을 직접 시행하는 것이다. 그러나, 위 조직을 이용한 샷건 메타지노믹스는 위가 분변 등과 비교하여 세균 DNA의 양이 적은 편인 데다가 전체 DNA 중 인간 DNA의 비율이 너무 높기 때문에 아직 한계가 있다. 따라서, 위액을 이용한 접근법이 시도되고 있다.49 이외에도 진균 미생물무리에 대한 분석,50 메타볼로믹스(metabolomics)를 포함한 분석,51 RNA 시퀀싱을 포함한 멀티오믹스 분석52 등 현재 위 마이크로바이옴 연구의 한계를 넘기 위한 많은 시도들이 이루어지고 있다.
위암과 연관된 위 미생물무리의 연관 관계가 실제 인과관계를 살펴볼 수 있는 방법으로 전향적 관찰 연구를 통한 변화를 보는 것 이외에 또 다른 방법은 동물 모델을 통한 연구이다. Lofgren 등53의 연구가 insulin-gastric transgenic (INS-GAS) 마우스 모델을 사용한 대표적인 연구이다. INS-GAS 마우스는 혈중 가스트린 수치가 높아 위축성 위염을 일으키고, 헬리코박터 파일로리에 감염되면 위 상피내종양을 일으키는 모델이다. 이 연구에서는 무균(germ-free) INS-GAS 마우스보다 여러 미생물에 노출된 INS-GAS 마우스에서 헬리코박터 파일로리 감염 후 더 빨리 위 상피내종양이 발생했다.53 Lertpiriyapong 등54은 INS-GAS 마우스가 헬리코박터 파일로리와 함께 Lactobacillus murinus ASF361, Clostridium sp. ASF356, 및 Bacteroides sp. ASF519 등 제한된 미생물에 노출되는 것이 좀 더 복잡한 미생물무리에 노출되는 것과 유사한 정도의 위 상피내종양 발생률을 보였다고 보고하였다. 또한, 위 점막에서는 종양유전자(oncogenes) 및 염증 유발 유전자(proinflammatory genes)들의 강한 발현 증가가 관찰되었다. 반면, Whary 등55은 헬리코박터 파일로리와 함께 기생충인 Heligmosomoides polygyrus를 감염시키는 경우 위 점막의 위축과 상피내종양이 적게 발생한다고 보고하였다. 최근에는 Kwon 등56이 무균 마우스에 위암 환자의 위 미생물무리를 이식하여 위암의 전구 병변들이 발생함을 보였다. 이들 연구는 위암 환자에서 발견되는 위 미생물무리가 위암 발병에 대한 기여 없이 위암이 발생하는 환경에서 존재하는 균이라기보다는 위암 발생에 기여할 수 있다는 근거를 제공하는 결과라고 할 수 있다.
인간 미생물무리와 관련된 연구들은 최근 급격히 발전하고 있고, 미생물무리와 여러 질환과의 관계가 연구되었다. 위 미생물무리에 관한 연구도 위 점막의 미생물로서 헬리코박터 파일로리 이외에서 많은 미생물들이 미생물무리의 형태로 존재한다는 것을 보여주었고, 위암 환자에서 그 구성이 위암이 없는 환자들과 뚜렷하게 다르다는 것 또한 보여주었다. 위암 환자의 위 미생물무리는 위암이 없는 경우와 비교하여 뚜렷하게 다른 구성을 보여주었다. 다양성의 변화에 관해서는 엇갈린 결과가 있으나 다수의 연구가 위암과 관련된 위 미생물무리는 다양성이 감소하는 것으로 보고하고 있다. 위암과 연관된 특성 미생물 무리로는 대표적으로 유산균과 구강 미생물이 보고되고 있다. 이들은 위 점막에서 만성 염증을 유발하고, 질소화합물의 생성을 증가시키는 것이 지금까지 언급되는 주요 기전이다. 일련의 동물 실험들은 이러한 위 미생물무리의 변화가 위암 발생과정의 결과라기보다는 원인으로 기여할 가능성을 제시하고 있다. 그러나, 단순히 16S rRNA 유전자 분석만으로 위암 환자에서 위 미생물무리의 존재와 구성을 보여주는 연구는 서서히 그 차별성을 상실해가고 있다. 또한, 이러한 위 미생물무리 연구가 임상에 적용되기는 아직 더 많은 연구가 필요하다. 가장 가능성이 있는 분야는 위암 진단의 마커로서 역할이다. 구강 미생물무리가 위 미생물무리와 연관이 있고 위암과도 연관이 있어, 구강 미생물무리가 비침습적인 위암의 마커로 고려될 수 있다.42 대장암의 경우 분변 잠혈 검사에 DNA 메틸화와 분변 미생물무리 검사를 추가한 검진에 관한 탐구가 이루어지고 있다.57 위암에서도 헬리코박터 파일로리 감염 등에 의해서 위 점막에 발생하는 후성유전체 변화 중 하나인 DNA 메틸화가 위암 발생 위험과 연관이 있다고 알려져 있다.13 따라서, 위암에서 DNA 메틸화와 위 미생물무리를 결합한 바이오마커가 임상에 적용할 가능성이 있는 분야라고 할 수 있다. 나아가, 헬리코박터 파일로리 제균 치료와 같이 위 미생물무리의 변화를 유도하여 위암 발생 위험을 줄이는 것이 위 마이크로바이옴 연구의 궁극적인 목표이자 위 미생물무리 연구가 나아갈 방향 중 하나라고 할 수 있다.
REFERENCES
1. Sung H, Ferlay J, Siegel RL, et al. Global cancer statistics 2020: GLOBOCAN Estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 2021; 71:209–249. DOI: 10.3322/caac.21660. PMID: 33538338.

2. 2022. Cancer Statitics. [Internet]. National Cancer Information Center;Available from: https://www.cancer.go.kr/lay1/S1T639C640/contents.do. cited 2023 May 11.
3. Dunn BE, Cohen H, Blaser MJ. 1997; Helicobacter pylori. Clin Microbiol Rev. 10:720–741. DOI: 10.1128/CMR.10.4.720. PMID: 9336670. PMCID: PMC172942.

4. Parsonnet J, Friedman GD, Vandersteen DP, et al. 1991; Helicobacter pylori infection and the risk of gastric carcinoma. N Engl J Med. 325:1127–1131. DOI: 10.1056/NEJM199110173251603. PMID: 1891020.

5. Nomura A, Stemmermann GN, Chyou PH, Kato I, Perez-Perez GI, Blaser MJ. 1991; Helicobacter pylori infection and gastric carcinoma among Japanese Americans in Hawaii. N Engl J Med. 325:1132–1136. DOI: 10.1056/NEJM199110173251604. PMID: 1891021.

6. Correa P. 1992; Human gastric carcinogenesis: a multistep and multifactorial process--First American Cancer Society Award Lecture on Cancer Epidemiology and Prevention. Cancer Res. 52:6735–6740.
7. Ding SZ, Goldberg JB, Hatakeyama M. 2010; Helicobacter pylori infection, oncogenic pathways and epigenetic mechanisms in gastric carcinogenesis. Future Oncol. 6:851–862. DOI: 10.2217/fon.10.37. PMID: 20465395. PMCID: PMC2882595.

8. Sung JJY, Coker OO, Chu E, et al. 2020; Gastric microbes associated with gastric inflammation, atrophy and intestinal metaplasia 1 year after Helicobacter pylori eradication. Gut. 69:1572–1580. DOI: 10.1136/gutjnl-2019-319826. PMID: 31974133. PMCID: PMC7456733.

9. Wie GA, Cho YA, Kang HH, et al. 2014; Red meat consumption is associated with an increased overall cancer risk: a prospective cohort study in Korea. Br J Nutr. 112:238–247. DOI: 10.1017/S0007114514000683. PMID: 24775061.

10. Tramacere I, Negri E, Pelucchi C, et al. 2012; A meta-analysis on alcohol drinking and gastric cancer risk. Ann Oncol. 23:28–36. DOI: 10.1093/annonc/mdr135. PMID: 21536659.

11. Buckland G, Travier N, Huerta JM, et al. 2015; Healthy lifestyle index and risk of gastric adenocarcinoma in the EPIC cohort study. Int J Cancer. 137:598–606. DOI: 10.1002/ijc.29411. PMID: 25557932.

12. Murata-Kamiya N, Kurashima Y, Teishikata Y, et al. 2007; Helicobacter pylori CagA interacts with E-cadherin and deregulates the beta-catenin signal that promotes intestinal transdifferentiation in gastric epithelial cells. Oncogene. 26:4617–4626. DOI: 10.1038/sj.onc.1210251. PMID: 17237808.

13. Yang HJ, Kim SG, Lim JH, Choi JM, Kim WH, Jung HC. 2018; Helicobacter pylori-induced modulation of the promoter methylation of Wnt antagonist genes in gastric carcinogenesis. Gastric Cancer. 21:237–248. DOI: 10.1007/s10120-017-0741-6. PMID: 28643146.

14. Stewart OA, Wu F, Chen Y. 2020; The role of gastric microbiota in gastric cancer. Gut Microbes. 11:1220–1230. DOI: 10.1080/19490976.2020.1762520. PMID: 32449430. PMCID: PMC7524314.

15. Engstrand L, Graham DY. 2020; Microbiome and gastric cancer. Dig Dis Sci. 65:865–873. DOI: 10.1007/s10620-020-06101-z. PMID: 32040665. PMCID: PMC8697197.

16. Rajilic-Stojanovic M, Figueiredo C, Smet A, et al. 2020; Systematic review: gastric microbiota in health and disease. Aliment Pharmacol Ther. 51:582–602. DOI: 10.1111/apt.15650. PMID: 32056247.

17. Aviles-Jimenez F, Vazquez-Jimenez F, Medrano-Guzman R, Mantilla A, Torres J. 2014; Stomach microbiota composition varies between patients with non-atrophic gastritis and patients with intestinal type of gastric cancer. Sci Rep. 4:4202. DOI: 10.1038/srep04202. PMID: 24569566. PMCID: PMC3935187.

18. Eun CS, Kim BK, Han DS, et al. 2014; Differences in gastric mucosal microbiota profiling in patients with chronic gastritis, intestinal metaplasia, and gastric cancer using pyrosequencing methods. Helicobacter. 19:407–416. DOI: 10.1111/hel.12145. PMID: 25052961.

19. Li TH, Qin Y, Sham PC, Lau KS, Chu KM, Leung WK. 2017; Alterations in gastric microbiota after H. pylori eradication and in different histological stages of gastric carcinogenesis. Sci Rep. 7:44935. DOI: 10.1038/srep44935. PMID: 28322295. PMCID: PMC5359573.

20. Castaño-Rodríguez N, Goh KL, Fock KM, Mitchell HM, Kaakoush NO. 2017; Dysbiosis of the microbiome in gastric carcinogenesis. Sci Rep. 7:15957. DOI: 10.1038/s41598-017-16289-2. PMID: 29162924. PMCID: PMC5698432.

21. Ferreira RM, Pereira-Marques J, Pinto-Ribeiro I, et al. 2018; Gastric microbial community profiling reveals a dysbiotic cancer-associated microbiota. Gut. 67:226–236. DOI: 10.1136/gutjnl-2017-314205. PMID: 29102920. PMCID: PMC5868293.

22. Coker OO, Dai Z, Nie Y, et al. 2018; Mucosal microbiome dysbiosis in gastric carcinogenesis. Gut. 67:1024–1032. DOI: 10.1136/gutjnl-2017-314281. PMID: 28765474. PMCID: PMC5969346.

23. Wang Z, Gao X, Zeng R, et al. 2020; Changes of the gastric mucosal microbiome associated with histological stages of gastric carcinogenesis. Front Microbiol. 11:997. DOI: 10.3389/fmicb.2020.00997. PMID: 32547510. PMCID: PMC7272699.

24. Gantuya B, El Serag HB, Matsumoto T, et al. 2020; Gastric mucosal microbiota in a Mongolian population with gastric cancer and precursor conditions. Aliment Pharmacol Ther. 51:770–780. DOI: 10.1111/apt.15675. PMID: 32133670. PMCID: PMC8761497.

25. Pimentel-Nunes P, Barros A, Pita I, et al. 2021; Gastric microbiome profile throughout gastric carcinogenesis: beyond helicobacter. Scand J Gastroenterol. 56:708–716. DOI: 10.1080/00365521.2021.1902560. PMID: 33915074.

26. Zhang X, Li C, Cao W, Zhang Z. 2021; Alterations of gastric microbiota in gastric cancer and precancerous stages. Front Cell Infect Microbiol. 11:559148. DOI: 10.3389/fcimb.2021.559148. PMID: 33747975. PMCID: PMC7966516.

27. Park JY, Seo H, Kang CS, et al. 2022; Dysbiotic change in gastric microbiome and its functional implication in gastric carcinogenesis. Sci Rep. 12:4285. DOI: 10.1038/s41598-022-08288-9. PMID: 35277583. PMCID: PMC8917121.

28. Sun QH, Zhang J, Shi YY, Zhang J, Fu WW, Ding SG. 2022; Microbiome changes in the gastric mucosa and gastric juice in different histological stages of Helicobacter pylori-negative gastric cancers. World J Gastroenterol. 28:365–380. DOI: 10.3748/wjg.v28.i3.365. PMID: 35110955. PMCID: PMC8771614.

29. Kim HN, Kim MJ, Jacobs JP, Yang HJ. 2022; Altered gastric microbiota and inflammatory cytokine responses in patients with Helicobacter pylori-negative gastric cancer. Nutrients. 14:4981. DOI: 10.3390/nu14234981. PMID: 36501012. PMCID: PMC9740132.

30. Hassler HB, Probert B, Moore C, et al. 2022; Phylogenies of the 16S rRNA gene and its hypervariable regions lack concordance with core genome phylogenies. Microbiome. 10:104. DOI: 10.1186/s40168-022-01295-y. PMID: 35799218. PMCID: PMC9264627.

31. Park CH, Lee AR, Lee YR, Eun CS, Lee SK, Han DS. 2019; Evaluation of gastric microbiome and metagenomic function in patients with intestinal metaplasia using 16S rRNA gene sequencing. Helicobacter. 24:e12547. DOI: 10.1111/hel.12547. PMID: 30440093. PMCID: PMC6587566.

32. Kim J, Kim N, Jo HJ, et al. 2015; An appropriate cutoff value for determining the colonization of Helicobacter pylori by the pyrosequencing method: Comparison with conventional methods. Helicobacter. 20:370–380. DOI: 10.1111/hel.12214. PMID: 25664664.

33. Ding J, Man YG, Deng X, Chen T. 2022; Differences in community structure of gastrointestinal tract between Helicobacter pylori positive patients and negative patients with gastric cancer. J Cancer. 13:1905–1913. DOI: 10.7150/jca.69873. PMID: 35399728. PMCID: PMC8990434.

34. Noto JM, Peek RM Jr. 2017; The gastric microbiome, its interaction with Helicobacter pylori, and its potential role in the progression to stomach cancer. PLoS Pathog. 13:e1006573. DOI: 10.1371/journal.ppat.1006573. PMID: 28982167. PMCID: PMC5629027.

35. Tsujimoto H, Ono S, Ichikura T, Matsumoto Y, Yamamoto J, Hase K. 2010; Roles of inflammatory cytokines in the progression of gastric cancer: friends or foes? Gastric Cancer. 13:212–221. DOI: 10.1007/s10120-010-0568-x. PMID: 21128056.

36. Vinasco K, Mitchell HM, Kaakoush NO, Castaño-Rodríguez N. 2019; Microbial carcinogenesis: Lactic acid bacteria in gastric cancer. Biochim Biophys Acta Rev Cancer. 1872:188309. DOI: 10.1016/j.bbcan.2019.07.004. PMID: 31394110.

37. Hwang JW, Baek YM, Yang KE, et al. 2013; Lactobacillus casei extract induces apoptosis in gastric cancer by inhibiting NF-κB and mTOR-mediated signaling. Integr Cancer Ther. 12:165–173. DOI: 10.1177/1534735412442380. PMID: 22505595.

38. Li ZP, Liu JX, Lu LL, et al. 2021; Overgrowth of Lactobacillus in gastric cancer. World J Gastrointest Oncol. 13:1099–1108. DOI: 10.4251/wjgo.v13.i9.1099. PMID: 34616515. PMCID: PMC8465450.
39. Jones RM, Mercante JW, Neish AS. 2012; Reactive oxygen production induced by the gut microbiota: pharmacotherapeutic implications. Curr Med Chem. 19:1519–1529. DOI: 10.2174/092986712799828283. PMID: 22360484. PMCID: PMC4269156.

40. Nakatsu G, Li X, Zhou H, et al. 2015; Gut mucosal microbiome across stages of colorectal carcinogenesis. Nat Commun. 6:8727. DOI: 10.1038/ncomms9727. PMID: 26515465. PMCID: PMC4640069.

41. Read E, Curtis MA, Neves JF. 2021; The role of oral bacteria in inflammatory bowel disease. Nat Rev Gastroenterol Hepatol. 18:731–742. DOI: 10.1038/s41575-021-00488-4. PMID: 34400822.

42. Reitano E, déAngelis N, Gavriilidis P, et al. 2021; Oral bacterial microbiota in digestive cancer patients: A systematic review. Microorganisms. 9:2585. DOI: 10.3390/microorganisms9122585. PMID: 34946186. PMCID: PMC8707512.

43. Tang B, Wang K, Jia YP, et al. 2016; Fusobacterium nucleatum-induced impairment of autophagic flux enhances the expression of proinflammatory cytokines via ROS in Caco-2 cells. PLoS One. 11:e0165701. DOI: 10.1371/journal.pone.0165701. PMID: 27828984. PMCID: PMC5102440.

44. Liu X, Shao L, Liu X, et al. 2019; Alterations of gastric mucosal microbiota across different stomach microhabitats in a cohort of 276 patients with gastric cancer. EBioMedicine. 40:336–348. DOI: 10.1016/j.ebiom.2018.12.034. PMID: 30584008. PMCID: PMC6412016.

45. Montalban-Arques A, Wurm P, Trajanoski S, et al. 2016; Propionibacterium acnes overabundance and natural killer group 2 member D system activation in corpus-dominant lymphocytic gastritis. J Pathol. 240:425–436. DOI: 10.1002/path.4782. PMID: 27538697. PMCID: PMC5111592.
46. Png CW, Lee WJJ, Chua SJ, et al. 2022; Mucosal microbiome associates with progression to gastric cancer. Theranostics. 12:48–58. DOI: 10.7150/thno.65302. PMID: 34987633. PMCID: PMC8690935.

47. Johnson JS, Spakowicz DJ, Hong BY, et al. 2019; Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis. Nat Commun. 10:5029. DOI: 10.1038/s41467-019-13036-1. PMID: 31695033. PMCID: PMC6834636.

48. Langille MG, Zaneveld J, Caporaso JG, et al. 2013; Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nat Biotechnol. 31:814–821. DOI: 10.1038/nbt.2676. PMID: 23975157. PMCID: PMC3819121.

49. Hu YL, Pang W, Huang Y, Zhang Y, Zhang CJ. 2018; The gastric microbiome is perturbed in advanced gastric adenocarcinoma identified through shotgun metagenomics. Front Cell Infect Microbiol. 8:433. DOI: 10.3389/fcimb.2018.00433. PMID: 30619779. PMCID: PMC6299874.

50. Yang P, Zhang X, Xu R, et al. 2022; Fungal microbiota dysbiosis and ecological alterations in gastric cancer. Front Microbiol. 13:889694. DOI: 10.3389/fmicb.2022.889694. PMID: 35572666. PMCID: PMC9100745.

51. Dai D, Yang Y, Yu J, et al. 2021; Interactions between gastric microbiota and metabolites in gastric cancer. Cell Death Dis. 12:1104. DOI: 10.1038/s41419-021-04396-y. PMID: 34819503. PMCID: PMC8613192.

52. Park CH, Hong C, Lee AR, Sung J, Hwang TH. 2022; Multi-omics reveals microbiome, host gene expression, and immune landscape in gastric carcinogenesis. iScience. 25:103956. DOI: 10.1016/j.isci.2022.103956. PMID: 35265820. PMCID: PMC8898972.

53. Lofgren JL, Whary MT, Ge Z, et al. 2011; Lack of commensal flora in Helicobacter pylori-infected INS-GAS mice reduces gastritis and delays intraepithelial neoplasia. Gastroenterology. 140:210–220. DOI: 10.1053/j.gastro.2010.09.048. PMID: 20950613. PMCID: PMC3006487.

54. Lertpiriyapong K, Whary MT, Muthupalani S, et al. 2014; Gastric colonisation with a restricted commensal microbiota replicates the promotion of neoplastic lesions by diverse intestinal microbiota in the Helicobacter pylori INS-GAS mouse model of gastric carcinogenesis. Gut. 63:54–63. DOI: 10.1136/gutjnl-2013-305178. PMID: 23812323. PMCID: PMC4023484.

55. Whary MT, Muthupalani S, Ge Z, et al. 2014; Helminth co-infection in Helicobacter pylori infected INS-GAS mice attenuates gastric premalignant lesions of epithelial dysplasia and glandular atrophy and preserves colonization resistance of the stomach to lower bowel microbiota. Microbes Infect. 16:345–355. DOI: 10.1016/j.micinf.2014.01.005. PMID: 24513446. PMCID: PMC4030519.

56. Kwon SK, Park JC, Kim KH, et al. 2022; Human gastric microbiota transplantation recapitulates premalignant lesions in germ-free mice. Gut. 71:1266–1276. DOI: 10.1136/gutjnl-2021-324489. PMID: 34389621.

57. Mo S, Wang H, Han L, et al. 2021; Fecal multidimensional assay for non-invasive detection of colorectal cancer: Fecal immunochemical test, stool DNA mutation, methylation, and intestinal bacteria analysis. Front Oncol. 11:643136. DOI: 10.3389/fonc.2021.643136. PMID: 33718241. PMCID: PMC7947614.





 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download