초록
순환종양핵산을 이용한 액체 생검은 기존 조직검사의 한계를 극복할 수 있다. 순환종양핵산을 이용한 비침습적 분자 프로파일링은 암의 진단 및 위험도 계층화, 표적 치료제 선택 등의 목적으로 임상적 활용이 증가하고 있다. 순환종양핵산의 생물학적 특성과 순환종양핵산 농도에 영향을 미칠 수 있는 환자 요인을 이해하고 검사법을 최적화 함으로써 순환종양핵산 검사의 임상적 활용도를 높일 수 있을 것이다. 향후 순환종양핵산 검사의 수행 및 결과 보고에 대한 가이드라인의 마련에 대해서도 고려가 필요하다.
Abstract
Liquid biopsy using circulating tumor DNA helps overcome the limitations of conventional tissue testing. Non-invasive molecular profiling using circulating tumor DNA is increasingly being used to diagnose cancer, stratify risk, and select targeted treatments. By understanding the biological characteristics of circulating tumor DNA and patient factors that can affect concentrations of circulating tumor DNA as well as optimizing the techniques, the clinical utilization of circulating tumor DNA tests can be improved. It is also necessary to formulate guidelines for the administration and reporting of circulating tumor DNA tests.
1948년에 혈액 내 세포유리핵산(cell-free DNA, cfDNA)의 존재가 보고되었고[1], 1977년에는 암환자의 혈액에 건강한 사람에 비하여 높은 농도의 cfDNA가 존재한다고 보고되었다[2]. 그 이후로 분자 검사기법의 발전과 함께 혈액을 이용한 순환종양핵산(circulating tumor DNA, ctDNA) 검사, 즉 액체 생검(liquid biopsy)에 대한 연구가 활발히 진행되었으며, 최근에는 임상 검사 영역에서 사용도 증가하고 있다. 종양의 분자프로파일링은 전통적으로 조직을 사용하여 시행하였으나, 혈액의 경우 조직에 비하여 덜 침습적으로 검체 채취가 가능하여 조직검사나 수술이 불가능하거나 조직이 소량인 환자에서 유용하다. 또한 ctDNA는 전이 병소를 포함한 모든 종양 조직에서 혈액으로 유리되므로 종양 내 이질성(intra-tumor heterogeneity)을 반영한 분석이 가능하며, 반복 검사를 통하여 치료 중 발생하는 클론성 변이의 역동적인 변화도 추적이 가능하다는 장점이 있다[3-5]. 이러한 특성으로 ctDNA 검사는 암의 진단 및 치료제 선택, 치료 반응 모니터링, 예후 예측 등에 활용될 수 있다. 본 종설의 목적은 최근 진료 영역으로 빠르게 도입되고 있는 ctDNA 검사에 대한 이해를 돕기 위하여 ctDNA의 특징과 검사 방법, 임상적 이용에 대하여 정리하고자 하였다.
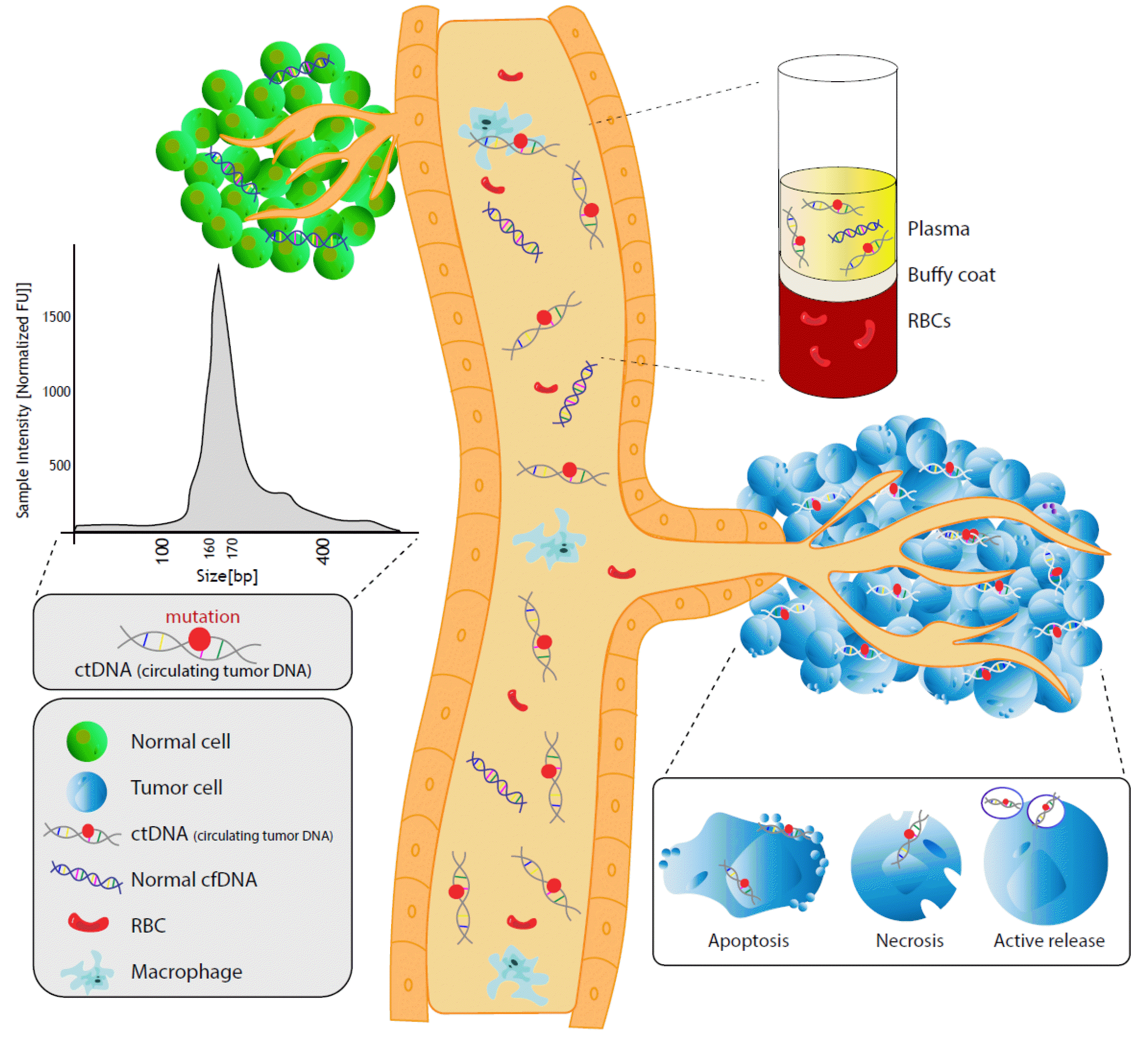
cfDNA는 세포 밖을 떠다니는 분절화된 DNA조각으로 주로 크기가160-170 bp이다[6-10]. 이들은 우리 몸의 혈액, 소변, 뇌척수액, 흉막액, 타액 등 체액에 존재하고 있으며, DNA가 유래된 세포의 유전 정보를 포함하고 있다[8-10]. 그리고 주로 세포자멸사(apoptosis), 괴사(necrosis) 및 활성분비(active release)에 의해 생성된다고 알려져 있다(Fig. 1) [11, 12]. 유핵세포의 유전체는 히스톤(histone) 단백질을 DNA가 감싸고 있는 뉴클레오좀(nucleosome)이 반복된 형태로 되어 있다[13]. 뉴클레오좀을 구성하는 DNA와 뉴클레오좀을 연결하는 linker DNA의 길이의 합은 세포자멸사로 인해 방출되는 cfDNA의 길이인 160-170 bp에 상응하며, 이것은 세포자멸사로 인해 세포 내의 DNA가 세포 밖으로 방출될 때 뉴클레오좀 단위로 끊어짐을 암시한다[6, 10]. 반면 괴사나 활성분비는 대개 이보다 더 큰 cfDNA 조각을 생성한다고 알려져 있다[14]. cfDNA는 뉴클레아제 활성, 신장 배설, 비장 및 간 흡수 등을 통해 제거되며[6], 반감기는 15분-2.5시간으로 매우 짧다[6, 9, 10]. 건강한 사람의 혈액에서는 cfDNA가 혈장 1 mL당 일반적으로 10 ng 이하로 발견되며 30 ng 이상이 되는 경우는 드물지만 운동, 감염, 외상, 암 등의 요인에 따라 양이 증가하기도 한다[6]. 암환자에서 종양세포는 세포자멸사와 괴사가 활발히 일어나 더 많은 DNA를 방출시키는데 이렇게 종양세포에서 유래한 cfDNA를 ctDNA라 부른다. ctDNA는 정상 세포에서 유래한 cfDNA 보다 짧다고 알려져 있으며[15, 16], 체세포 돌연변이의 검출을 통해 확인할 수 있다. ctDNA는 반감기가 매우 짧기 때문에 현재의 종양 부담(tumor burden)을 실시간으로 반영할 수 있다는 장점이 있다.
암 환자의 cfDNA에는 종양세포에서 유래한 ctDNA 뿐만 아니라 정상세포에서 유래한 DNA도 있기 때문에 정상 cfDNA에 비하여 ctDNA가 상대적으로 적은 경우 ctDNA의 검출률이 낮아지게 된다. ctDNA의 양이 많아지는 상황에는 여러 요인이 있다. 일반적으로 ctDNA의 양은 종양의 크기와 상관관계가 있다[17, 18]. 이러한 사실을 통해 ctDNA의 양을 확인하여 종양 부담을 예측하거나 반복적으로 측정하면서 치료 반응에 대한 정보를 얻을 수 있다. 또한 검출 가능한 수준의 ctDNA를 가진 환자 비율은 암 환자의 병기와도 상관 관계가 있다고 보고되었는데 초기 단계보다 진행된 병기의 암에서 ctDNA의 양이 더 높았다[19]. 암의 종류에 따라 발견되는 ctDNA의 검출률에도 차이가 있을 수 있다. 최근 10,000명 이상을 대상으로 한 광범위 암(pan-cancer) ctDNA 검사로부터 소세포성 폐암, 전립선암, 자궁암, 간암에서 높은 검출률 및 갑상선암, 신장암에서 낮은 검출률이 보고된 바 있다[20]. 이 밖에도 종양의 증식 및 회전률, 위치, 혈관 분포 등과도 관련된다고 보고되었지만 더 많은 연구가 필요하다[21].
ctDNA 검사에 가장 많이 사용되는 검체는 혈액이다. 혈액의 성분 중에서는 혈장(plasma)이 혈청(serum)보다 ctDNA 검사에 적합하다. 혈액 응고 과정에서 백혈구가 손상되면서 세포유래 DNA (cellular DNA)가 유리되므로, 혈장보다 혈청에서 genomic DNA의 농도가 높게 측정되고, 이는 검사의 타겟인 cfDNA 검사의 민감도를 저해하는 요인이 된다[22]. 이외에도 ctDNA 검사의 검사전 단계(pre-analytical stage)에 고려할 부분들에 대하여 이전 문헌들을 참고할 수 있으며[23-25], 본 문헌에서는 이를 자세히 다루지 않았다.
혈액 이외에도 암이 발생한 장기에 따라서 인접한 부위에 존재하는 체액(소변, 뇌척수액, 흉수, 복수)을 이용하여 ctDNA 검사를 시행할 수 있다. 소변의 cfDNA (uninary cfDNA)는 요로(urinary tract)의 세포가 세포자멸사 혹은 괴사되면서 유리된 DNA와, 혈액에 존재하다가 사구체 여과(glomerular filtration) 과정을 거쳐 소변으로 나온 DNA (transrenal DNA)로 구성되어 있다[26]. Transrenal DNA는 사구체 여과 장벽 때문에 10-150 bp 정도로 분자량이 매우 작은(ultrashort) DNA로 구성되어 있다는 보고가 있으며[27, 28], 혈액의 cfDNA에 비하여 반감기가 매우 짧았다는 보고도 있어[29] 분석에 고려가 필요하다. 뇌종양의 경우 혈뇌장벽(blood-brain barrier) 때문에 혈액에서 ctDNA의 검출률이 낮을 수 있고[19], 뇌척수액을 이용하여 혈액보다 나은 결과를 얻을 수 있다는 보고들이 있다[30, 31]. 폐암 환자에서는 흉수를 사용하여 종양유래변이의 확인이 가능하다[32, 33]. 체액을 사용한 ctDNA 검사의 경우 혈중 ctDNA 농도가 낮아 분석이 어려운 환자에서 대안이 될 수 있을 것으로 기대되고 있으나 혈액에 비하여 근거 데이터가 부족하며 검체 처리에 대한 표준 프로토콜의 정립이 필요하다.
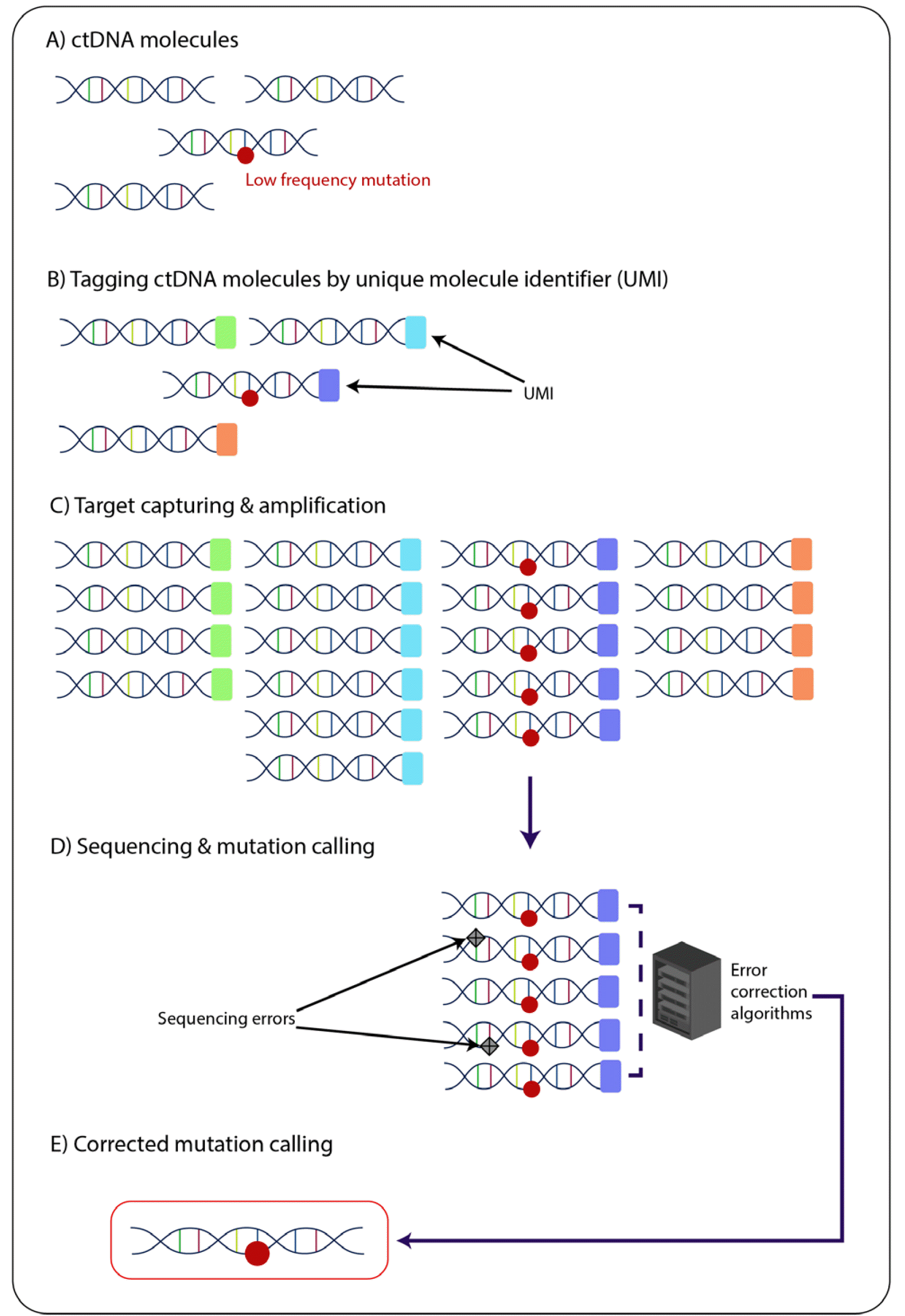
ctDNA는 정상세포에서 유래한 cfDNA의 양에 비해 상대적으로 낮은 경우가 많기 때문에 민감도가 높은 검사법을 사용해야 한다. ctDNA 분석에 사용되는 검사법은 하나 혹은 몇 개의 변이를 타겟으로 하는 접근법과, 여러 변이 혹은 유전자 하나 이상의 단위로 넓은 부위를 타겟으로 하는 접근법으로 나눌 수 있다. 전자는 실시간중합효소연쇄반응법(real-time quantitative polymerase chain reaction, RQ-PCR)과 디지털중합효소연쇄반응법(digital PCR)이 대표적이며, 후자는 차세대염기서열분석(next-generation sequencing, NGS)이 대표적이다(Table 1). RQ-PCR의 경우 알려진 호발부위(hotspot) 돌연변이를 타겟으로 하며 다중화(multiplexing)를 통하여 대상 변이의 수를 늘릴 수 있다. Digital PCR의 경우 주로 드롭렛(droplet)을 사용하거나 미세유동 칩(microfluidic chip) 등을 사용하여 타겟 DNA를 많은 공간에 분리시키고 PCR 반응 후 증폭 여부를 계수하는 방식을 사용한다. 이러한 digital PCR은 PCR 저해제(inhibitor)에 의한 영향 및 PCR 효율에 의한 오차가 적어 높은 민감도와 특이도를 얻을 수 있으며 표준곡선(standard curve)이 없이도 절대 정량이 가능하여 ctDNA 분석에 많이 이용된다[34, 35]. NGS의 경우 타겟의 범위에 따라 타겟 시퀀싱(targeted sequencing)과 엑솜 시퀀싱(whole exome sequencing) 혹은 전장유전체 시퀀싱(whole genome sequencing)으로 나눌 수 있으며, 기존에 알려지지 않은 단일염기변이(single nucleotide variant, SNV), 유전자량변이(copy number variation), 구조변이(structural variation)의 검출이 가능하다. 타겟 시퀀싱에서 원하는 부위를 타겟 선별(target enrichment)하는 방법은 앰플리콘(amplicon) 방법과 프로브(probe)를 이용하여 교잡(hybridization)하는 캡쳐(capture) 방법으로 나눈다. 앰플리콘 방식은 캡쳐 방식보다 간단하고 빠르며 상대적으로 적은 양의 DNA로 검사가 가능하다. 캡쳐 방식은 패널의 크기가 큰 경우에 더 선호된다. ctDNA 분석에 적합한 높은 민감도를 얻기 위해서는 NGS의 시퀸싱 리드(read) 수를 늘리고(deep sequencing), 시퀀싱 과정에서 생기는 에러를 제거하기 위한 추가적인 방법을 사용해야 한다. 에러를 제거하기 위한 방법으로 대표적인 것이 분자바코드(molecular barcode), 즉 고유분자식별자(unique molecular identifier, UMI)를 사용하는 것이다(Fig. 2) [12]. UMI는 무작위의 시퀀스로서, 주로 4-14개의 뉴클레오티드로 구성되어 있으며 얻어진 시퀀싱 리드를 원래의(original) DNA 주형(template)으로 추적할 수 있게 한다. 만약 같은 UMI가 연결된 리드의 일부에서만 변이가 나타난다면, 이는 시퀀싱 과정에서 생긴 에러로 간주할 수 있어 DNA 중합효소나 고르지 않은 증폭에 의하여 생긴 에러를 교정할 수 있도록 한다[36].
폐암의 조직형은 크게 비소세포폐암(non-small cell lung cancer, NSCLC)과 소세포암(small cell lung cancer)으로 나뉜다. 비소세포폐암에 속하는 선암(adenocarcinoma)은 EGFR, ALK, MET, BRAF, ROS1, NTRK, RET 등의 종양유발 드라이버 돌연변이(oncogenic driver mutation)에 의해서 발생하는데, 대상 돌연변이에 따라 서로 다른 표적치료제가 사용될 수 있어 한번에 여러 유전자의 검사가 가능한 NGS 기반의 패널검사가 선호된다[37]. 비소세포폐암의 경우 유전자 검사를 위한 조직을 충분히 얻을 수 있는 경우가 약 18% 정도로 매우 적으며, 재조직검사를 시행하는데 시간 소요가 크다. 반면에 ctDNA 검사의 경우 검체를 얻기 쉽고, 검사소요시간이 짧으며, 추적검사가 가능하다는 장점이 있어 비소세포폐암 환자의 진단, 항암치료 반응평가, 약물내성 확인 및 향후 치료방침 결정에 매우 유용하다[38]. ctDNA 검사가 가장 먼저 임상검사로 도입된 것은 비소세포폐암 환자에서 EGFR 돌연변이를 확인하여 EGFR 티로신키나아제 억제제(tyrosine kinase inhibitor, TKI) 적응증을 확인하기 위한 동반 진단(companion diagnostics) 목적이었다. 국제폐암연구협회(International Association for the Study of Lung Cancer, IASLC)에서 2021년에 진행성 비소세포폐암에서 액체 생검에 대한 개정된 합의문을 발표하였다[39]. 합의문에서는 이전의 표적치료제 사용 이후 질환의 진행을 보일 때, 즉 획득 내성을 보이는 상황에서 ctDNA검사부터(plasma-first) 시행하는 방법이 조직 채취의 어려움을 극복하고 치료방향 결정을 위한 유용한 정보를 얻을 수 있어 적합하다고 권고하였다. 또한 암유전자중독(oncogene-addicted) 비소세포폐암의 경우 치료방향 결정을 위한 유용한 정보를 얻을 수 있어 진단 시에 조직을 사용한 유전자검사와 동시에 시행하여 치료 타겟이 되는 돌연변이의 검출률을 높이는 전략에 대해서도 언급하고 있다.
전립선암은 전립선 특이 항원(prostate specific antigen, PSA)의 도입으로 조기 발견이 가능하게 되었고, 국소화되어 있을 경우 조직검사는 비교적 어렵지 않다. 전립선 암세포가 남성호르몬에 의존해서 성장하는 성질이 있기 때문에 전립선암의 성장을 억제하는 호르몬 치료는 전이성 전립선암에서 효과를 거둘 수 있으나 결국 일정기간 이후에는 호르몬 불응성인 전이성 거세 저항성 전립선암(metastatic castration-resistant prostate cancer, mCRPC)으로 진행하게 된다[40]. 이런 경우 PARP (poly ADP-ribose polymerase) 억제제 치료의 대상자 선정은 ctDNA 분석의 가장 강력한 장점이 될 수 있다. 2020년 5월 PARP 억제제인 olaparib은 상동 재조합 복구(homologous recombination repair, HRR) 유전자의 생식세포 또는 체세포 돌연변이를 가진 mCRPC에서 미국 식품의약청(Food and Drug Administration, FDA) 승인을 받았다[41]. 새로 진단된 전립선암 환자의 약 10%에서 뼈 전이가 나타나며, 진행된 단계에서는 80%로 증가하지만[42, 43], 전이된 뼈에서 조직검사를 하는 것은 어렵다. ctDNA 검사는 비침습적으로 HRR 유전자 돌연변이를 확인하여 PARP 억제제 대상자를 선정하는데 도움을 줄 수 있다. 또한 전이성 전립선암 환자에서 호르몬 치료를 시작하거나 또는 호르몬 치료를 하는 도중에 ctDNA 검사를 통한 AR 유전자의 돌연변이를 확인하는 것은 호르몬 치료 결정에 도움을 줄 수 있다[44]. 이 밖에 국소 전립선암에서 일반적으로 ctDNA는 잘 검출되지 않는다고 알려져 있지만, 수술 전에 ctDNA가 검출될 경우 나쁜 예후와 연관되어 있다[45]. 또한 mCRPC에서 ctDNA의 발견과 정량은 PSA와 별개로 유용한 정보를 제공해줄 수 있다고 보고되었다[46].
대장암 발생률은 최근에 선진국을 중심으로 증가하여 서방국가들의 암 관련 사망률의 10%로 상승하였고, 우리나라에서도 2018년 암 사망률의 3번째를 차지한다[47]. 대장 샘종의 조기 발견과 제거는 대장암을 예방하는 가장 효과적인 방법 중 하나이며, 대장암에 대한 내시경 검진이 도입되면서 많은 사람들이 전임상 단계에서 진단되기도 한다[48]. 태아성암항원(carcinoembryonic antigen, CEA)과 탄수화물 항원 19-9 (carbohydrate antigen 19-9, CA19-9)는 대장암의 종양표지자로 사용되지만 민감도와 특이도는 만족할 만한 수준은 아니며[48], 종양 생검은 대장암 모니터링을 위해 반복적으로 사용하기는 어렵다.
ctDNA는 대장암의 조기 발견, 수술 후 모니터링, 치료 반응 및 약물 내성 확인[49]에 도움을 줄 수 있다. 현재 대장암의 조기 진단을 위한 일차적 방법으로 대변 잠혈 검사, 직장수지검사, 종양표지자 검사를 이용한다. ctDNA 검출은 기존 종양표지자 검사와 병합했을 때 진단에 이점을 줄 수 있었으며[50], ctDNA의 메틸화 정도가 대장암 진단에 도움을 줄 수 있는 마커로서의 가능성도 확인되었다[51]. 또한 대장암 치료 직후 ctDNA 양의 변화는 치료 반응성을 확인하게 해주고, 치료 이후 지속적인 ctDNA 양의 모니터링을 통해 대장암 재발을 조기에 확인할 수 있음이 보고되었다[52]. 또한 ctDNA를 통한 체세포 돌연변이 확인은 전이성 대장암에서 다양한 표적치료제 사용에 도움을 줄 수 있다. KRAS, NRAS, BRAF 돌연변이는 전이성 대장암의 cetuximab이나 panitumumab과 같은 EGFR 억제제에 저항성을 유발한다[53]. BRAF V600E 돌연변이가 있는 대장암에서는 BRAF 억제제가 이용될 수 있고[54], HER 2 증폭이 있는 대장암에서는 HER 2 억제제가 이용될 수 있다[55]. 이렇게 ctDNA 검사는 비침습적으로 특정 표적 치료제로 치료하거나 특정 표적 치료제를 사용하지 않아야 하는 대장암 환자의 선정에 도움을 준다.
최근 호르몬 수용체(hormone receptor, HR) 양성, 사람상피세포성장인자수용체2 (human epidermal growth factor receptor 2, HER 2) 음성인 진행성 유방암 환자를 대상으로 혈장과 조직을 사용한 PIK3CA 돌연변이 검사가 α-selective phosphatidylinositol 3-kinase (PI3K) inhibitor인 alpelisib 투여 적응증 확인을 위한 동반진단검사로 허가되었다[56]. ctDNA 검사는 선행화학요법을 시행하는 유방암 환자들에서 위험도 평가 및 치료 반응 평가에 사용될 수 있으며[57], 삼중음성 유방암(triple-negative breast cancer, TNBC) 환자에서 PARP 억제제 치료의 근거를 제공한다[58]. 이외에도 표적 치료제 대상이 되는 변이를 확인하거나, 약물 내성 모니터링[59, 60] 및 획득 내성의 기전을 확인하기 위한 목적으로 주목받고 있다[61].
췌장암은 치명적인 암으로 2017년 기준 우리나라 암 관련 사망 원인 5번째이고 5년 생존율은 약 12.2%이다. 췌장암은 10-20% 환자만이 절제가 가능한 상태에서 진단되며 췌장절제술을 받은 환자들도 70-80%에서 재발한다[57]. 췌장암의 바이오마커(biomarker)로 널리 사용되는 CA19-9의 민감도와 특이도는 증상이 있는 환자에게서 각각 약 80%, 86%인 것으로 알려져 있으나 낮은 양성예측도로 인해 스크리닝 목적으로 사용하기 어렵다. 또한 담관폐쇄질환을 포함한 양성 질환 또는 다른 악성 종양에 의해서 비특이적인 증가를 보일 수 있고 특정 혈액형을 가지는 환자에게서 위음성을 유발할 수 있다는 단점이 있다[62]. 췌장암의 새로운 바이오마커 발굴을 위해 혈액으로부터 얻어진 ctDNA를 분석하여 췌장암의 조기 발견, 진단, 예후 판정에 활용하려는 연구들이 진행되었다[63, 64]. ctDNA 분석을 통하여 90% 이상의 췌장암 환자에서 중요 드라이버 유전자로 알려진 KRAS, TP53, SMAD4, CDKN2A로부터 돌연변이가 검출되었고, DNA 손상반응결함(DNA damage response defective, DDR defective)과 연관된 유전자를 포함하여 다양한 표적 치료제의 대상이 되는 돌연변이가 관찰되었다. 치료 전후 KRAS를 포함한 ctDNA 검출 여부가 전체 생존기간(overall survival, OS) 및 무진행 질병 생존 기간(progression free survival, PFS) 감소와 연관된다는 다양한 연구 결과들이 보고되었다. 스크리닝 목적으로 시행되는 ctDNA 분석의 경우 민감도가 약 64%로 낮고 만성췌장염 환자에서 관찰될 수 있는 KRAS 돌연변이로 인한 위양성 가능성이 있어 추가적인 연구가 필요하다. 이를 보완하기 위해 CA19-9와 조합하였을 때 민감도가 97%까지 향상되었으며, 다표적다적중모델(multitarget model)을 적용하여 정확도를 개선하려는 노력이 진행되고 있다[63].
난소암의 발생률과 사망률은 우리나라에서 지속적으로 증가하는 추세에 있으며, 전 세계에서 2번째로 치명적인 부인암이다[47, 65]. 20대, 30대 여성에서 4번째로 호발하는 암종이지만 난소암의 조기 발견을 위한 실질적인 방법은 없으며, 환자의 70%가 초진 시에 진행기(advanced stage) 진단을 받는다[47, 66]. 난소암의 치료는 외과적 절제와 백금 기반 화학 요법(platinum-based chemotherapy)에 기초한다. 많은 난소암환자들은 첫 치료 후 완전관해를 보이지만, 50%에서는 궁극적으로 재발한다[67]. 현재 재발을 감시하는 방법으로 탄수화물 항원(carbohydrate antigen 125, CA 125)과 인간부고환단백(human epididymis protein 4, HE4)과 같은 종양표지자와 CT (computed tomography) 촬영을 이용하고 있지만, 종양표지자가 상승하지 않거나 비특이적으로 상승하는 경우가 있으며, CT촬영의 경우 재발이 상당히 진행된 후에 발견할 수 있다는 단점이 있다[68-70]. 난소암에서 ctDNA 검사는 종양에서 유래한 돌연변이를 비침습적으로 추적관찰할 수 있어 기존 방법의 단점을 보완하기 위한 방법으로 주목받고 있다(Fig. 3). 또한 2019년 미국 FDA로부터 승인 받은 난소암 치료제인 PARP 억제제의 투여 대상 확인 목적으로 BRCA1/BRCA2 유전자로 대표되는 HRR 유전자 변이를 검출하거나, 치료 내성과 관련된 변이를 확인하는 목적 및 BRCA1/BRCA2 유전자의 복귀(reversion) 변이를 확인하는 목적으로도 ctDNA 검사가 이용되고 있다[60, 66].
진행성 고형암에서 현미부수체 불안정성(microsatellite instability, MSI), 높은 종양변이 부담(tumor mutation burden-high, TMB-H)이 발견되는 것은 종양의 면역성 신생항원(immunogenic neoantigen)의 발현이 많다는 것을 의미하기 때문에 pembrolizumab과 같은 면역관문 억제제(immune checkpoint inhibitor)에 효능을 보일 수 있다[60, 71]. 따라서 암세포에서 발견되는 MSI와 TMB-H는 면역관문 억제제 치료를 위한 환자를 선택하고 모니터링하는데 중요한 바이오마커가 될 수 있다. 하지만 조직을 통한 MSI와 TMB-H의 확인은 종양의 절제가 불가능한 경우에는 어렵고, 전이로 인하여 종양의 이질성이 생기는 경우 전체 종양을 대표할 수 없다. 따라서 ctDNA 검사를 이용하여 MSI와 TMB-H를 확인하는 방법이 대안으로 주목 받고 있으며, 조직검사와의 일치도나 약물에 대한 반응을 예측하는 바이오마커로서의 성능에 대한 연구결과들이 보고되고 있다[72-74].
REFERENCES
1. Mandel P, Metais P. 1948; Nuclear acids in human blood plasma. C R Seances Soc Biol Fil. 142:241–3.
2. Leon SA, Shapiro B, Sklaroff DM, Yaros MJ. 1977; Free DNA in the serum of cancer patients and the effect of therapy. Cancer Res. 37:646–50.
3. Siravegna G, Mussolin B, Buscarino M, Corti G, Cassingena A, Crisafulli G, et al. 2015; Clonal evolution and resistance to EGFR blockade in the blood of colorectal cancer patients. Nat Med. 21:795–801. DOI: 10.1038/nm.3870. PMID: 26030179. PMCID: PMC4868598.

4. Ulz P, Belic J, Graf R, Auer M, Lafer I, Fischereder K, et al. 2016; Whole-genome plasma sequencing reveals focal amplifications as a driving force in metastatic prostate cancer. Nat Commun. 7:12008. DOI: 10.1038/ncomms12008. PMID: 27328849. PMCID: PMC4917969.

5. De Mattos-Arruda L, Weigelt B, Cortes J, Won HH, Ng CKY, Nuciforo P, et al. 2014; Capturing intra-tumor genetic heterogeneity by de novo mutation profiling of circulating cell-free tumor DNA: a proof-of-principle. Ann Oncol. 25:1729–35. DOI: 10.1093/annonc/mdu239. PMID: 25009010. PMCID: PMC6276937.

6. Andersson D, Kristiansson H, Kubista M, Ståhlberg A. 2021; Ultrasensitive circulating tumor DNA analysis enables precision medicine: experimental workflow considerations. Expert Rev Mol Diagn. 21:299–310. DOI: 10.1080/14737159.2021.1889371. PMID: 33683971.

7. Galarza Fortuna GM, Dvir K. 2020; Circulating tumor DNA: Where are we now? A mini review of the literature. World J Clin Oncol. 11:723–31. DOI: 10.5306/wjco.v11.i9.723. PMID: 33033694. PMCID: PMC7522544.

8. Heitzer E, Ulz P, Geigl JB. 2015; Circulating tumor DNA as a liquid biopsy for cancer. Clin Chem. 61:112–23. DOI: 10.1373/clinchem.2014.222679. PMID: 25388429.

9. Lin C, Liu X, Zheng B, Ke R, Tzeng CM. 2021; Liquid biopsy, ctDNA diagnosis through NGS. Life (Basel). 11:890. DOI: 10.3390/life11090890. PMID: 34575039. PMCID: PMC8468354.

10. Wan JCM, Massie C, Garcia-Corbacho J, Mouliere F, Brenton JD, Caldas C, et al. 2017; Liquid biopsies come of age: towards implementation of circulating tumour DNA. Nat Rev Cancer. 17:223–38. DOI: 10.1038/nrc.2017.7. PMID: 28233803.

11. Hu Z, Chen H, Long Y, Li P, Gu Y. 2021; The main sources of circulating cell-free DNA: Apoptosis, necrosis and active secretion. Crit Rev Oncol Hematol. 157:103166. DOI: 10.1016/j.critrevonc.2020.103166. PMID: 33254039.

12. Stroun M, Lyautey J, Lederrey C, Olson-Sand A, Anker P. 2001; About the possible origin and mechanism of circulating DNA apoptosis and active DNA release. Clin Chim Acta. 313:139–42. DOI: 10.1016/S0009-8981(01)00665-9.
13. Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ. 1997; Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature. 389:251–60. DOI: 10.1038/38444. PMID: 9305837.

14. Bronkhorst AJ, Ungerer V, Holdenrieder S. 2019; The emerging role of cell-free DNA as a molecular marker for cancer management. Biomol Detect Quantif. 17:100087. DOI: 10.1016/j.bdq.2019.100087. PMID: 30923679. PMCID: PMC6425120.

15. Mouliere F, Chandrananda D, Piskorz AM, Moore EK, Morris J, Ahlborn LB, et al. 2018; Enhanced detection of circulating tumor DNA by fragment size analysis. Sci Transl Med. 10:eaat4921. DOI: 10.1126/scitranslmed.aat4921. PMID: 30404863. PMCID: PMC6483061.

16. Mouliere F, Robert B, Arnau Peyrotte E, Del Rio M, Ychou M, Molina F, et al. 2011; High fragmentation characterizes tumour-derived circulating DNA. PLoS One. 6:e23418. DOI: 10.1371/journal.pone.0023418. PMID: 21909401. PMCID: PMC3167805.

17. Abbosh C, Birkbak NJ, Wilson GA, Jamal-Hanjani M, Constantin T, Salari R, et al. 2017; Phylogenetic ctDNA analysis depicts early-stage lung cancer evolution. Nature. 545:446–51. DOI: 10.1038/nature22364. PMID: 28445469. PMCID: PMC5812436.
18. Kamat AA, Bischoff FZ, Dang D, Baldwin MF, Han LY, Lin YG, et al. 2006; Circulating cell-free DNA: a novel biomarker for response to therapy in ovarian carcinoma. Cancer Biol Ther. 5:1369–74. DOI: 10.4161/cbt.5.10.3240. PMID: 16969071.

19. Bettegowda C, Sausen M, Leary RJ, Kinde I, Wang Y, Agrawal N, et al. 2014; Detection of circulating tumor DNA in early- and late-stage human malignancies. Sci Transl Med. 6:224ra24.
20. Zhang Y, Yao Y, Xu Y, Li L, Gong Y, Zhang K, et al. 2021; Pan-cancer circulating tumor DNA detection in over 10,000 Chinese patients. Nat Commun. 12:11. DOI: 10.1038/s41467-020-20162-8. PMID: 33397889. PMCID: PMC7782482.

21. Grabuschnig S, Soh J, Heidinger P, Bachler T, Hirschböck E, Rosales Rodriguez I, et al. 2020; Circulating cell-free DNA is predominantly composed of retrotransposable elements and non-telomeric satellite DNA. J Biotechnol. 313:48–56. DOI: 10.1016/j.jbiotec.2020.03.002. PMID: 32165241.

22. Fleischhacker M, Schmidt B. 2007; Circulating nucleic acids (CNAs) and cancer -a survey. Biochim Biophys Acta. 1775:181–232. DOI: 10.1016/j.bbcan.2006.10.001. PMID: 17137717.
23. Meddeb R, Pisareva E, Thierry AR. 2019; Guidelines for the preanalytical conditions for analyzing circulating cell-free DNA. Clin Chem. 65:623–33. DOI: 10.1373/clinchem.2018.298323. PMID: 30792266.

24. Shin S, Woo HI, Kim JW, Kim Y, Lee KA. 2022; Clinical practice guidelines for pre-analytical procedures of plasma epidermal growth factor receptor variant testing. Ann Lab Med. 42:141–9. DOI: 10.3343/alm.2022.42.2.141. PMID: 34635607. PMCID: PMC8548242.

25. El Messaoudi S, Rolet F, Mouliere F, Thierry AR. 2013; Circulating cell free DNA: Preanalytical considerations. Clin Chim Acta. 424:222–30. DOI: 10.1016/j.cca.2013.05.022. PMID: 23727028.

26. Martignano F. 2019; Cell-free DNA: An overview of sample types and isolation procedures. Methods Mol Biol. 1909:13–27. DOI: 10.1007/978-1-4939-8973-7_2. PMID: 30580420.

27. Melkonyan HS, Feaver WJ, Meyer E, Scheinker V, Shekhtman EM, Xin Z, et al. 2008; Transrenal nucleic acids: from proof of principle to clinical tests. Ann N Y Acad Sci. 1137:73–81. DOI: 10.1196/annals.1448.015. PMID: 18837928.

28. Tsui NB, Jiang P, Chow KC, Su X, Leung TY, Sun H, et al. 2012; High resolution size analysis of fetal DNA in the urine of pregnant women by paired-end massively parallel sequencing. PLoS One. 7:e48319. DOI: 10.1371/journal.pone.0048319. PMID: 23118982. PMCID: PMC3485143.

29. Yao W, Mei C, Nan X, Hui L. 2016; Evaluation and comparison of in vitro degradation kinetics of DNA in serum, urine and saliva: A qualitative study. Gene. 590:142–8. DOI: 10.1016/j.gene.2016.06.033. PMID: 27317895.

30. Bobillo S, Crespo M, Escudero L, Mayor R, Raheja P, Carpio C, et al. 2021; Cell free circulating tumor DNA in cerebrospinal fluid detects and monitors central nervous system involvement of B-cell lymphomas. Haematologica. 106:513–21. DOI: 10.3324/haematol.2019.241208. PMID: 32079701. PMCID: PMC7849551.

31. De Mattos-Arruda L, Mayor R, Ng CKY, Weigelt B, Martínez-Ricarte F, Torrejon D, et al. 2015; Cerebrospinal fluid-derived circulating tumour DNA better represents the genomic alterations of brain tumours than plasma. Nat Commun. 6:8839. DOI: 10.1038/ncomms9839. PMID: 26554728. PMCID: PMC5426516.

32. Kim Y, Shin S, Lee KA. 2021; Exosome-based detection of EGFR T790M in plasma and pleural fluid of prospectively enrolled non-small cell lung cancer patients after first-line tyrosine kinase inhibitor therapy. Cancer Cell Int. 21:50. DOI: 10.1186/s12935-021-01761-x. PMID: 33435996. PMCID: PMC7802208.
33. Asano H, Toyooka S, Tokumo M, Ichimura K, Aoe K, Ito S, et al. 2006; Detection of EGFR gene mutation in lung cancer by mutant-enriched polymerase chain reaction assay. Clin Cancer Res. 12:43–8. DOI: 10.1158/1078-0432.CCR-05-0934. PMID: 16397022.
34. Azuara D, Ginesta MM, Gausachs M, Rodriguez-Moranta F, Fabregat J, Busquets J, et al. 2012; Nanofluidic digital PCR for KRAS mutation detection and quantification in gastrointestinal cancer. Clin Chem. 58:1332–41. DOI: 10.1373/clinchem.2012.186577. PMID: 22745110.
35. Taly V, Pekin D, Benhaim L, Kotsopoulos SK, Le Corre D, Li X, et al. 2013; Multiplex picodroplet digital PCR to detect KRAS mutations in circulating DNA from the plasma of colorectal cancer patients. Clin Chem. 59:1722–31. DOI: 10.1373/clinchem.2013.206359. PMID: 23938455.
36. Kinde I, Wu J, Papadopoulos N, Kinzler KW, Vogelstein B. 2011; Detection and quantification of rare mutations with massively parallel sequencing. Proc Natl Acad Sci U S A. 108:9530–5. DOI: 10.1073/pnas.1105422108. PMID: 21586637. PMCID: PMC3111315.

37. Mosele F, Remon J, Mateo J, Westphalen CB, Barlesi F, Lolkema MP, et al. 2020; Recommendations for the use of next-generation sequencing (NGS) for patients with metastatic cancers: a report from the ESMO Precision Medicine Working Group. Ann Oncol. 31:1491–505. DOI: 10.1016/j.annonc.2020.07.014. PMID: 32853681.

38. Filipska M, Rosell R. 2021; Mutated circulating tumor DNA as a liquid biopsy in lung cancer detection and treatment. Mol Oncol. 15:1667–82. DOI: 10.1002/1878-0261.12983. PMID: 33969622. PMCID: PMC8169447.

39. Rolfo C, Mack P, Scagliotti GV, Aggarwal C, Arcila ME, Barlesi F, et al. 2021; Liquid Biopsy for Advanced NSCLC: A Consensus Statement From the International Association for the Study of Lung Cancer. J Thorac Oncol. 16:1647–62. DOI: 10.1016/j.jtho.2021.06.017. PMID: 34246791.

40. Litwin MS, Tan HJ. 2017; The diagnosis and treatment of prostate cancer: A review. JAMA. 317:2532–42. DOI: 10.1001/jama.2017.7248. PMID: 28655021.
41. de Bono J, Mateo J, Fizazi K, Saad F, Shore N, Sandhu S, et al. 2020; Olaparib for metastatic castration-resistant prostate cancer. N Engl J Med. 382:2091–102. DOI: 10.1056/NEJMoa1911440. PMID: 32343890.

42. Berruti A, Dogliotti L, Bitossi R, Fasolis G, Gorzegno G, Bellina M, et al. 2000; Incidence of skeletal complications in patients with bone metastatic prostate cancer and hormone refractory disease: predictive role of bone resorption and formation markers evaluated at baseline. J Urol. 164:1248–53. DOI: 10.1016/S0022-5347(05)67149-2.

43. Bubendorf L, Schöpfer A, Wagner U, Sauter G, Moch H, Willi N, et al. 2000; Metastatic patterns of prostate cancer: an autopsy study of 1,589 patients. Hum Pathol. 31:578–83. DOI: 10.1053/hp.2000.6698. PMID: 10836297.

44. Dang HX, Chauhan PS, Ellis H, Feng W, Harris PK, Smith G, et al. 2020; Cell-free DNA alterations in the AR enhancer and locus predict resistance to AR-directed therapy in patients with metastatic prostate cancer. JCO Precis Oncol. 4:680–713. DOI: 10.1200/PO.20.00047. PMID: 32903952. PMCID: PMC7446541.
45. Lau E, McCoy P, Reeves F, Chow K, Clarkson M, Kwan EM, et al. 2020; Detection of ctDNA in plasma of patients with clinically localised prostate cancer is associated with rapid disease progression. Genome Med. 12:72. DOI: 10.1186/s13073-020-00770-1. PMID: 32807235. PMCID: PMC7430029.

46. Shaya J, Nonato T, Cabal A, Randall JM, Millard F, Stewart T, et al. 2021; Analysis of the prognostic significance of circulating tumor DNA in metastatic castrate resistant prostate cancer. Clin Genitourin Cancer. 19:564.e1–e10. DOI: 10.1016/j.clgc.2021.07.012. PMID: 34452870.

47. Hong S, Won YJ, Lee JJ, Jung KW, Kong HJ, Im JS, et al. 2021; Cancer statistics in Korea: incidence, mortality, survival, and prevalence in 2018. Cancer Res Treat. 53:301–15. DOI: 10.4143/crt.2021.291. PMID: 33735559. PMCID: PMC8053867.

48. Kim Y, Jun JK, Choi KS, Lee HY, Park EC. 2011; Overview of the national cancer screening programme and the cancer screening status in Korea. Asian Pac J Cancer Prev. 12:725–30.
49. Dasari A, Morris VK, Allegra CJ, Atreya C, Benson AB 3rd, Boland P, et al. 2020; ctDNA applications and integration in colorectal cancer: an NCI Colon and Rectal-Anal Task Forces whitepaper. Nat Rev Clin Oncol. 17:757–70. DOI: 10.1038/s41571-020-0392-0. PMID: 32632268. PMCID: PMC7790747.

50. Flamini E, Mercatali L, Nanni O, Calistri D, Nunziatini R, Zoli W, et al. 2006; Free DNA and carcinoembryonic antigen serum levels: an important combination for diagnosis of colorectal cancer. Clin Cancer Res. 12:6985–8. DOI: 10.1158/1078-0432.CCR-06-1931. PMID: 17145818.

51. Luo H, Zhao Q, Wei W, Zheng L, Yi S, Li G, et al. 2020; Circulating tumor DNA methylation profiles enable early diagnosis, prognosis prediction, and screening for colorectal cancer. Sci Transl Med. 12:eaax7533. DOI: 10.1126/scitranslmed.aax7533. PMID: 31894106.

52. Tie J, Wang Y, Springer S, Kinde I, Wong HL, Kosmider S, et al. 2016; Serial circulating tumor DNA (ctDNA) and recurrence risk in patients (pts) with resectable colorectal liver metastasis (CLM). J Clin Oncol. 34(S):e15131–e15131. DOI: 10.1200/JCO.2016.34.15_suppl.e15131.

53. Venook AP, Niedzwiecki D, Lenz HJ, Innocenti F, Fruth B, Meyerhardt JA, et al. 2017; Effect of first-line chemotherapy combined with cetuximab or bevacizumab on overall survival in patients with KRAS wild-type advanced or metastatic colorectal cancer: A randomized clinical trial. JAMA. 317:2392–401. DOI: 10.1001/jama.2017.7105. PMID: 28632865. PMCID: PMC5545896.

54. Kopetz S, Grothey A, Yaeger R, Van Cutsem E, Desai J, Yoshino T, et al. 2019; Encorafenib, binimetinib, and cetuximab in BRAF V600E-mutated colorectal cancer. N Engl J Med. 381:1632–43. DOI: 10.1056/NEJMoa1908075. PMID: 31566309.

55. Meric-Bernstam F, Hurwitz H, Raghav KPS, McWilliams RR, Fakih M, VanderWalde A, et al. 2019; Pertuzumab plus trastuzumab for HER2-amplified metastatic colorectal cancer (MyPathway): an updated report from a multicentre, open-label, phase 2a, multiple basket study. Lancet Oncol. 20:518–30. DOI: 10.1016/S1470-2045(18)30904-5.
56. Tay TKY, Tan PH. 2021; Liquid biopsy in breast cancer: A focused review. Arch Pathol Lab Med. 145:678–86. DOI: 10.5858/arpa.2019-0559-RA. PMID: 32045277.

57. Magbanua MJM, Swigart LB, Wu HT, Hirst GL, Yau C, Wolf DM, et al. 2021; Circulating tumor DNA in neoadjuvant-treated breast cancer reflects response and survival. Ann Oncol. 32:229–39. DOI: 10.1016/j.annonc.2020.11.007. PMID: 33232761. PMCID: PMC9348585.

58. Chopra N, Tovey H, Pearson A, Cutts R, Toms C, Proszek P, et al. 2020; Homologous recombination DNA repair deficiency and PARP inhibition activity in primary triple negative breast cancer. Nat Commun. 11:2662. DOI: 10.1038/s41467-020-16142-7. PMID: 32471999. PMCID: PMC7260192.

59. Rose M, Burgess JT, O'Byrne K, Richard DJ, Bolderson E. PARP inhibitors: Clinical relevance, mechanisms of action and tumor resistance. Front Cell Dev Biol. 2020; 8:564601. DOI: 10.3389/fcell.2020.564601. PMID: 33015058. PMCID: PMC7509090.

60. Weigelt B, Comino-Méndez I, de Bruijn I, Tian L, Meisel JL, García-Murillas I, et al. 2017; Diverse BRCA1 and BRCA2 reversion mutations in circulating cell-free DNA of therapy-resistant breast or ovarian cancer. Clin Cancer Res. 23:6708–20. DOI: 10.1158/1078-0432.CCR-17-0544. PMID: 28765325. PMCID: PMC5728372.
61. Cheng ML, Pectasides E, Hanna GJ, Parsons HA, Choudhury AD, Oxnard GR. 2021; Circulating tumor DNA in advanced solid tumors: Clinical relevance and future directions. CA Cancer J Clin. 71:176–90. DOI: 10.3322/caac.21650. PMID: 33165928.

62. Ballehaninna UK, Chamberlain RS. 2012; The clinical utility of serum CA 19-9 in the diagnosis, prognosis and management of pancreatic adenocarcinoma: An evidence based appraisal. J Gastrointest Oncol. 3:105–19.
63. Sivapalan L, Kocher HM, Ross-Adams H, Chelala C. 2021; Molecular profiling of ctDNA in pancreatic cancer: Opportunities and challenges for clinical application. Pancreatology. 21:363–78. DOI: 10.1016/j.pan.2020.12.017. PMID: 33451936. PMCID: PMC7994018.

64. Grunvald MW, Jacobson RA, Kuzel TM, Pappas SG, Masood A. 2020; Current status of circulating tumor DNA liquid biopsy in pancreatic cancer. Int J Mol Sci. 21:7651. DOI: 10.3390/ijms21207651. PMID: 33081107. PMCID: PMC7589736.

65. Giornelli GH. 2016; Management of relapsed ovarian cancer: a review. Springerplus. 5:1197. DOI: 10.1186/s40064-016-2660-0. PMID: 27516935. PMCID: PMC4963348.

66. Asante DB, Calapre L, Ziman M, Meniawy TM, Gray ES. 2020; Liquid biopsy in ovarian cancer using circulating tumor DNA and cells: Ready for prime time? Cancer Lett. 468:59–71. DOI: 10.1016/j.canlet.2019.10.014. PMID: 31610267.

67. Vargas AN. 2014; Natural history of ovarian cancer. Ecancermedicalscience. 8:465.
68. Bogani G, Matteucci L, Tamberi S, Ditto A, Sabatucci I, Murgia F, et al. 2019; RECIST 1.1 criteria predict recurrence-free survival in advanced ovarian cancer submitted to neoadjuvant chemotherapy. Eur J Obstet Gynecol Reprod Biol. 237:93–9. DOI: 10.1016/j.ejogrb.2019.04.007. PMID: 31029972.

69. Charkhchi P, Cybulski C, Gronwald J, Wong FO, Narod SA, Akbari MR. 2020; CA125 and ovarian cancer: A comprehensive review. Cancers (Basel). 12:3730. DOI: 10.3390/cancers12123730. PMID: 33322519. PMCID: PMC7763876.

70. Hentze JL, Høgdall C, Kjær SK, Blaakær J, Høgdall E. 2017; Searching for new biomarkers in ovarian cancer patients: Rationale and design of a retrospective study under the Mermaid III project. Contemp Clin Trials Commun. 8:167–74. DOI: 10.1016/j.conctc.2017.10.003. PMID: 29696206. PMCID: PMC5898550.

71. Asaoka Y, Ijichi H, Koike K. 2015; PD-1 Blockade in tumors with mismatch-repair deficiency. N Engl J Med. 373:1979. DOI: 10.1056/NEJMc1510353.

72. Georgiadis A, Durham JN, Keefer LA, Bartlett BR, Zielonka M, Murphy D, et al. 2019; Non-invasive detection of microsatellite instability and high tumor mutation burden in cancer patients treated with PD-1 blockade. Clin Cancer Res. 25:7024–34. DOI: 10.1158/1078-0432.CCR-19-1372. PMID: 31506389. PMCID: PMC6892397.

73. Friedlaender A, Nouspikel T, Christinat Y, Ho L, McKee T, Addeo A. 2020; Tissue-plasma TMB comparison and plasma TMB monitoring in patients with metastatic non-small cell lung cancer receiving immune checkpoint inhibitors. Front Oncol. 10:142. DOI: 10.3389/fonc.2020.00142. PMID: 32117779. PMCID: PMC7028749.

74. Qiu P, Poehlein CH, Marton MJ, Laterza OF, Levitan D. 2019; Measuring tumor mutational burden (TMB) in plasma from mCRPC patients using two commercial NGS assays. Sci Rep. 9:114. DOI: 10.1038/s41598-018-37128-y. PMID: 30643180. PMCID: PMC6331610.

75. Miyazawa H, Tanaka T, Nagai Y, Matsuoka M, Sutani A, Udagawa K, et al. 2008; Peptide nucleic acid-locked nucleic acid polymerase chain reaction clamp-based detection test for gefitinib-refractory T790M epidermal growth factor receptor mutation. Cancer Sci. 99:595–600. DOI: 10.1111/j.1349-7006.2007.00706.x. PMID: 18271876.

76. Freidin MB, Freydina DV, Leung M, Montero Fernandez A, Nicholson AG, Lim E. 2015; Circulating tumor DNA outperforms circulating tumor cells for KRAS mutation detection in thoracic malignancies. Clin Chem. 61:1299–304. DOI: 10.1373/clinchem.2015.242453. PMID: 26272233.
77. Sefrioui D, Sarafan-Vasseur N, Beaussire L, Baretti M, Gangloff A, Blan-chard F, et al. 2015; Clinical value of chip-based digital-PCR platform for the detection of circulating DNA in metastatic colorectal cancer. Dig Liver Dis. 47:884–90. DOI: 10.1016/j.dld.2015.05.023. PMID: 26160500.

78. Taniguchi K, Uchida J, Nishino K, Kumagai T, Okuyama T, Okami J, et al. 2011; Quantitative detection of EGFR mutations in circulating tumor DNA derived from lung adenocarcinomas. Clin Cancer Res. 17:7808–15. DOI: 10.1158/1078-0432.CCR-11-1712. PMID: 21976538.
79. Newman AM, Bratman SV, To J, Wynne JF, Eclov NCW, Modlin LA, et al. 2014; An ultrasensitive method for quantitating circulating tumor DNA with broad patient coverage. Nat Med. 20:548–54. DOI: 10.1038/nm.3519. PMID: 24705333. PMCID: PMC4016134.

80. Gale D, Lawson ARJ, Howarth K, Madi M, Durham B, Smalley S, et al. 2018; Development of a highly sensitive liquid biopsy platform to detect clinically-relevant cancer mutations at low allele fractions in cell-free DNA. PLoS One. 13:e0194630. DOI: 10.1371/journal.pone.0194630. PMID: 29547634. PMCID: PMC5856404.

81. Narayan A, Carriero NJ, Gettinger SN, Kluytenaar J, Kozak KR, Yock TI, et al. 2012; Ultrasensitive measurement of hotspot mutations in tumor DNA in blood using error-suppressed multiplexed deep sequencing. Cancer Res. 72:3492–8. DOI: 10.1158/0008-5472.CAN-11-4037. PMID: 22581825. PMCID: PMC3426449.

82. Dai P, Wu LR, Chen SX, Wang MX, Cheng LY, Zhang JX, et al. 2021; Calibration-free NGS quantitation of mutations below 0.01% VAF. Nat Commun. 12:6123. DOI: 10.1038/s41467-021-26308-6. PMID: 34675197. PMCID: PMC8531361.

Fig. 3
Representative patient case (ovarian cancer) showing the feasibility of ctDNA analysis. The patient underwent debulking surgery and chemotherapy after diagnosis. The increased ctDNA levels were detected four months earlier than conventional diagnostic methods, showing that ctDNA could detect cancer recurrence earlier than CA-125 and PET-CT scans.
Abbreviations: VAF, variant allele frequency; PET-CT, positron emission tomography-computed tomography; CA-125, carbohydrate antigen 125; PARPi, poly (ADP-ribose) polymerase inhibitor.
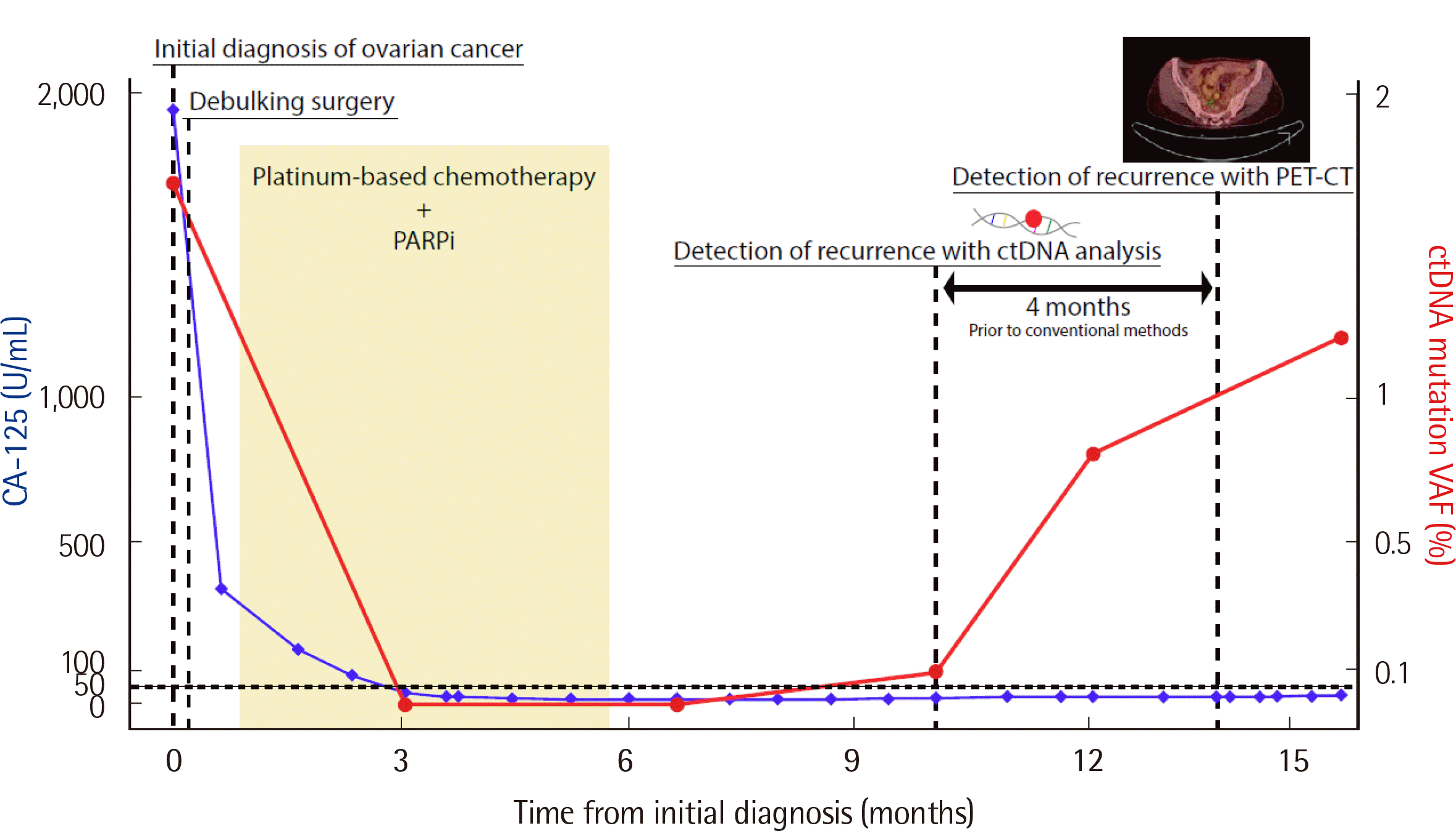
Table 1
Characteristics of the techniques used for circulating tumor DNA analysis
| Technique | Target | Types of mutations that can be detected | Analytical sensitivity | Relative cost | Relative turn-around time |
|---|---|---|---|---|---|
| Real-time PCR | Known mutations | SNV, indel | 0.1–5% [75, 76] | Low | Fast |
| Digital droplet PCR | Known mutations | SNV, indel, CNV | 0.01–1% [34, 35, 77, 78] | Low | Fast |
| Next-generation sequencing | Known/unknown mutations | SNV, indel, CNV, gene fusion | 0.01–5% [79-82] | High | Long |




 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download