Abstract
Background/Aims
There is a recent increase in the use of stool multiplex PCR assay-based diagnostic tests in patients with acute diarrhea. We used multiplex PCR assays to analyze the distribution of diarrhea-causing bacteria and viruses, as well as the clinical features of patients with acute diarrhea.
Methods
We retrospectively reviewed stool specimens of inpatients complaining of acute diarrhea from October 2018 to July 2020. The stool specimens had been tested for bacteria and viruses using multiplex PCR assays.
Results
A total of 414 stool specimens from 346 patients were tested, and 152 pathogens were detected in 131 stool samples (131/414, 31.6%). Co-infection was detected in 20 patients (20/346, 5.8%). The common pathogens detected as causes of acute diarrhea, including co-infection, were Clostridium perfringens (34.9%), Clostridioides difficile (19.7%), and Campylobacter spp. (18.4%). The average age of patients with multiplex PCR-positive tests was lower than those with multiplex PCR-negative tests (p=0.001). In patients with suspected C. difficile infection (CDI), the RT-PCR for toxin gene assay was performed in 370 stool samples, 35 of which were positive (9.5%). Furthermore, 16 of the 35 samples were positive on the multiplex PCR assay (45.7%).
감염성 설사는 매년 전 인구의 10명 중 1명에서 발생하는 흔한 질환이다.1 감염성 설사는 개발도상국에서 불충분한 위생과 연관되어 흔히 발생하는 것으로 알려져 있지만, 경제적으로 발전된 국가에서도 여행, 식량 수입 및 유통, 상수도 시스템, 아이 돌봄, 간병, 입원 등 여러 요인에 의해 장내 미생물이 전파되어 발생하기도 하며, 오염된 음식물과 물에 의해 집단적으로도 발생한다.2 병원에 내원하는 급성 설사 환자는 감염성 원인이 의심되면 필요에 따라 대변 검사를 시행하게 된다. 최종 진단을 위해 시행하는 대변 배양 검사는 고전적인 검사 방법으로 급성 설사 환자에서 흔하게 시행되어 왔다.3 그러나 이러한 대변 배양 검사는 검사 결과가 나오기까지 수일이 소요되며, 배양하는 조건이 까다로워 실제 균이 있어도 음성이 나오는 경우가 많은 단점이 있다.4
최근 중합효소연쇄반응 검사와 같은 분자단위의 진단기법이 대변 검체에도 적용되어 많은 장내 병원체 검출에 대한 진단 민감도를 향상시켰다.5 특히 최근 출시된 단일 대변 검체에서 복수의 설사 유발 병원체를 동시에 검출할 수 있는 다중중합효소연쇄반응 패널(multiplex PCR assays)은 급성 설사 증상이 있는 환자에서 세균과 바이러스를 높은 민감도로 빠르게 검출할 수 있다는 장점이 있다. 그중 Seeplex® Diarrhea-B1 ACE detection assay (Seegene, Seoul, Korea)는 다중중합효소연쇄반응 검사로 Clostridioides difficile (C. difficile) 및 기타 장내 병원체(Salmonella, Shigella, Vibrio, Campylobacter spp.) 특이 프라이머를 사용한다. 이번 연구에서는 급성 설사 환자를 대상으로 다중중합효소연쇄반응 검사를 이용해 실제 임상에서 세균과 바이러스를 검출하는 데 있어 분포와 임상 양상을 분석하였다. 또한 최근 많은 검사실에서 시행하는 C. difficile의 tcdB 검출을 위한 PCR 분석 방법인 Xpert C. difficile assay (Cepheid, Sunnyvale, CA, USA)와 비교하여 C. difficile을 대상으로 한 Seeplex® Diarrhea-B1 ACE 검사의 유용성을 함께 확인하였다.
2018년 10월부터 2020년 7월까지 인제대학교 서울백병원에 급성 설사를 호소하여 입원하여 대변 다중중합효소연쇄반응 검사를 시행한 만 19세 이상의 성인 환자의 의무기록을 후향적으로 수집하였다. 총 346명의 환자에서 채취된 총 414개의 검체를 대상으로 하였다. 설사는 하루에 대변량이 200 g을 넘거나 배변 횟수가 3회를 초과하는 경우로 정의하였으며, 그 기간이 14일 이내인 경우 급성 설사로 정의하였다.4,6 검체는 4℃에서 보관되었고 검사실에서 받은 후 48시간 이내에 검사하였다. 또한, 혈변 혹은 점액변이 있거나, 발열이 있거나, 경련성 복통이 있는 환자는 경험적 항생제를 사용하였다. 본 연구는 후향적 관찰 단면 연구로서 서울백병원 연구윤리위원회의 승인을 받았으며(IRB No. 2020-10-004), 후향적 연구임을 감안하여 개별 환자에 관한 동의서는 면제되었다.
대변 검체에 대해 세가지 패널인 Seeplex® Diarrhea -B1 ACE detection kit, Diarreha -B2 ACE detection kit (DR6501Y/DR6502Y, Seegene), Allplex™ GI-Virus Assay (GI9701Y, Seegene)를 이용하여 제조사의 지침에 따라 시행되었다. 전처리된 대변 검체에서 총 핵산을 추출하였으며, ThermoCycler PCR System (SeeAMP™, Seegene)을 이용하여 동시 증폭 및 표적검출을 시행하였다.7
Bacteria Seeplex® diarrhea-B1/B2 ACE detection kit로 총 11종의 세균(Campylobacter spp., C. difficile toxin B, Salmonella spp., Shigella spp., Vibrio spp., Aeromonas spp., Clostridium perfringens (C. perfringens), Escherichia coli (E. coli) H7, E. coli O157, Verocytotoxin-producing E. coli, Yersinia enterocolitica)을 확인하였으며, Virus Allplex GI-Virus assay로 총 6종의 바이러스(Adenovirus, Astrovirus, Norovirus Group 1 (G1), Norovirus Group 2 (G2), Group A Rotavirus, Sapovirus)를 확인하였다.
전체 급성 설사 환자 총 346명에서 채취된 총 414개의 대변 검체를 대상으로 검사가 시행되었다. 그중 총 117명의 환자에서 채취된 검체 131개의 검체에서 다중중합효소연쇄반응 검사 양성(31.6%)이 나왔으며, 중복감염(co-infection)을 포함하면 총 152건의 미생물이 검출되었다(Fig. 1). 다중중합효소연쇄반응 검사 양성 환자군은 음성 환자군과 비교하여 평균연령이 낮았고(57.41±24.35 vs. 66.38±19.53, p=0.001), 양성 환자군은 음성 환자군과 비교하였을 때 고혈압, 당뇨, 간경화, 염증성 장질환의 유병률은 두 군 간 유의한 차이는 없었다(Table 1). 또한 다중중합효소연쇄반응 검사 양성 환자군은 음성 환자군과 비교하여 복통이 동반된 경우가 흔하였고(50.4% vs. 37.6%, p=0.011), 대변잠혈 양성인 경우가 더 많았으며(25.81% vs. 11.76%, p=0.002), 대변 백혈구가 나오는 경우가 더 많았다(18.87% vs. 4.20%, p<0.001).
다중중합효소연쇄반응에서 검출된 미생물은 중복감염을 포함하여 C. perfringens 53건(34.9%), C. difficile 30건(19.7%), Campylobacter spp. 28건(18.4%), Norovirus G2 18건(11.8%), Aeromonas spp. 10건(6.6%), Group A Rotavirus 4건(2.6%), Shigella spp. 3건(2.0%), Salmonella spp. 3건(2.0%)이었다. 그중 20건의 검체에서 중복감염(15.3%)이 확인되었다(Table 2).
총 346명의 환자군 중 급성 설사로 입원하여, 입원 후 48시간 이내 다중중합효소연쇄반응 검사를 진행하였고, 재원일수 20일 미만이며, 재입원한 경우의 환자를 제외한 총 159명(46.0%)에 대해 다중중합효소연쇄반응 검사 양성 여부, 재원일수, 항생제 사용 여부를 비교하였다. 재원일수는 다중중합효소연쇄반응 검사 양성군(n=73)은 음성군(n=86)에 비교해 유의한 차이는 없었다(6.60±3.17 vs. 7.13±3.67, p=0.340). 다중중합효소연쇄 반응 검사 양성군과 음성군 간에 항생제 치료 비율 역시 두 군 간의 유의한 차이는 없었다(p=0.162). 또한, 다중중합효소연쇄반응 검사 양성군 73명 내에서 항생제 치료군(n=55)을 항생제 치료 미시행군(n=18)과 비교하였을 때 평균 재원일수의 유의한 차이는 없었다(6.38±2.94 vs. 7.28±3.79, p=0.301). 그러나, 다중중합효소연쇄반응 검사 음성군(n=86) 내에서 항생제 치료군(n=56)과 항생제 치료 미시행군(n=30)을 비교하였을 때 평균 재원일수는 오히려 항생제 치료군이 더 길었다(7.73±3.61 vs. 6.00±3.59, p=0.036).
다중중합효소연쇄반응 검사에서 가장 많이 검출된 미생물은 C. perfringens 였으며, 검체 53개 중 11개는 중복감염이었다. 60세 이상 환자에서 감염률이 높았고(42건, 79%), 동반 질환으로 고혈압(22건, 41.5%), 당뇨(15건, 28.3%) 염증성 장질환(8건, 15.1%)이 관찰되었다. 복통이 동반된 환자의 경우 24건(45.3%)에서 양성이었으며, 발열이 동반된 환자는 6건(11.3%)에서 양성이었다. 대변 잠혈반응 검사를 시행한 45명 중 12명이 양성이었으며 대변 백혈구 검사를 시행한 51개의 검체 중 5건이 양성이었다. 평균 말초혈액 백혈구 검사는 8.78±5.38/mm3였으며, 평균 CRP는 4.8±6.7 mg/dL였다(Table 3).
다중중합효소연쇄반응 검사에서 두 번째로 높은 빈도로 검출된 미생물은 C. difficile 였다(30/152, 19.7%). 414건의 대변 검체 중 CDI가 의심되었던 370건의 검체(89.4%)에서 toxin B gene에 대한 실시간 중합효소연쇄반응 검사를 동시에 진행하였다. 370개 검체 중 실시간 중합효소연쇄반응 검사 양성은 총 35건이었으며(9.5%), 이들 중 다중중합효소연쇄반응 검사로 C. difficile 양성이 나온 건수는 16건(45.7%)이었다.
감염성 설사 질환은 나라별로 환경 및 생활양식, 상이한 음식 등에 의해 주된 원인 미생물 및 발생 빈도의 차이가 있다. 국내에서도 감염성 설사 질환이 최근 증가 추세에 있으며, 이는 육류 소비 및 익히지 않은 해산물의 섭취, 단체 사내 및 급식 운영 등으로 인한 집단 발생 등이 원인으로 보인다. 집단 발생의 경우 2000년대 후반 기준으로 Norovirus, E.coli, Salmonella spp. 등이 원인으로 나타났다.10
2012년 급성 설사 질환 감시사업으로 전국 21,180건의 설사 환자에서 수집된 분변 검체를 대상으로 검사한 결과, 병원체가 확인된 검체는 전체 검체의 11.5%인 2,444건이었다. 전체 양성 검체의 34.0%가 S. aureus로 가장 많았고, 이어서 E. coli (30.1%), Salmonella spp. (17%), B. cereus (7.1%)였으며, Campylobacter spp. (5.8%), C. perfringens (4.0%)가뒤를 이어 분포하였다.11 해당 자료는 식중독의 집단 발생이나바이러스를 제외한 결과이지만 그것을 감안하더라도 세균의분포 및 그 빈도에서 이번 연구와는 현격한 차이가 있었다. 그 이유는 선행 연구에서 고전적 대변 검사에서 가장 많은 분포를 보였던 S. aureus가 본 연구의 다중중합효소연쇄반응 검사 대상에 포함되지 않았다. 또한, 검사법의 차이도 주요 원인으로 판단된다. Campylobacter spp. 및 C. perfringens는 앞선 연구에서는 배양 후 중합효소연쇄반응 등 생화학적으로 최종 동정하였던 것에 반해, 이번 연구에서는 배양 과정 없이 바로 다중중합효소연쇄반응 검사를 시행하였다. 다른 연구에서는 실제로 Campylobacter spp.의 경우 대변 도말에서는 관찰되나 배양에서는 자라지 않는 경우가 흔하였으며,12 또 다른 연구에서도 Campylobacter spp. 음성이 나왔던 상당수의 검체에서 다중중합효소연쇄반응 검사는 양성으로 나와, 배양보다 다중중합효소연쇄반응 검사가 더 높은 민감도를 보였다.13
이번 연구에서는 급성 설사 환자의 414개의 대변 검체 중 다중중합효소연쇄반응 검사에서 131개의 검체(31.6%)에서 양성이 나왔으며, 이는 국내 다른 연구에서의 양성률 범위내(20.8-45.8%)였다.13-15 이는 2012년 급성 설사 질환 감시사업 당시 대변 배양 전체 검체 중 11.5%만이 양성이 나왔던 것에 비하면 괄목할 만한 변화이다.11 이렇듯 최근 다중중합효소연쇄반응 검사가 소화기 설사 환자에서 임상적으로 널리 쓰이게 되면서 기존 배양 검사보다 원인 미생물에 대한 진단 민감도가 상승하였으며, 검사 시간 또한 단축되면서 여러 이점이 생겼다.15-17 기존에 일반 배양으로는 진단이 어려우며 특수한 방법이 필요하였던 Vibrio spp., Y. enterocolitica, E. coli O157:H7, typhoidal salmonellosis와 같은 미생물도 다중중합효소연쇄반응 검사로 매우 민감하고 빠르게 확인이 가능해졌다. 다만 다중중합효소연쇄반응 검사에서 상재균이 확인되거나, 중복감염이 확인되었을 경우 환자의 설사의 원인 병원체를 결정하기 위해 임상의의 신중한 판단이 필요하다. 즉,여러 개의 병원체가 확인될 경우 각 병원체의 증상, 잠복기,현재 유행 여부 등을 종합적으로 고려하여 치료 방침을 판단하여야 한다.18 본 연구에서는 대상 환자군이 급성 설사와 동반된 증상으로 입원을 하였거나 입원 중인 환자였기 때문에다중중합효소연쇄반응 검사에서 단일 병원체 양성이 나온 환자들의 경우 단순 상재균보다는 증상을 유발시킨 병원체로 간주하였다. 본 연구는 다중중합효소연쇄반응 검사를 시행한 환자 중 비교 대상이 될 수 있는 환자를 일부 선택하여 항생제사용 여부 및 재원일수를 조사하였다. 본 연구 이전에 이미다중중합효소연쇄반응 검사를 이용한 선행 연구들이 많았고,이에 대한 메타분석 연구19도 있으나 미생물 분포 및 고전적검사와 비교가 주를 이뤘다. 임상의들의 주 관심사인 항생제사용 및 재원일수를 비교한 국내 연구는 없었다. 본 연구에서는 다중중합효소연쇄반응 검사를 시행한 급성 설사 환자군을선별하여 159명에 대해 항생제 사용 여부와 재원일수를 조사하였으며, 다중중합효소연쇄반응 검사 음성군의 경우 항생제 치료군이 항생제 치료 미시행군과 비교해 재원일수가 유의하게 길었다는 결과를 보여줘 다중중합효소연쇄반응 검사 결과 음성인 경우 항생제 치료 시행에 대해 면밀한 검토가 필요할 수 있음을 시사하였다. 다만, 항생제 치료군이 항생제 치료 미시행군과 비교해 임상적인 중증도가 심하여 재원기간이 길어졌을 가능성은 염두해야 한다. 대다수의 급성 설사는 특히 병원체가 동정이 되지 않았을 경우, 항생제 치료보다는 위생, 수분 공급, 동반 질환 여부 등이 더 중요할 수 있다. 물론 다중중합효소연쇄반응 검사 결과 단독으로 판단은 위험하다. 선행 메타분석 연구19에서 Salmonella spp.의 경우 오히려 다중중합효소연쇄반응 검사에서 고전적 배양에 비해 양성율이 낮았다. 발열, 심한 설사, 대변 백혈구 확인 등 전반적으로 Salmonella spp.를 배제할 수 없는 상황에서는 전파위험성이 있어 격리가 필요하므로, 임상 증상이 중한 환자들은 비록 다중중합효소연쇄반응 검사 음성이다 하더라도 추가적 배양 결과를 확인하기 전까지 항생제 치료를 고려하는 등 개별화 전략은 필요하다.
이번 연구에서 다중중합효소연쇄반응 검사에서 양성이 나온 환자군과 음성인 환자군을 비교하였을 때 음성인 환자군에서 평균연령이 높았는데, 이는 고령 환자군에서 세균이나 바이러스 감염보다 다른 원인에 의한 설사가 더 흔함을 시사한다. 조사한 동반 질환의 경우 다중중합효소연쇄반응 검사 양성군과 음성군 간의 평균연령이 유의하게 차이가 있어 로지스틱 회귀분석을 통해 연령에 대해 보정 후 동반 질환의 유병률을 비교하였을 때 두 군 간의 유의한 차이는 없었다. 미생물에 의한 장염이 염증성 장질환을 유발할 수 있다는 연구가 있어 동반 질환에 염증성 장질환군도 포함하여 비교를 해보았으나 본 연구에서는 양성군과 음성군 간의 유의한 차이는 없었다.20
이번 연구에서 다중중합효소연쇄반응을 통해 검출된 가장 높은 빈도의 미생물은 혐기성 간균인 C. perfringens였다. 미국 및 캐나다에서는 C. perfringens가 2010년대까지만해도 식중독의 두 번째로 흔한 원인균이었으며 최근 미국에서는 매년 1백만 명 가까이 C. perfringens로 인한 식중독이 나타나는 것으로 조사되었다.21,22 C. perfringens는 익히지 않은 음식 또는 혐기성 조건에서 요리된 음식에 주로 포자형태로 생존하며,23 인체에 들어가면 A, B, C, D, E형의 5유형 중 주로 A형에서 주로 생산하는 알파독소에 의해 식중독이 일어난다.24 대변 검사를 통해 진단될 수 있으나 C. difficile과 마찬가지로 장내 정상세균으로 존재할 수 있어 다중중합효소연쇄반응 검사 양성이 나온 환자의 경우 바로 설사의 원인으로 단정짓기보다는 임상 증상과 연관하여 생각하여야 한다. 예를 들면, 급성 설사로 입원한 C. perfringens 단일 양성 환자에서는 C. perfringens를 그 원인으로 추정할 수 있지만, 중복감염에서 C. perfringens가 검출되면 동시 검출된 상대 병원체에 비해 원인 병원체로서의 가치가 떨어질 수 있다. 이번 연구에서는 총 20건의 중복감염 검체 중 11건의 검체에서 C. perfringens가 검출되었으며, 이러한 중복감염의 경우 C. perfringens보다 타 병원체를 급성 설사의 원인으로 생각할 수 있다.
이번 연구에서 Norovirus는 바이러스와 세균을 포함하여 빈도수 4위(19/152, 12.5%)에 머물렀다. 미국에서 9.4백만 식중독 환자를 대상으로 31가지 병원체에 대해 조사한 대규모 연구가 있었는데, 이 연구에서 가장 흔한 식중독의 원인은 Norovirus (58%)였으며 이어서 nontyphoidal Salmonella spp. (11%), C. perfringens (10%), Campylobacter spp. (9%) 순이었다.22 반면 식중독으로 인해 입원까지 한 경우로 한정하면 nontyphoidal Salmonella spp. (35%), Norovirus (26%), Campylobacter spp. (15%)로 빈도의 차이가 있었다. 이번 연구는 입원 환자를 대상으로 분석한 것으로 Norovirus 환자는 상대적으로 증상이 경하여 대상 환자군에 적게 포함되었을 것으로 추정된다.
또한, 이번 연구에서 C. difficile 가능성이 있는 급성 설사 환자에게 시행한 370개 검체를 대상으로 다중중합효소연쇄반응 검사와 C. difficile toxin B에 대한 실시간 중합효소연쇄반응 검사를 함께 검사하여 결과를 비교하였다. 370개 검체 중 실시간 중합효소연쇄반응 검사 양성은 총 35건이었으며 이들 중 다중중합효소연쇄반응 검사로 C. difficile 양성이 나온 건수는 16건으로 일치율이 45.7%였다. 이와 같은 결과는 CDI 가능성이 있는 급성 설사 환자에서는 보다 진단 확률을 높이기 위해 다중중합효소연쇄반응 검사뿐만 아니라 기존 CDI 진단 방법을 병행하는 것이 도움이 될 것으로 보인다. 그중 toxin B gene에 대한 실시간 중합효소연쇄반응 검사는 CDI 진단 민감도와 특이도가 모두 95%가 넘고, 결과를 얻기까지 평균 2.27시간이 소요되는 매우 정확하고 신속한 도구이다.9,25
또한, 본 연구에서는 Campylobacter spp.는 검출된 환자의 반수 이상에서 발열이 관찰되었다. Campylobacter spp., Salmonella spp., Shigella spp.에 의한 감염성 설사에서 발열은 흔한 임상 양상으로 알려져 있으나,26,27 Salmonella spp., Shigella spp.는 충분한 검체 수가 확보되지 않아 두 세균의 발열의 빈도를 분석하기에 한계가 있었다. 대변 백혈구 검사는 염증성 유무를 알 수 있는 비교적 저렴하고 신속한 검사이며, 설사 환자의 입원 기간 예측 인자라는 연구도 있다.28 본 연구에서는 세균성 설사 환자가 바이러스성 설사 환자보다 대변 백혈구 양성 소견이 다수였다.
이번 연구의 제한점은 후향적으로 자료를 분석하였기에 대변검사를 시행하였지만 임상자료가 충분치 않았던 환자의 임상 특징을 분석할 수 없었고, 증상이 심하지 않아 입원 치료가 필요하지 않거나, 입원을 원하지 않았던 외래 환자는 대상에 포함되지 못하였다. 또한, 이번 연구가 국내 성인 급성 설사 입원 환자의 최근 병원체의 분포를 살펴본 역학 연구이지만, S. aureus가 검사 대상 병원체에 포함되지 않은 제한점이 있다.
진단 검사의 발전으로 급성 설사 환자에서 검출되는 미생물 분포와 임상 양상이 변화하였다. 이전 국내에서 시행한 대변 배양 검사를 이용한 연구6에서 S. aureus와 E. coli가 흔한 소견이었던 것11과는 달리, 이번 연구에서는 C. perfringens가 가장 흔하게 검출되었고 C. difficile, Campylobacter spp.가 그 뒤를 따랐다. 이러한 분포는 다중중합효소연쇄반응 검사를 이용한 국내 다른 연구와 비슷하였다.13,16 하지만 CDI가 의심되는 환자에서는 다중중합효소연쇄반응 검사와 C. difficile toxin에 대한 실시간 중합효소연쇄반응 검사의 일치율이 낮아 기존의 CDI 검사가 여전히 필요함을 알 수 있었다. 또한, 다중중합효소연쇄반응 검사 시행군의 항생제 사용과 재원일수 비교를 통하여 급성 설사 환자의 진료 시 다중중합효소연쇄반응 검사로 원인 미생물에 대한 신속한 진단 및 알맞은 치료를 시행할 수 있다는 유용성을 확인하였다.
목적: 최근 급성 설사 환자에서 대변 다중중합연쇄반응 검사의 이용이 증가하고 있다. 이에 다중중합효소연쇄반응 검사를 이용하여 설사를 유발하는 세균과 바이러스의 분포와 급성 설사 환자의 임상 양상을 알아보고자 한다.
대상 및 방법: 일개 대학병원에서 입원한 급성 설사 환자의대변 검체 자료를 후향적으로 수집하였다. 대변 검체는 세균과 바이러스 검출을 위해 다중중합효소연쇄반응 검사를 시행하였다.
결과: 2018년 10월부터 2020년 7월까지 일개 대학병원에서입원한 급성 설사 환자 346명으로부터 총 414개의 대변 검체를검사하였고, 131건(31.6%)에서 양성이 나왔다. 동시 감염을포함한 급성 설사의 원인으로 가장 흔한 미생물은 Clostridium perfringens (C. perfringens) (34.9%), Clostridioides difficile (C. difficile) (19.7%) 그리고 Campylobacter spp. (18.4%)순이었다. CDI가 의심되는 환자의 370개의 대변 검체에서 toxin B gene에 대한 실시간 중합효소연쇄반응 검사를 동시에진행하였고, 총 35건(9.5%)에서 실시간 중합효소연쇄반응 검사 양성이었으며, 이들 중 다중중합효소연쇄반응 검사로 C. difficile 양성이 나온 건수는 16건(45.7%)이었다.
결론: 다중중합효소연쇄반응 검사 결과 C. perfringens 는 가장 흔한 급성 설사 원인균이었다. 또한, CDI가 의심되는 환자의 경우 다중중합효소연쇄반응 검사만으로는 병원체를 진단하는 데 어려움이 있어 기존의 CDI 검사를 추가로 시행할 필요가 있다.
색인단어: 설사; 다중중합효소연쇄반응; 세균; 바이러스
REFERENCES
1. WHO estimates of the global burden of foodborne diseases: foodborne disease burden epidemiology reference group 2007-2015. [Internet]. Geneva: World Health Organization;2015. cited 2021 Jul 1. Available from: https://apps.who.int/iris/handle/10665/199350.
2. Shane AL, Mody RK, Crump JA, et al. 2017; 2017 Infectious Diseases Society of America clinical practice guidelines for the diagnosis and management of infectious diarrhea. Clin Infect Dis. 65:1963–1973. DOI: 10.1093/cid/cix959. PMID: 29194529. PMCID: PMC5848254.

3. DuPont HL. 2014; Acute infectious diarrhea in immunocompetent adults. N Engl J Med. 370:1532–1540. DOI: 10.1056/NEJMra1301069. PMID: 24738670.

4. Guerrant RL, Van Gilder T, Steiner TS, et al. 2001; Practice guidelines for the management of infectious diarrhea. Clin Infect Dis. 32:331–351. DOI: 10.1086/318514. PMID: 11170940.

5. Drancourt M. 2014; Multiplex testing of diarrhoea breaks down microbial barriers. Lancet Infect Dis. 14:663–664. DOI: 10.1016/S1473-3099(14)70844-8. PMID: 25022436.

6. Camilleri M. 2004; Chronic diarrhea: a review on pathophysiology and management for the clinical gastroenterologist. Clin Gastroenterol Hepatol. 2:198–206. DOI: 10.1016/s1542-3565(04)00003-5. PMID: 15017602.

7. Martín A, Pérez-Ayala A, Chaves F, Lora D, Orellana MÁ. 2018; Evaluation of the multiplex PCR Allplex-GI assay in the detection of bacterial pathogens in diarrheic stool samples. J Microbiol Methods. 144:33–36. DOI: 10.1016/j.mimet.2017.10.016. PMID: 29100990.

8. Owens RC Jr, Donskey CJ, Gaynes RP, Loo VG, Muto CA. 2008; Antimicrobial-associated risk factors for Clostridium difficile infection. Clin Infect Dis. 46(Suppl 1):S19–S31. DOI: 10.1086/521859. PMID: 18177218.
9. Song PH, Min JH, Kim YS, et al. 2018; Rapid and accurate diagnosis of Clostridium difficile infection by real-time polymerase chain reaction. Intest Res. 16:109–115. DOI: 10.5217/ir.2018.16.1.109. PMID: 29422805. PMCID: PMC5797257.

10. Cho SH, Shin HH, Choi YH, Park MS, Lee BK. 2008; Enteric bacteria isolated from acute diarrheal patients in the Republic of Korea between the year 2004 and 2006. J Microbiol. 46:325–330. DOI: 10.1007/s12275-008-0015-4. PMID: 18604503.

11. Kim NO, Cha I, Kim JS, Chung GT, Kang YH, Hong S. 2013; The prevalence and characteristics of bacteria causing acute diarrhea in Korea, 2012. Ann Clin Microbiol. 16:174–181.

12. Bessède E, Delcamp A, Sifré E, Buissonnière A, Mégraud F. 2011; New methods for detection of campylobacters in stool samples in comparison to culture. J Clin Microbiol. 49:941–944. DOI: 10.1128/JCM.01489-10. PMID: 21209172. PMCID: PMC3067684.

13. Lee J, Kim J, Cho H, Oh K, Uh Y, Yoon KJ. 2015; Detection of bacterial and viral pathogens in stool specimens using multiplex PCR. Lab Med Qual Assur. 37:141–147.

14. Lee S, Park YJ, Lee HK, et al. 2013; Detection of 13 enteric bacteria and 5 viruses causing acute infectious diarrhea using multiplex PCR from direct stool specimens. Ann Clin Microbiol. 16:33–38.

15. Cho MC, Noh SA, Kim MN, Kim KM. 2010; Direct application of multiplex PCR on stool specimens for detection of enteropathogenic bacteria. Korean J Clin Microbiol. 13:162–168.

16. Kwack WG, Lim YJ, Kwon KH, Chung JW, Oh JY. 2020; Outcomes and clinical relevance of stool multiplex bacterial polymerase chain reaction in patients with acute diarrhea: single center experience. Korean J Intern Med. 35:300–309. DOI: 10.3904/kjim.2017.189. PMID: 30685964. PMCID: PMC7061005.

17. Park JO, Jeon JS, Kim JK. 2019; Epidemiologic trends of diarrhea-causing virus infection analyzed by multiplex reverse transcription PCR in Cheonan, Korea, 2010-2018. Microbiol Biotechnol Lett. 47:317–322.

18. Connor BA. 2018; Multiplex PCR testing for travelers' diarrhea-friend or foe? Int J Infect Dis. 66:96–98. DOI: 10.1016/j.ijid.2017.11.022. PMID: 29175449.

19. Freeman K, Tsertsvadze A, Taylor-Phillips S, et al. 2017; Agreement between gastrointestinal panel testing and standard microbiology methods for detecting pathogens in suspected infectious gastroenteritis: test evaluation and meta-analysis in the absence of a reference standard. PLoS One. 12:e0173196. DOI: 10.1371/journal.pone.0173196. PMID: 28253337. PMCID: PMC5333893.

20. Micic D, Hirsch A, Setia N, Rubin DT. 2018; Enteric infections complicating ulcerative colitis. Intest Res. 16:489–493. DOI: 10.5217/ir.2018.16.3.489. PMID: 30090049. PMCID: PMC6077301.

21. Grass JE, Gould LH, Mahon BE. 2013; Epidemiology of foodborne disease outbreaks caused by Clostridium perfringens, United States, 1998-2010. Foodborne Pathog Dis. 10:131–136. DOI: 10.1089/fpd.2012.1316. PMID: 23379281. PMCID: PMC4595929.

22. Scallan E, Hoekstra RM, Angulo FJ, et al. 2011; Foodborne illness acquired in the United States--major pathogens. Emerg Infect Dis. 17:7–15. DOI: 10.3201/eid1701.p11101. PMID: 21192848. PMCID: PMC3375761.

23. Doyle MP, Diez-Gonzalez F, Hill C. 2019. Food microbiology: fundamentals and frontiers. 5th ed. John Wiley & Sons;Hoboken (NJ):
24. Juneja VK, Sofos JN. 2009. Pathogens and toxins in foods: challenges and interventions. ASM Press;Washington (DC):
25. Viala C, Le Monnier A, Maataoui N, Rousseau C, Collignon A, Poilane I. 2012; Comparison of commercial molecular assays for toxigenic Clostridium difficile detection in stools: BD GeneOhm Cdiff, XPert C. difficile and illumigene C. difficile. J Microbiol Methods. 90:83–85. DOI: 10.1016/j.mimet.2012.04.017. PMID: 22565213.

26. Dekker JP, Frank KM. 2015; Salmonella, Shigella, and yersinia. Clin Lab Med. 35:225–246. DOI: 10.1016/j.cll.2015.02.002. PMID: 26004640. PMCID: PMC4443274.

27. Acheson D, Allos BM. 2001; Campylobacter jejuni infections: update on emerging issues and trends. Clin Infect Dis. 32:1201–1206. DOI: 10.1086/319760. PMID: 11283810.

28. Kim H, Lee SW, Chung SP, Kim SW, Yoo IS, Ha YR. 2002; Stool white-cell count as a predictor of long-term admission in healthy patients with acute diarrhea. J Korean Soc Emerg Med. 13:381–384.
Fig. 1
Flow diagram of acute diarrheal patients who underwent stool multiplex polymerase chain reaction assays.
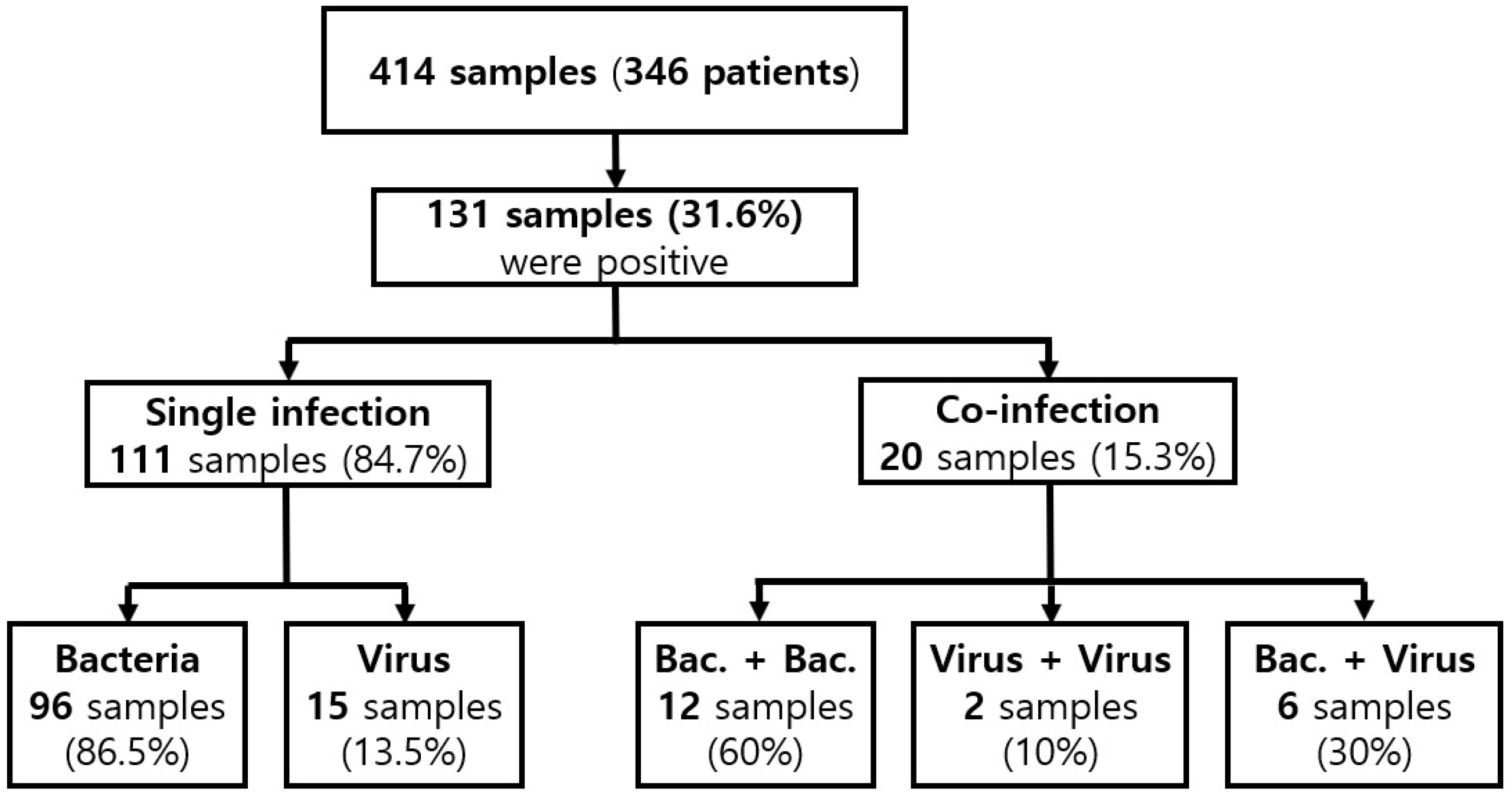
Table 1
Epidemiological Characteristics of the Enrolled Patients
| Variable | All | PCR positive | PCR negative | p-value | Adjusted p-valuea |
|---|---|---|---|---|---|
| Number | 346 | 117 | 229 | ||
| Age (years) | 63.35±21.70 | 57.41±24.35 | 66.38±19.53 | 0.001 | |
| Gender, female | 177 (51.2) | 52 (44.4) | 125 (54.6) | 0.074 | |
| Underlying disease | |||||
| HTN | 135 (39.0) | 37 (31.6) | 98 (42.8) | 0.044 | 0.506 |
| DM | 95 (27.4) | 25 (21.3) | 70 (30.6) | 0.070 | 0.376 |
| LC | 14 (4.0) | 4 (3.4) | 10 (4.4) | 0.672 | 0.821 |
| CKD | 33 (9.5) | 10 (8.5) | 23 (10.0) | 0.654 | 0.973 |
| IBD | 28 (8.1) | 9 (7.7) | 19 (8.3) | 0.845 | 0.155 |
| Symptoms | |||||
| Fever | 65 (18.7) | 24 (20.5) | 41 (17.9) | 0.483 | |
| Abdominal pain | 145 (41.9) | 59 (50.4) | 86 (37.6) | 0.011 | |
| WBC (/mm3) | 8.57±4.79 | 8.64±3.86 | 8.54±5.21 | 0.865 | |
| Hb (g/dL) | 11.82±2.37 | 12.19±2.65 | 11.63±2.19 | 0.050 | |
| BUN (mg/dL) | 20.41±15.99 | 19.52±14.68 | 20.86±16.62 | 0.464 | |
| Cr (mg/dL) | 1.18±1.50 | 1.17±1.47 | 1.18±1.52 | 0.973 | |
| CRP (mg/dL) | 5.15±6.68 | 4.99±5.96 | 5.24±7.03 | 0.750 | |
| Stool | |||||
| OB | 48/297 (16.2) | 24/93 (25.8) | 24/204 (11.8) | 0.002 | |
| WBC | 29/320 (1.1) | 20/106 (18.9) | 9/214 (4.2) | <0.001 |
Table 2
Characteristics of the Pathogenic Organism
| Pathogenic organism | Number of positive resultsa | Number of co-infection | Underlying disease | Symptoms | |||
|---|---|---|---|---|---|---|---|
|
|
|
||||||
| HTN (%) | DM (%) | CKD (%) | Fever (%) | Abdominal pain (%) | |||
| Bacteria | 127 (100.0) | ||||||
| Clostridium perfringens | 53 (41.7) | 11 | 41.5 | 28.3 | 5.7 | 11.3 | 45.3 |
| Clostridioides difficile | 30 (23.6) | 8 | 36.7 | 26.7 | 16.7 | 20.0 | 36.7 |
| Campylobacter spp. | 28 (22.0) | 5 | 7.1 | 7.1 | 0.0 | 53.6 | 75.0 |
| Aeromonas spp. | 10 (7.9) | 5 | 40.0 | 20.0 | 0.0 | 0.0 | 30.0 |
| Shigella spp. | 3 (2.4) | 0 | 0.0 | 0.0 | 0.0 | 33.3 | 33.3 |
| Salmonella spp. | 3 (2.4) | 2 | 33.3 | 0.0 | 0.0 | 0.0 | 100.0 |
| Viruses | 25 (100.0) | ||||||
| Norovirus G2 | 18 (72.0) | 6 | 44.4 | 38.9 | 33.3 | 5.6 | 61.1 |
| Group A Rotavirus | 4 (16.0) | 2 | 25.0 | 0.0 | 0.0 | 25.0 | 25.0 |
| Astrovirus | 2 (8.0) | 1 | 0.0 | 0.0 | 0.0 | 0.0 | 100.0 |
| Norovirus G1 | 1 (4.0) | 1 | 100.0 | 0.0 | 0.0 | 0.0 | 100.0 |
Table 3
Number of Stool OB, Stool WBC, and Means of WBC, CRP
| Pathogenic organism | Stool | WBCa (/mm3) | CRPa (mg/dL) | |
|---|---|---|---|---|
|
|
||||
| OBa | WBCa | |||
| Bacteria | ||||
| Clostridium perfringens | 12/45 (26.7) | 5/51 (9.8) | 8.78±5.38 | 4.8±6.7 |
| Clostridioides difficile | 1/23 (4.3) | 1/25 (4.0) | 9.23±4.58 | 3.7±5.4 |
| Campylobacter spp. | 10/24 (41.7) | 13/27 (48.2) | 8.80±2.86 | 8.4±6.3 |
| Salmonella spp. | 1/3 (33.3) | 1/3 (33.3) | 5.75±2.40 | 7.2±4.2 |
| Shigella spp. | 2/3 (66.7) | 1/3 (33.3) | 6.55±3.78 | 0.7±0.2 |
| Aeromonas spp. | 3/8 (37.5) | 1/10 (10.0) | 8.64±3.98 | 4.5±4.4 |
| Viruses | ||||
| Group A Rotavirus | 0/4 (0.0) | 1/4 (25.0) | 7.96±2.94 | 3.5±4.8 |
| Norovirus G1 | 0/1 (0.0) | 0/1 (0.0) | 6.76±0.00 | 1.8±0.0 |
| Norovirus G2 | 0/11 (0.0) | 0/15 (0.0) | 8.29±3.60 | 1.4±1.9 |
| Astrovirus | 0/1 (0.0) | 0/1 (0.0) | 5.99±1.43 | 1.6±1.9 |




 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download