1. National Research Council Committee on A Framework for Developing a New Taxonomy of Disease. 2011. Toward precision medicine: building a knowledge network for biomedical research and a new taxonomy of disease. National Academies Press;Washington, DC:
3. Connolly JL, Schnitt SJ, editors. 2003. Role of the surgical pathologist in the diagnosis and management of the cancer patient. 6th ed. Holland-Frei Cancer Medicine;Hamilton, ON:
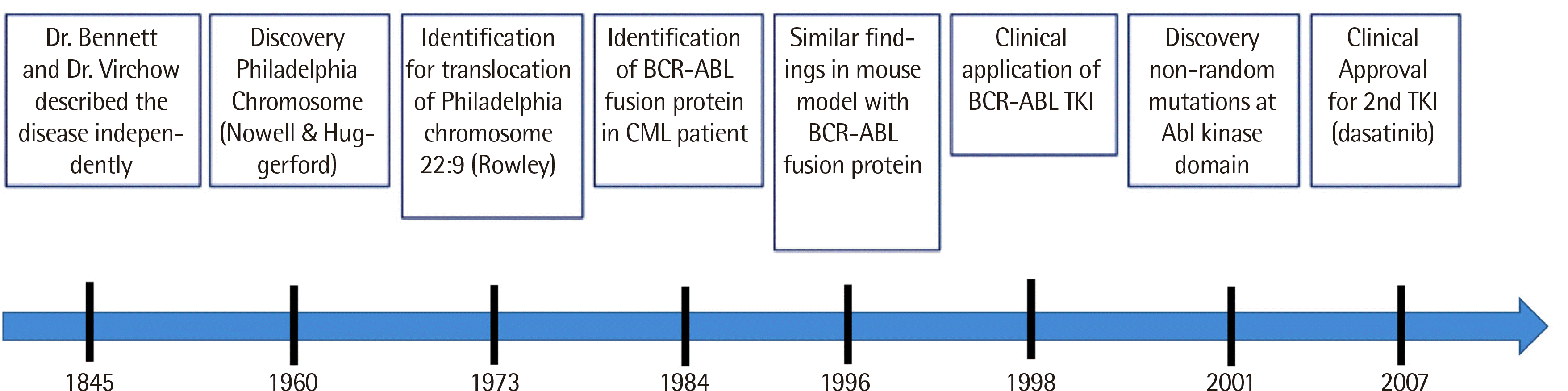
7. Hochhaus A, Larson RA, Guilhot F, Radich JP, Branford S, Hughes TP, et al. 2017; Long-term outcomes of imatinib treatment for chronic myeloid leukemia. N Engl J Med. 376:917–27. DOI:
10.1056/NEJMoa1609324. PMID:
28273028. PMCID:
PMC5901965.

8. O'Brien SG, Guilhot F, Larson RA, Gathmann I, Baccarani M, Cervantes F, et al. 2003; Imatinib compared with interferon and low-dose cytarabine for newly diagnosed chronic-phase chronic myeloid leukemia. N Engl J Med. 348:994–1004. DOI:
10.1056/NEJMoa022457. PMID:
12637609.
9. Cortes JE, Saglio G, Kantarjian HM, Baccarani M, Mayer J, Boqué C, et al. 2016; Final 5-year study results of DASISION: The dasatinib versus imatinib study in treatment-naïve chronic myeloid leukemia patients trial. J Clin Oncol. 34:2333–40. DOI:
10.1200/JCO.2015.64.8899. PMID:
27217448. PMCID:
PMC5118045.

10. Hochhaus A, Saglio G, Hughes TP, Larson RA, Kim DW, Issaragrisil S, et al. 2016; Long-term benefits and risks of frontline nilotinib vs imatinib for chronic myeloid leukemia in chronic phase: 5-year update of the randomized ENESTnd trial. Leukemia. 30:1044–54. DOI:
10.1038/leu.2016.5. PMID:
26837842. PMCID:
PMC4858585.

11. Saussele S, Hehlmann R, Fabarius A, Jeromin S, Proetel U, Rinaldetti S, et al. 2018; Defining therapy goals for major molecular remission in chronic myeloid leukemia: results of the randomized CML Study IV. Leukemia. 32:1222–8. DOI:
10.1038/s41375-018-0055-7. PMID:
29479070. PMCID:
PMC5940636.

12. Veronesi U, Saccozzi R, Del Vecchio M, Banfi A, Clemente C, De Lena M, et al. 1981; Comparing radical mastectomy with quadrantectomy, axillary dissection, and radiotherapy in patients with small cancers of the breast. N Engl J Med. 305:6–11. DOI:
10.1056/NEJM198107023050102. PMID:
7015141.

13. Veronesi U, Cascinelli N, Mariani L, Greco M, Saccozzi R, Luini A, et al. 2002; Twenty-year follow-up of a randomized study comparing breast-conserving surgery with radical mastectomy for early breast cancer. N Engl J Med. 347:1227–32. DOI:
10.1056/NEJMoa020989. PMID:
12393819.

14. Early Breast Cancer Trialists' Collaborative Group. 1998; Tamoxifen for early breast cancer: an overview of the randomised trials. Lancet. 351:1451–67. DOI:
10.1016/S0140-6736(97)11423-4. PMID:
9605801.
16. Pegram MD, Pauletti G, Slamon DJ. 1998; HER-2/neu as a predictive marker of response to breast cancer therapy. Breast Cancer Res Treat. 52:65–77. DOI:
10.1023/A:1006111117877. PMID:
10066073.

18. Malhotra GK, Zhao X, Band H, Band V. 2010; Histological, molecular and functional subtypes of breast cancers. Cancer Biol Ther. 10:955–60. DOI:
10.4161/cbt.10.10.13879. PMID:
21057215. PMCID:
PMC3047091.

19. Robson M, Im SA, Senkus E, Xu B, Domchek SM, Masuda N, et al. 2017; Olaparib for metastatic breast cancer in patients with a germline BRCA mutation. N Engl J Med. 377:523–33. DOI:
10.1056/NEJMoa1706450. PMID:
28578601.

21. Tomczak K, Czerwińska P, Wiznerowicz M. 2015; The Cancer Genome Atlas (TCGA): an immeasurable source of knowledge. Contemp Oncol (Pozn). 19:A68–77. DOI:
10.5114/wo.2014.47136. PMID:
25691825. PMCID:
PMC4322527.
23. Hoadley KA, Yau C, Wolf DM, Cherniack AD, Tamborero D, Ng S, et al. 2014; Multiplatform analysis of 12 cancer types reveals molecular classification within and across tissues of origin. Cell. 158:929–44. DOI:
10.1016/j.cell.2014.06.049. PMID:
25109877. PMCID:
PMC4152462.

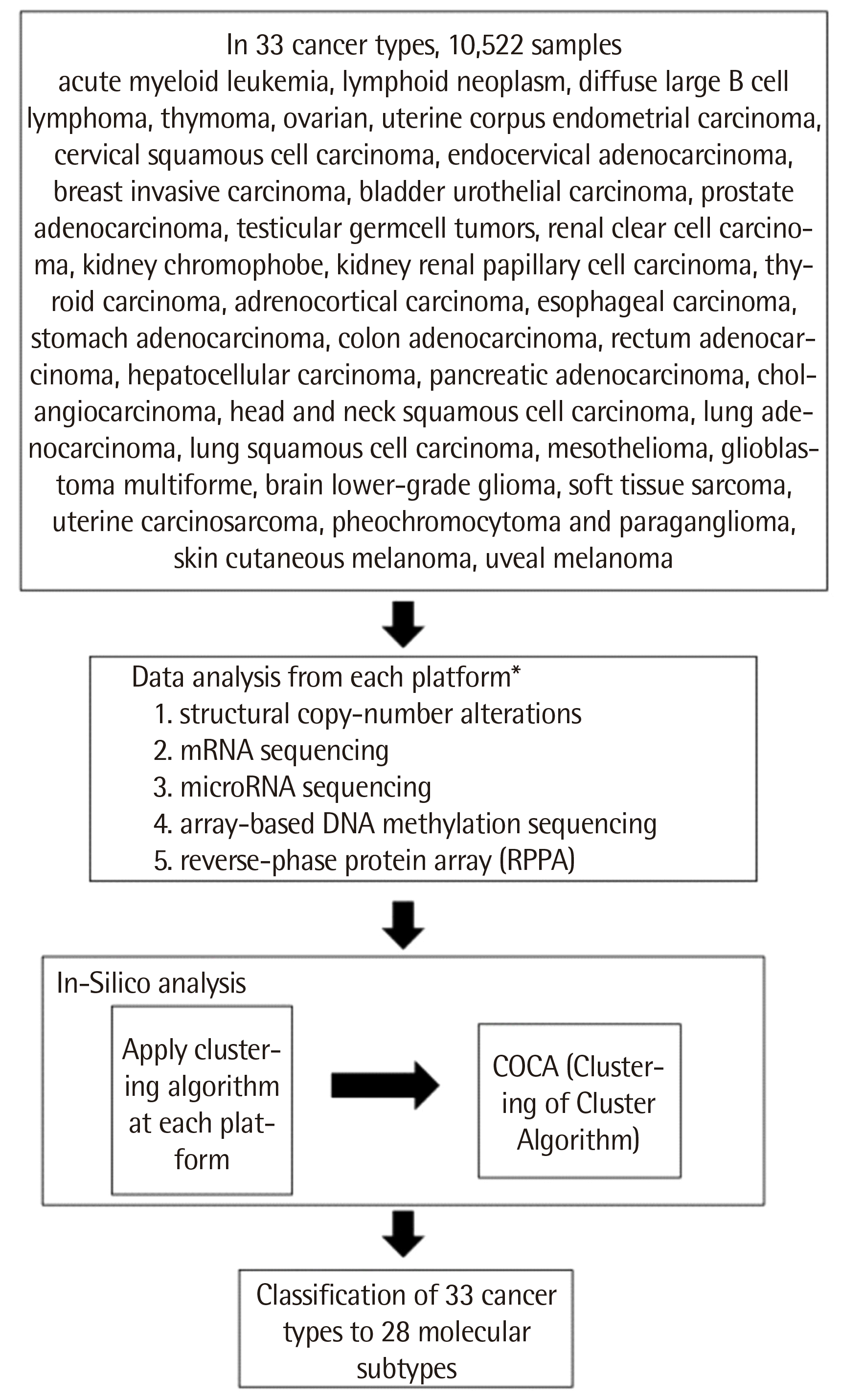
24. Hoadley KA, Yau C, Hinoue T, Wolf DM, Lazar AJ, Drill E, et al. 2018; Cell-of-origin patterns dominate the molecular classification of 10,000 tumors from 33 types of cancer. Cell. 173:291–304.e6. DOI:
10.1016/j.cell.2018.03.022. PMID:
29625048. PMCID:
PMC5957518.
25. Ricketts CJ, De Cubas AA, Fan H, Smith CC, Lang M, Reznik E, et al. 2018; The cancer genome atlas comprehensive molecular characterization of renal cell carcinoma. Cell Rep. 23:313–26.e5. DOI:
10.1016/j.celrep.2018.03.075. PMID:
29617669. PMCID:
PMC6075733.
26. Liu Y, Sethi NS, Hinoue T, Schneider BG, Cherniack AD, Sanchez-Vega F, et al. 2018; Comparative molecular analysis of gastrointestinal adenocarcinomas. Cancer Cell. 33:721–35.e8. DOI:
10.1016/j.ccell.2018.03.010. PMID:
29622466. PMCID:
PMC5966039.
27. Campbell JD, Yau C, Bowlby R, Liu Y, Brennan K, Fan H, et al. 2018; Genomic, pathway network, and immunologic features distinguishing squamous carcinomas. Cell Rep. 23:194–212.e6. DOI:
10.1016/j.celrep.2018.03.063. PMID:
29617660. PMCID:
PMC6002769.
28. Berger AC, Korkut A, Kanchi RS, Hegde AM, Lenoir W, Liu W, et al. 2018; A comprehensive pan-cancer molecular study of gynecologic and breast cancers. Cancer Cell. 33:690–705.e9. DOI:
10.1016/j.ccell.2018.03.014. PMID:
29622464. PMCID:
PMC5959730.
29. Taylor AM, Shih J, Ha G, Gao GF, Zhang X, Berger AC, et al. 2018; Genomic and functional approaches to understanding cancer aneuploidy. Cancer Cell. 33:676–89.e3. DOI:
10.1016/j.ccell.2018.03.007. PMID:
29622463. PMCID:
PMC6028190.
30. Thorsson V, Gibbs DL, Brown SD, Wolf D, Bortone DS, Ou Yang TH, et al. 2018; The immune landscape of cancer. Immunity. 48:812–30.e14.
31. Arora A, Olshen AB, Seshan VE, Shen R. 2020; Pan-cancer identification of clinically relevant genomic subtypes using outcome-weighted integrative clustering. Genome Med. 12:110. DOI:
10.1186/s13073-020-00804-8. PMID:
33272320. PMCID:
PMC7716509.

33. Sanchez-Vega F, Mina M, Armenia J, Chatila WK, Luna A, La KC, et al. 2018; oncogenic signaling pathways in the cancer genome atlas. Cell. 173:321–37.e10. DOI:
10.1016/j.cell.2018.03.035. PMID:
29625050. PMCID:
PMC6070353.
36. Sartore-Bianchi A, Pizzutilo EG, Marrapese G, Tosi F, Cerea G, Siena S. 2020; Entrectinib for the treatment of metastatic NSCLC: safety and efficacy. Expert Rev Anticancer Ther. 20:333–41. DOI:
10.1080/14737140.2020.1747439. PMID:
32223357.

37. Prahallad A, Sun C, Huang S, Di Nicolantonio F, Salazar R, Zecchin D, et al. 2012; Unresponsiveness of colon cancer to BRAF(V600E) inhibition through feedback activation of EGFR. Nature. 483:100–3. DOI:
10.1038/nature10868. PMID:
22281684.

39. Hong DS, Fakih MG, Strickler JH, Desai J, Durm GA, Shapiro GI, et al. 2020; KRASG12C inhibition with sotorasib in advanced solid tumors. N Engl J Med. 383:1207–17. DOI:
10.1056/NEJMoa1917239. PMID:
32955176. PMCID:
PMC7571518.

40. Corcoran RB, Ebi H, Turke AB, Coffee EM, Nishino M, Cogdill AP, et al. 2012; EGFR-mediated re-activation of MAPK signaling contributes to insensitivity of BRAF mutant colorectal cancers to RAF inhibition with vemurafenib. Cancer Discov. 2:227–35. DOI:
10.1158/2159-8290.CD-11-0341. PMID:
22448344. PMCID:
PMC3308191.




 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download