2. Kim KK. 2003; The role of immune response in periodontal disease. Immune Netw. 3:261–267. DOI:
10.4110/in.2003.3.4.261.

3. Statistics. 2008. Korea National Health and Nutrition Examination Survey (KNHANES). Korean MOHW.
4. Statistics. 2019. Future population estimates: 2017~2067. Korean MOHW.
5. Kassebaum NJ, Smith AG, Bernabé E, Fleming TD, Reynolds AE, Vos T, et al. 2017; GBD 2015 Oral Health Collaborators. Global, regional, and national prevalence, incidence, and disability-adjusted life years for oral conditions for 195 countries, 1990-2015: a systematic analysis for the global burden of diseases, injuries, and risk factors. J Dent Res. 96:380–387. DOI:
10.1177/0022034517693566. PMID:
28792274. PMCID:
PMC5912207.
7. Kim YT, Lee JH, Kweon HHI, Lee JS, Choi JK, Kim DW, et al. 2015; Evaluation of national health insurance coverage of periodontal scaling: a nationwide cohort study in Korea. J Korean Dent Assoc. 54:604–612.
8. Nonnenmacher C, Dalpke A, Rochon J, Flores JL, Mutters R, Heeg K. 2005; Real time polymerase chain reaction for detection and quantification of bacteria in periodontal patients. J Periodontol. 76:1542–1549. DOI:
10.1902/jop.2005.76.9.1542. PMID:
16171445.
9. Hong MH. 2014; Study on detection of oral bacteria in the saliva and risk factors of adults. J Korean Acad Industr Coop Soc. 15:5675–5682. DOI:
10.5762/KAIS.2014.15.9.5675.

10. Eick S, Straube A, Guentsch A, Pfister W, Jentsch H. 2011; Comparison of real-time polymerase chain reaction and DNA-strip technology in microbiological evaluation of periodontitis treatment. Diagn Micr Infec Dis. 69:12–20. DOI:
10.1016/j.diagmicrobio.2010.08.017. PMID:
21146709.

11. Caton JG, Armitage G, Berglundh T, Chapple IL, Jepsen S, Kornman KS, et al. 2018; A new classification scheme for periodontal and peri implant diseases and conditions-Introduction and key changes from the 1999 classification. J Periodontol. 89:S1–8. DOI:
10.1002/JPER.18-0157. PMID:
29926946.
12. Peters IR, Helps CR, Hall EJ, Day MJ. 2004; Real-time RT-PCR: considerations for efficient and sensitive assay design. J Immunol Methods. 286:203–217. DOI:
10.1016/j.jim.2004.01.003. PMID:
15087233.

13. Jung JH, Kim KY, Jeong SH, Kim KS, Lee YM. 2014; The effect of oral health behaviors on the periodontal status of teenager. J Korean Soc Dent Hyg. 14:163–171. DOI:
10.13065/jksdh.2014.14.02.163.
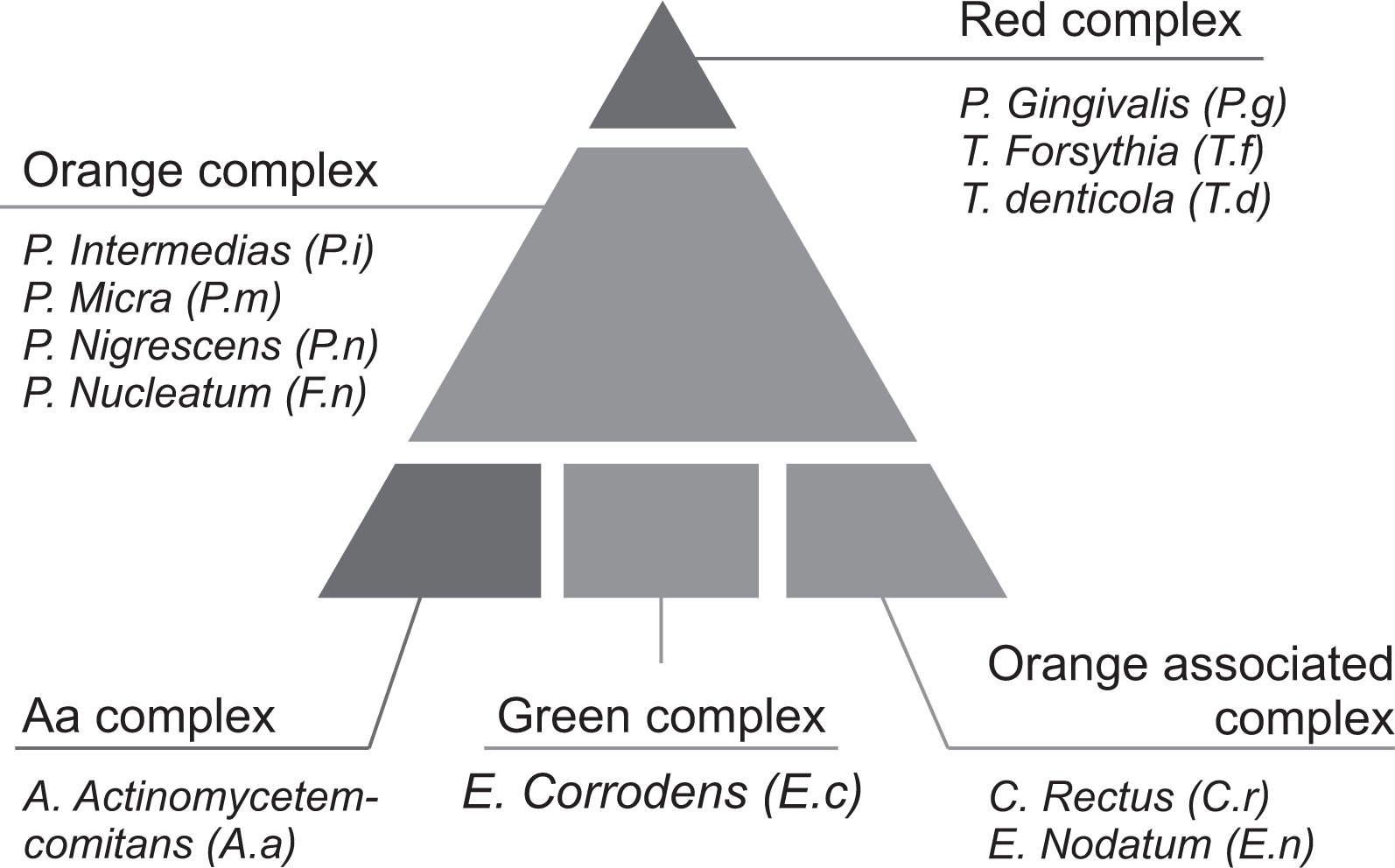
14. Socransky SS, Haffajee AD. 1992; The bacterial etiology of destructive periodontal disease: current concepts. J Periodontol. 63:322–331. DOI:
10.1902/jop.1992.63.4s.322. PMID:
1573546.

15. Choi HS. 2017; Influencing factors of dental caries across the life cycle of Koreans. J Korean Soc Dent Hyg. 10:889–898.
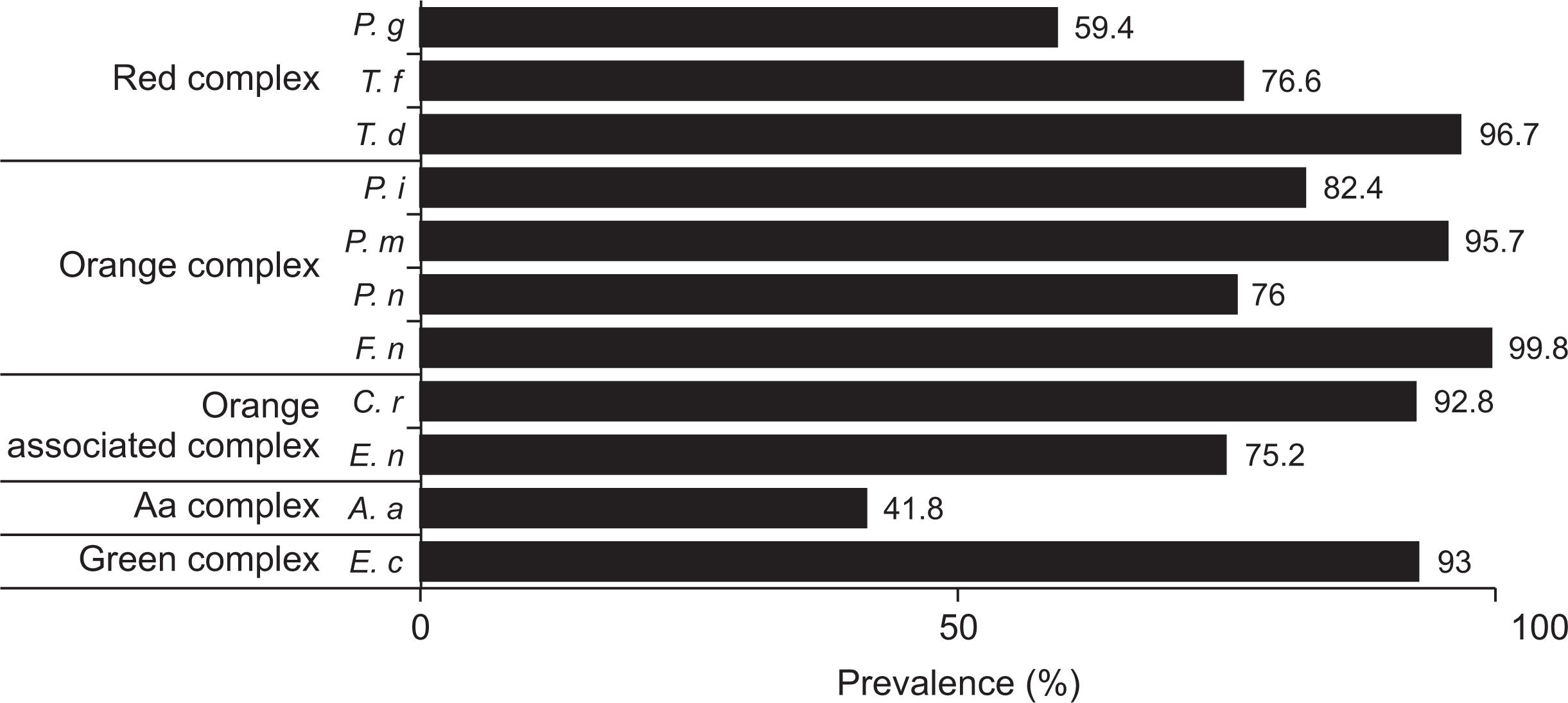
16. Yun JH, Park JE, Kim DI, Lee SI, Choi SH, Cho KS, et al. 2008; Identification of putative periodontal pathogens in Korean chronic periodontitis patients. J Korean Acad Periodontol. 38:143–152. DOI:
10.5051/jkape.2008.38.2.143.

17. Bodet C, Chandad F, Grenier D. 2007; Pathogenic potential of
Porphyromonas gingivalis,
Treponema denticola and
Tannerella forsythia, the red bacterial complex associated with periodontitis. Pathol Biol. 55:154–162. DOI:
10.1016/j.patbio.2006.07.045. PMID:
17049750.
18. Mandell RL. 1984; A longitudinal microbiological investigation of
Actinobacillus actinomycetemcomitans and
Eikenella corrodens in juvenile periodontitis. Infect Immun. 45:778–780. DOI:
10.1128/IAI.45.3.778-780.1984. PMID:
6381313. PMCID:
PMC263366.
19. Haffajee AD, Bogren A, Hasturk H, Feres M, Lopez NJ, Socransky SS. 2004; Subgingival microbiota of chronic periodontitis subjects from different geographic locations. J Clin Periodontol. 31:996–1002. DOI:
10.1111/j.1600-051X.2004.00597.x. PMID:
15491316.

20. Schenkein HA, Burmeister JA, Koertge TE, Brooks CN, Best AM, Moore LVH, et al. 1993; The influence of race and gender on periodontal microflora. J Periodontol. 64:292–296. DOI:
10.1902/jop.1993.64.4.292. PMID:
8387107.

22. Kandelman D, Arpin S, Baez RJ, Baehni PC, Petersen PE. 2012; Oral health care systems in developing and developed countries. Periodontol. 60:98–109. DOI:
10.1111/j.1600-0757.2011.00427.x. PMID:
22909109.





 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download