서 론
재료 및 방법
1. 환자와 검체
2. NGS 라이브러리(library) 제작과 염기서열 분석
3. 결과 해석
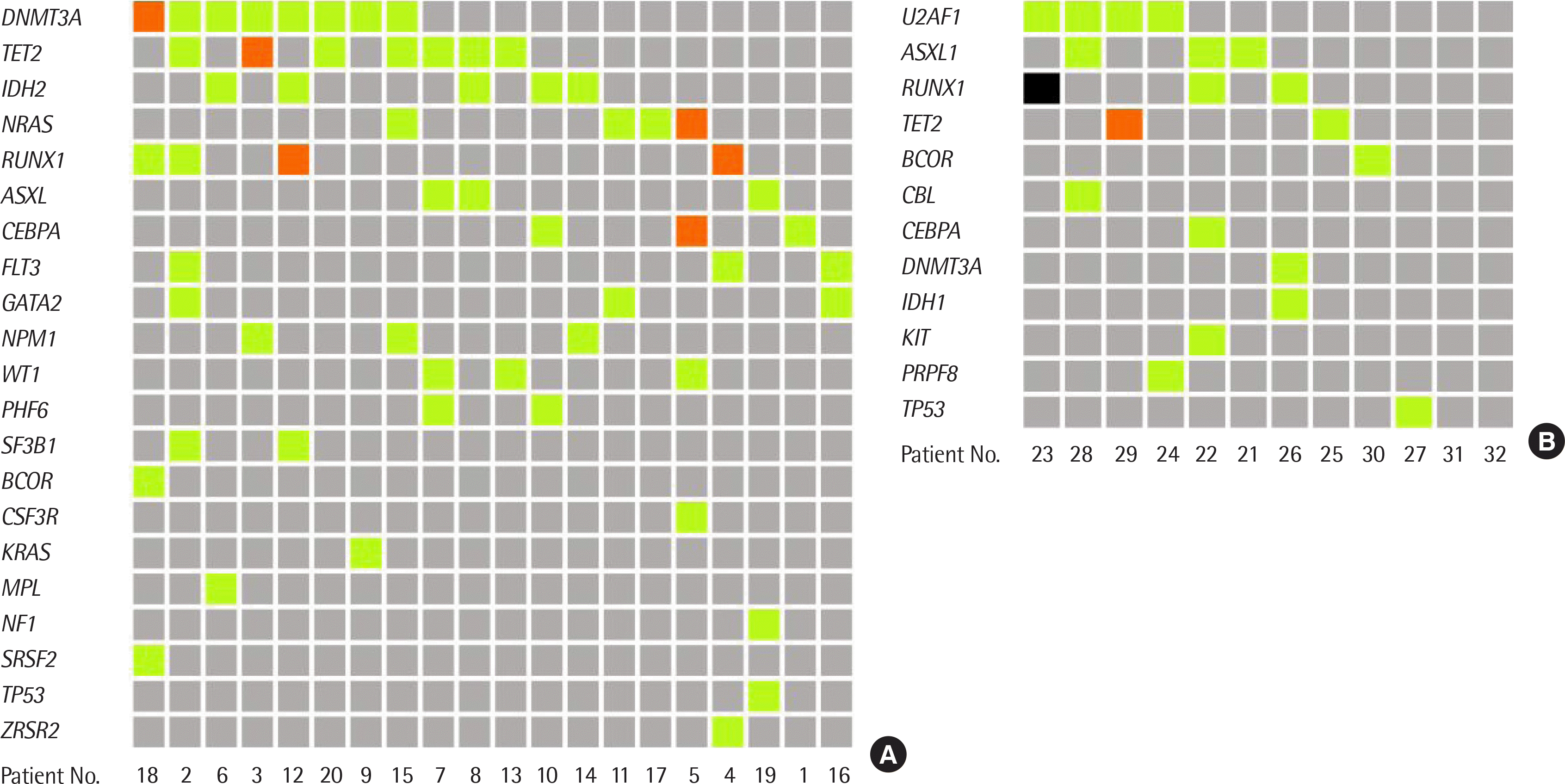
결 과
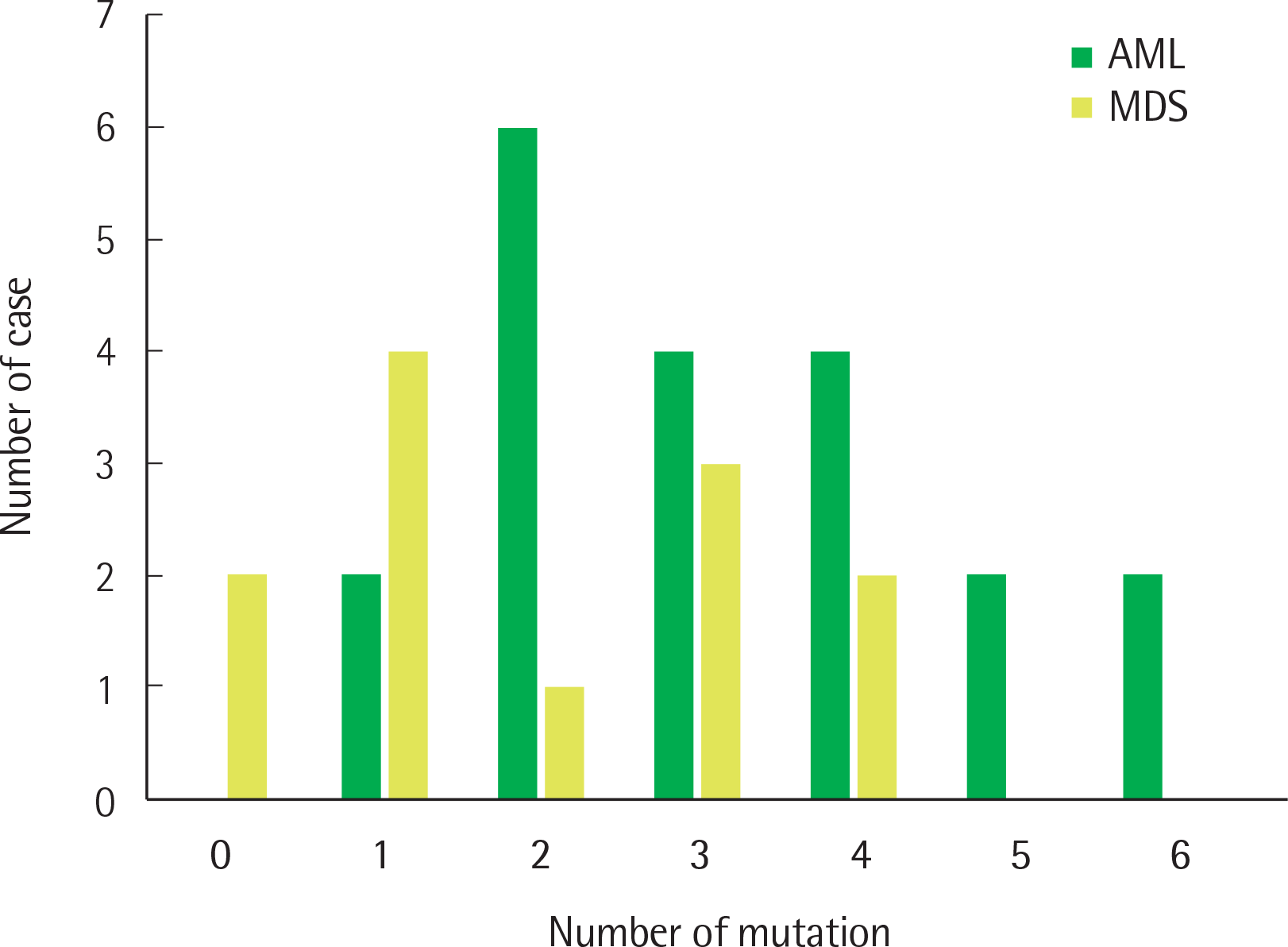 | Fig. 1Number of mutations per patient detected using the NGS myeloid panel test. Data include those for 20 patients with AML and 12 patients with MDS. |
Table 1
| No. | Sex/Age | Subtype* | Tier 1 variant† | Tier 2 variant† | Tier 3 variant† | Karyotype | Conventional genetic test results |
|---|---|---|---|---|---|---|---|
| 1 | F/77 | AMMoL | CEBPA p.E148fs | Not detected | Not detected | Normal | NPM1-, FLT3-, CEBPA+ |
| 2 | M/59 | AMMoL |
FLT3 ITD (33 bp) RUNX1 p.D198G |
DNMT3A p.D531fs |
GATA2 p.G320S TET2 p.S69R |
Normal | NPM1-, FLT3+, CEBPA- |
| 3 | F/83 | AMMoL | NPM1 p.W289fs |
DNMT3A p.R882C TET2 p.Q916* |
TET2 p.C1273F | Normal | NPM1+, FLT3-, CEBPA- |
| 4 | M/73 | AMMoL |
FLT3 ITD (24 bp) RUNX1 p.R107G RUNX1 p.G87fs |
ZRSR2 p.E118fs | Not detected | Normal | Not tested |
| 5 | F/57 | AML with biallelic mutation in CEBPA |
CEBPA p.P23fs CEBPA p.L315_ E316insT |
NRAS p.Q61H NRAS p.G13D WT1 p.R462W |
CSF3R p.T648I | +21 | NPM1-, FLT3-, CEBPA+ |
| 6 | F/66 | AML with maturation | IDH2 p.R140Q | DNMT3A p.R882C | MPL p.*636Wext*13 | add(22p) | NPM1-, FLT3-, CEBPA- |
| 7 | F/75 | AML with maturation | ASXL1 p.Q588* |
TET2 p.E1411fs WT1 p.A382fs |
PHF6 p.E152* | Normal | NPM1-, FLT3-, CEBPA- |
| 8 | F/65 | AML with maturation |
IDH2 p.R172K ASXL1 p.E635fs |
TET2 p.S1758* | Not detected | Normal | Not tested |
| 9 | M/73 | AML with maturation | DNMT3A p.R882H | KRAS p.G12D | Not detected | -7, add(8q) | NPM1-, FLT3-, CEBPA- |
| 10 | F/37 | AML with minimal differentiation |
CEBPA p.K298N IDH2 p.R172K |
Not detected | PHF6 p.L324P | t(1;2) | NPM1-, FLT3-, CEBPA+ |
| 11 | F/68 | AML-MRC | Not detected | NRAS p.G12D | GATA2 p.L359V | del(5q) | NPM1-, FLT3-, CEBPA- |
| 12 | M/79 | AML-MRC |
IDH2 p.R140Q RUNX1 p.I342fs RUNX1 p.S322* |
DNMT3A p.F827fs SF3B1 p.K700E |
Not detected | Normal | NPM1-, FLT3-, CEBPA- |
| 13 | F/51 | AML-MRC | Not detected | TET2 p.F868L | WT1 p.H465R | Normal | Not tested |
| 14 | F/79 | AML with mutated NPM1 |
IDH2 p.R140Q NPM1 p.W288fs |
Not detected | Not detected | Normal | NPM1+, FLT3-, CEBPA- |
| 15 | F/84 | AML with mutated NPM1 | NPM1 p.W288fs |
NRAS p.G13D DNMT3A p.R659P |
TET2 p.L1899H | Normal | Not tested |
| 16 | M/77 | AML with RUNX1-RUNX1T1 | FLT3 ITD (39 bp) | Not detected | GATA2 p.A372T | -Y, t(8;21) | Not tested |
| 17 | F/54 | AML with RUNX1-RUNX1T1 | Not detected | NRAS p.G13D | Not detected | t(8;21) | Not tested |
| 18 | F/77 | AML without maturation | RUNX1 p.A149E |
BCOR p.K1972fs DNMT3A p.R882H DNMT3A p.W860R SRSF2 p.P95L |
Not detected | Normal | Not tested |
| 19 | M/66 | AML without maturation | ASXL1 p.E705* | NF1 p.S2597fs | TP53 p.E271K | add(7q), add(17p) | NPM1-, FLT3-, CEBPA- |
| 20 | F/66 | Pure erythroid leukemia | Not detected |
DNMT3A p.R729W TET2 p.D1587fs |
Not detected | Complex > 3 abnormality | NPM1-, FLT3-, CEBPA- |
| 21 | F/47 | MDS with isolated del(5q) | ASXL1 p.D1000G | Not detected | Not detected | del(5q) | Not tested |
| 22 | M/62 | MDS-EB1 |
ASXL1 p.A640fs KIT p.D816V RUNX1 p.Q185fs |
Not detected | CEBPA p.Q83fs | Normal | Not tested |
| 23 | F/58 | MDS-EB1 |
RUNX1 p.S293fs RUNX1 p.P263fs |
U2AF1 p.Y158_E159dup | Not detected | +1, der(1;7)(q10;p10) | Not tested |
| 24 | M/60 | MDS-EB1 | U2AF1 p.Q157P | Not detected | PRPF8 p.I1571M | Normal | Not tested |
| 25 | F/70 | MDS-EB1 | Not detected | TET2 p.F868L | Not detected | Normal | Not tested |
| 26 | F/70 | MDS-EB2 | RUNX1 p.R107C | IDH1 p.R132C | DNMT3A p.R882H | Normal | Not tested |
| 27 | M/77 | MDS-EB2 | TP53 p.R273H | Not detected | Not detected | Complex > 3 abnormality | Not tested |
| 28 | F/74 | MDS-MLD |
ASXL1 p.Y591* CBL p.P417S U2AF1 p.Q157P |
Not detected | Not detected | Normal | Not tested |
| 29 | M/76 | MDS-MLD | U2AF1 p.Q157H | TET2 p.Q831* | TET2 p.K944del | Normal | Not tested |
| 30 | F/60 | MDS-MLD | Not detected | Not detected | BCOR p.E630K | Normal | Not tested |
| 31 | F/50 | MDS-SLD | Not detected | Not detected | Not detected | Normal | Not tested |
| 32 | M/58 | MDS-SLD | Not detected | Not detected | Not detected | Complex > 3 abnormality | Not tested |




 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download