This article has been
cited by other articles in ScienceCentral.
초록
배경
본 연구는 혈액 종양의 진단에 있어 국내의 차세대염기서열 분석(next generation sequencing, NGS) 기반 유전자 검사의 현황 을 파악하기 위하여 2017년, 2018년 2개년간 진행하였다.
방법
각 기관의 혈액 종양 관련 유전자 검사 담당 전문가에게 전 자 메일을 통한 설문을 시행하였다. 설문 항목은 각 해당 연도의 기관의 상황을 반영할 수 있도록 혈액종양 NGS 검사 현황(19 문 항)과 혈액종양 패널에 대한 기관의 의견(18 문항)에 대한 평가로 총 37 문항으로 구성되었다.
결과
총 12개(2017년도) 및 14개(2018년도)의 기관이 설문 조사에 응답하였고 그 답변을 추가로 분석하였다. 답변에 응한 대부분의 기관에서 급성 백혈병과 골수성 혈액 종양에 대해 NGS 검사를 수 행하는 반면 림프종과 다발골수종의 혈액 종양에 대해 소수의 기 관에서 검사를 수행하였다. 다수의 기관은 NGS 검사가 혈액 종양 의 초기 진단에 필수 검사 혹은 추천 검사로 시행되어야 한다는 데 동의하였다.
결론
응답한 기관마다 운용하는 NGS 패널의 구성과 플랫폼의 차 이가 있었다. 이에 NGS 유전자 검사의 검사실 간 차이를 줄이기 위 해 학회를 중심으로 한 표준화된 패널의 구성과 도출된 검사 결과 해석의 지침이 필요할 것이다.
Go to :

Abstract
Background
The aim of this study was to investigate the status of next generation sequencing (NGS)-based genetic diagnosis in hematologic malignancies in Korea in 2017 and 2018.
Methods
A structured questionnaire was provided to specialists in charge of the genetic testing of hematologic malignancies via e-mail. The questionnaire consisted of 37 questions reflecting the situation of the institutions for each year and were based on an assessment of the status of the hematologic malignancy NGS test (19 questions) and the institution’s opinion on the NGS test (18 questions).
Results
A total of 12 and 14 laboratories, in 2017 and 2018, respectively, replied to our survey and their answers were further analyzed. Most laboratories were performing NGS panel testing for acute leukemia and myeloid malignancies, and a small proportion of laboratories were testing NGS for lymphoid malignancies. The majority of participants agreed that NGS testing should be essential for the initial diagnostic workup.
Conclusions
Variation in NGS panel tests, including choice of gene and platform by different laboratories, were observed. Standardized panels and interpretation, centered around the Korean Society for Genetic Diagnostics, is needed to reduce inter-laboratory variation in NGS test results.
Go to :

Keywords: Genetic testing, Questionnaires and surveys, Hematologic malignancies, Next generation sequencing (NGS)
서 론
차세대염기서열분석(next generation sequencing, NGS) 기술은 다양한 분야에서 임상에 응용되고 있다. 최근 혈액종양의 진단에 해당 기술을 도입함으로써 질병의 시작, 진행 및 치료 반응에 대한 이해의 폭이 넓어졌으며 이는 추후 혈액종양 환자들에게 보다 정밀한 맞춤 치료의 토대가 될 것으로 기대된다[
1]. 하지만, 진단검사의학과 임상검사실에서 혈액종양에 NGS 검사법을 도입, 운영할 경우 플랫폼의 결정, 검사실 간 어떤 중요 유전자(key gene)를 포함하고 얼마나 많은 수의 유전자를 확인할 것인지, 도출된 결과를 어떻게 해석할 것인지 등의 차이가 존재하고 있어 검사 방법의 표준화 및 정도 관리의 필요성이 제기되고 있다[
2,
3]. 이에 본 연구에서는 국내 임상검사실의 혈액종양 NGS 패널의 구성, 검사 방법과 분석법에 대한 현황을 파악하고, 추후 검사 방법의 표준화를 이루기 위한 기초자료로 사용하고자 해당 검사를 시행하는 기관을 대상으로 혈액종양 NGS 검사 방법에 대해 설문 조사를 시행하고 응답자의 답변을 평가하였다.
Go to :

재료 및 방법
본 연구는 2017, 2018년도 2년 동안 실시하였다. 연구대상자는 각 해당 연도 기준 NGS 검사 시행 53개의 기관에 설문을 보내고 참여 의사를 확인한 후 답변에 응한 각각 12, 14개의 의료기관을 대상으로 선정하여 답변을 분석하였다. 설문은 각 기관은 유전자 검사 담당 전문가에게 이메일로 전달하여 2017년 10월 17일부터 11월 6일까지, 2018년 8월 28일부터 9월 14일까지 총 2차례 온라인 설문조사를 실시하고 답변을 받았다.
설문 항목은 혈액종양 NGS 검사 현황(19 문항)과 혈액종양 패널에 대한 기관의 의견(18 문항)에 대한 평가로 총 37 문항으로 이루어졌다.
각 문항별 설문 응답 빈도를 분석하였고, 중복 및 주관식 질문의 경우 별도로 정리하였다.
Go to :

결 과
설문에 응답한 기관은 대학병원이 9기관(75.0%, 64.3%)으로 가장 많았다. 선택형 설문 응답 결과는
Table 1,
Fig. 1과 같다. 문항 1에서 확인한 2017년도에 혈액종양 NGS 검사를 시행 또는 시행 예정인 기관은 12개 기관 중 10기관(83.4%) 이었으며 2018년도에는 14개 기관 중 12개 기관(85.7%)이었다. 해당 연도에 각 기관에서 시행 혹은 시행 예정인 혈액종양 패널의 종류는
Fig. 1과 같다. 급성골수성백혈병(acute myeloid leukemia, AML), 골수형성이상증후군(myelodysplastic syndrome, MDS), 골수증식종양(myeloproliferative neoplasm, MPN) 및 급성림프모구백혈병(acute lymphoblastic leukemia, ALL) 패널이 가장 많은 기관에서 시행하고 있거나 시행 예정이었으며 2017년도에 비해 2018년도에 시행 기관의 수가 각 종양별로 모두 증가하였다.
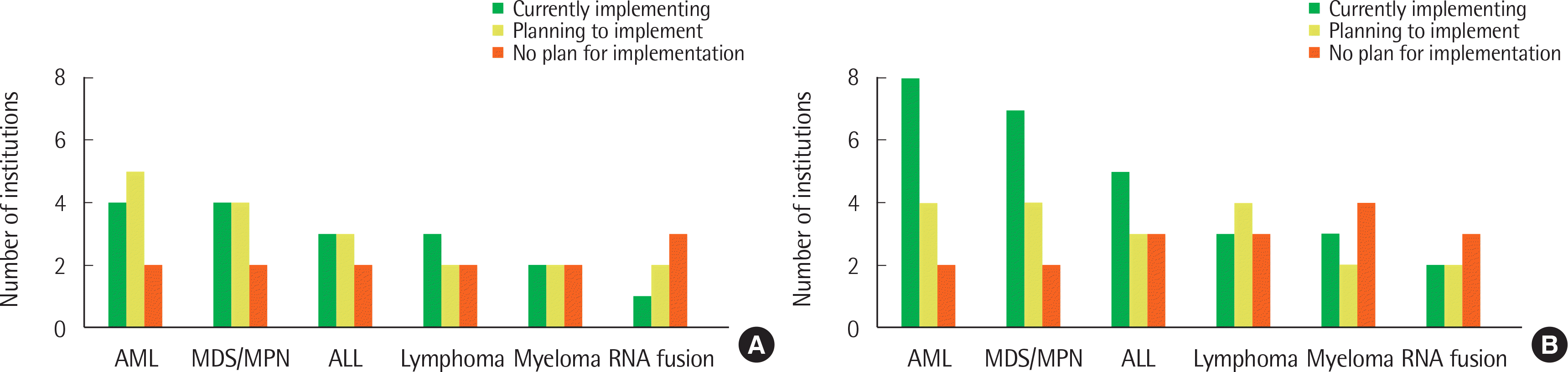 | Fig. 1
The number of institutions implementing each of the hematologic malignancy NGS panels. Survey results for (A) 2017 and (B) 2018. The green bar indicates the number of institutions that are currently implementing NGS for diagnosis of hematologic malignancies. The yellow bar indicates the number of institutions that are planning to implement NGS for diagnosis of hematologic malignancies. The red bar indicates the number of institutions that have no plan for setting up an NGS panel for diagnosis.
Abbreviations: AML, acute myeloid leukemia; MDS/MPN, myelodysplastic syndrome/myeloproliferative neoplasm; ALL, acute lymphoblastic leukemia.

|
Table 1
Survey questions and answer frequencies
|
Questions/answer Options |
Answer (2017) N (%) |
Answer (2018) N (%) |
|
1. Does your institution currently have an NGS panel for hematologic malignancies? |
|
|
|
A. Yes |
5 (41.7) |
8 (57.1) |
|
B. Planning to set up the NGS panel |
5 (41.7) |
4 (28.6) |
|
C. No |
2 (16.7) |
2 (14.3) |
|
2. Does your institution use (or is planning to use) a comprehensive panel for hematologic malignancies? If so, please write down the panel your institution is using. |
|
|
|
A. Yes |
6 (50.0) |
9 (64.3) |
|
Myeloid panel (AML+MDS+MPN) |
4 (33.3) |
8 (57.1) |
|
Myeloid panel (AML+MDS+MPN) with Mature lymphoid panel (plasma cell myeloma+lymphoma) |
1 (8.3) |
1 (7.1) |
|
Pan hematology panel |
1 (8.3) |
0 (0.0) |
|
B. No |
6 (50.0) |
5 (35.8) |
|
3. Do you think NGS panel for hematologic malignancies can replace Sanger sequencing? |
|
|
|
A. Yes, either partially or entirely |
8 (66.7) |
6 (42.9) |
|
B. Yes, but only partially |
4 (33.3) |
8 (57.1) |
|
C. No, it is irreplaceable |
0 (0.0) |
0 (0.0) |
|
4. Do you think NGS panel for hematologic malignancies can replace real-time polymerase chain reaction (RT-PCR)? |
|
|
|
A. Yes, either partially or entirely |
4 (33.3) |
6 (42.9) |
|
B. Yes, but only partially |
8 (66.7) |
8 (57.1) |
|
C. No, it is irreplaceable |
0 (0.0) |
0 (0.0) |
|
5. Do you think NGS panel for hematologic malignancies can replace fluorescence in situ hybridization (FISH)? |
|
|
|
A. Yes, either partially or entirely |
3 (25.0) |
1 (7.1) |
|
B. Yes, but only partially |
7 (56.3) |
10 (71.4) |
|
C. No, it is irreplaceable |
2 (16.7) |
3 (21.4) |
|
6. What do you think is the appropriate average turnaround time (TAT) for NGS test? |
|
|
|
A. 7 days |
0 (0.0) |
0 (0.0) |
|
B. 15 days |
5 (41.7) |
5 (35.7) |
|
C. 30 days |
6 (50.0) |
9 (64.3) |
|
D. 60 days |
1 (8.3) |
0 (0.0) |
|
7. Do you think it is necessary to perform genetic test for germline predisposing mutations? |
|
|
|
A. It should be performed for all patients |
3 (25.0) |
3 (21.4) |
|
B. It should be performed for patients with certain condition, e.g. family history |
9 (75.0) |
11 (78.6) |
|
C. There is no need to perform the test |
0 (0.0) |
0 (0.0) |
|
8. What did you find difficult with respect to performing or preparing the NGS panel for hematologic malignancies (multiple answers possible)? |
|
|
|
A. Costs associated with set-up and implementation |
8 (66.7) |
6 (46.2) |
|
B. Limitation in the number of genes that can be included in the panel |
0 (0.0) |
2 (15.4) |
|
C. Validation of a gene panel |
5 (41.7) |
11 (84.6) |
|
D. Personnel training time |
2 (16.7) |
5 (38.5) |
|
E. Efforts to meet the turnaround time (TAT) |
2 (16.7) |
10(76.9) |
|
F. Bioinformatics analysis (human resource, program, etc.) |
3 (25.0) |
6 (46.2) |
|
G. Result interpretation and reporting |
3 (25.0) |
8 (61.5) |
|
H. Communication with physicians |
2 (16.7) |
4 (30.8) |
|
9. What do you think about creating a consensus panel for hematologic malignancies by Korean Society for Genetic Diagnostics (KSGD)? |
|
|
|
A. Need for a consensus panel and willing to use it when the panel is made |
6 (50.0) |
5 (35.7) |
|
B. Each institution uses its own panel independently but shares the crucial genes for the diagnosis of malignancies |
6 (50.0) |
9 (64.3) |
|
C. No need for a consensus panel |
1 (8.3) |
0 (0.0) |
|
10. What do you think is the right application of an NGS panel at initial diagnosis of AML? |
|
|
|
A. Mandatory test |
4 (33.3) |
7 (50.0) |
|
B. Recommended test |
8 (66.7) |
7 (50.0) |
|
C. Research based test |
0 (0.0) |
0 (0.0) |
|
11. What do you think is the right application of an NGS panel at initial diagnosis of MDS/MPN? |
|
|
|
A. Mandatory test |
4 (33.3) |
9 (64.3) |
|
B. Recommended test |
8 (66.7) |
5 (35.7) |
|
C. Research based test |
0 (0.0) |
0 (0.0) |
|
12. What do you think is the right application of an NGS panel at initial diagnosis of ALL? |
|
|
|
A. Mandatory test |
4 (33.3) |
3 (21.4) |
|
B. Recommended test |
8 (66.7) |
11 (78.6) |
|
C. Research based test |
0 (0.0) |
0 (0.0) |
|
13. What do you think is the right application of an NGS panel at initial diagnosis of plasma cell myeloma? |
|
|
|
A. Mandatory test |
1 (8.3) |
1 (8.3) |
|
B. Recommended test |
8 (66.7) |
10 (76.9) |
|
C. Research based test |
3 (25.0) |
2 (15.4) |
|
Not answered |
0 (0.0) |
1 (8.3) |
|
14. What do you think is the right application of an NGS panel at initial diagnosis of lymphoma? |
|
|
|
A. Mandatory test |
3 (25.0) |
3 (21.4) |
|
B. Recommended test |
9 (75.0) |
10 (76.9) |
|
C. Research based test |
0 (0.0) |
1 (8.3) |
|
Not answered |
0 (0.0) |
1 (8.3) |

혈액종양 NGS 검사가 기존 검사를 대체 가능할 지 여부를 묻는 질문에 대해 답변을 한 모든 기관에서 Sanger 시퀀싱 및 실시간중합효소연쇄반응(real-time polymerase chain reaction, RT-PCR) 검사 방법을 완전히 혹은 일부 대체 가능하다고 대답하였으며 형광제자리부합법(˜uorescence in situ hybridization, FISH) 검사 방법은 16.7% (2/12), 21.4% (3/14)의 기관에서 대체가 불가능하다고 답변하였고, 56.3% (7/12), 71.4% (10/14)에서는 부분적으로 대체 가능하다고 답변하였다.
NGS 검사의 적절한 소요시간(turnaround time, TAT)은 30일 이내라고 답변한 기관이 50.0% (6/12), 64.3% (9/14)로 가장 많았으며 생식세포 소인(germ-line predisposition) 관련 유전자 검사는 75.0% (9/12) 및 78.6% (11/14)의 기관에서 가족력과 같은 환자의 조건에 따라 의심이 될 경우에 시행하여야 한다고 답변하였다.
2017년도 및 2018년도에 설문에 응답한 기관들은 혈액종양 NGS 패널을 시행 및 준비하면서 높은 초기 준비 비용(66.7%, 46.2%) 및 패널의 초기 검증 작업(41.7%, 84.6%)을 어려운 부분으로 생각하였으며 2018년도에는 그 외에 TAT를 준수하는 문제(76.9%), NGS 결과의 해석 및 보고서 작성(61.5%), 생물정보(bioinformatics) 분석(46.2%) 등을 추가로 답변하였다. 학회 차원에서 혈액종양의 합의(consensus) 패널을 제작하는 것에 관하여 2018년도 답변한 기관들의 의견에 따르면 필수 유전자를 공유하는 것은 모두 동의하였으나 다수의 기관(64.3%)에서 독자적인 패널의 운용을 선호한다고 답변하였다.
2년간 답변한 모든 기관에서 AML, MDS, MPN 및 ALL의 초진단에 NGS 검사가 필수 검사 혹은 추천 검사로 필요하다고 답변하였으며 다발골수종의 초진단에는 75.0%, 85.2%의 기관에서만 필수 혹은 추천 검사로 필요하다고 답변하였다. 주관식 질문에 관한 결과는 다음과 같다. 2018년도 기준, 각 기관의 혈액종양 NGS 패널에 포함된 혹은 포함 예정인 유전자의 분포는
Fig. 2와 같다. 해당 질문에 대해서는 현재 각 패널을 시행 및 추후 시행예정으로 구체적인 패널 구성이 완료된 기관에서 답변하였다. 각 패널별 포함하는 유전자의 수는 보건복지부 고시 필수 유전자(각 종양별 3-11개)만 포함하는 경우부터 200개 이상, 많은 경우 500개 이상으로 각 기관별로 다양하였다. 고시된 각 패널별 필수 유전자 이외 초진단에서 꼭 추가해야 할 것으로 생각되는 유전자로 설문에 대답한 기관에서 제시한 유전자는
Table 2와 같다. 2018년도 기준, 각 종양 별 맞춤제작(custom-designed) NGS 패널 사용 현황은 AML (8/11, 72.7%), MDS 또는 MPN (7/9, 77.8%), ALL (6/6, 100.0%), 다발골수종(3/3, 100.0%) 및 림프종(4/4, 100.0%)으로 파악되었다.
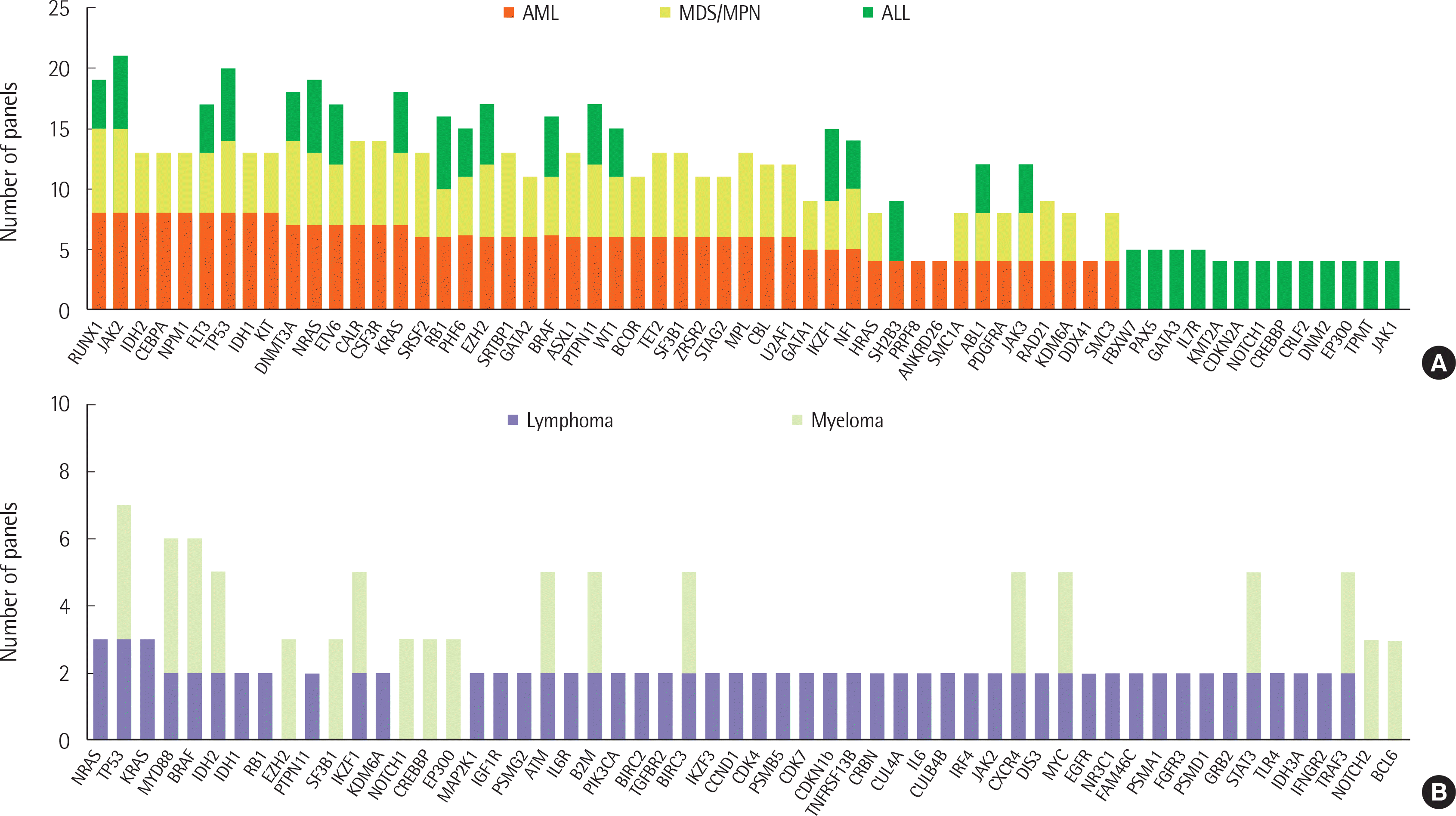 | Fig. 2The genes included in each hematologic malignancy NGS panel, based on 2018 findings. Distribution of genes across the panels for (A) acute myeloid leukemia (AML), myelodysplastic syndrome/myeloproliferative neoplasm (MDS/MPN), acute lymphoblastic leukemia (ALL), and (B) for lymphoma and plasma cell myeloma (Myeloma). 
|
Table 2
The core genes suggested by the Ministry of Health and Welfare, Korea and the genes that respondent institutions answered should be included in each panel
|
Panel |
Core genes |
Candidates for additional core genes |
|
AML |
RUNX1, JAK2, IDH2, CEBPA, NPM1, FLT3, TP53, IDH1, KIT
|
DNMT3A, NRAS, ETV6, KRAS, GATA2, ASXL1, WT1, TET2, ANKRD26
|
|
MDS/MPN |
SETBP1, MPL, ASXL1, SRSF2, RUNX1, CALR, SF3B1, CSF3R, TET2, DNMT3A, JAK2
|
NRAS, TP53, U2AF1, EZH2, GATA2, IDH2, STAG2, BCOR, IDH1, ZRSR2, CBL, LNK
|
|
ALL |
NRAS, JAK2, RB1, TP53, IKZF1
|
PAX5, ETV6, CDKN2A, NOTCH1, CREBBP, FBXW7, PTEN, PTPN11,
|
|
Lymphoma |
NRAS, TP53, KRAS
|
BRAF, SF3B1, NOTCH1, NOTCH2, MAP2K1, CD79B, ARID1B, KMT2D, ALK, ATM, BCL2, BIRC3, TCF3, ATM,
|
|
Myeloma |
MYD88, TP53, BRAF
|
NOTCH2, SF2B1, DIS3, KMT2A, NRAS, KRAS, TP53
|

혈액 종양 NGS에 관한 기관별 기타 추가 의견으로는 변이 해석에 대해 기관별 차이가 발생할 수 있어 한국인의 합의 데이터베이스를 구축하고 주기적으로 데이터베이스를 갱신하여 공유하면 좋을 것이라는 의견과 검사의 해석 및 결과 보고에 있어 국내의 표준화된 지침이 필요하다는 의견이 있었다. 또한 검사 시행 및 분석에 필요한 인력 확보 방안에 대해서도 논의를 하면 좋겠다는 의견이 있었다.
2년 연속 진행한 동일 설문에 응답한 기관은 총 7개 기관이었으며 모두 혈액종양 NGS 검사 시행 및 시행 예정 기관이었다. 설문 당시 2개의 기관에서 2017년에 혈액종양 NGS 검사가 시행 예정이었으나 2018년에는 검사 시행 중으로 현황의 변경이 있어 검사를 진행하는 기관은 3곳에서 5곳으로 증가하였다. 7개의 기관에서 혈액 종양 NGS 검사의 기존 검사들의 대체 가능 여부 문항에 대한 답변의 변화가 있었다. NGS 검사의 Sanger 시퀀싱의 대체 가능 여부는 2개의 기관에서 완전히 혹은 일부 대체 가능하다는 의견에서 일부 대체 가능하다는 의견으로 변경되었으며, FISH 검사의 대체 가능 여부는 1개의 기관에서 완전히 혹은 일부 대체 가능하다는 의견에서 일부 대체 가능하다는 의견으로 변경되었다. RNA NGS 검사의 기존 RT-PCR 검사 대체 가능 여부에 관한 문항에도 1개의 기관에서 완전히 혹은 일부 대체 가능하다는 의견에서 일부 대체 가능하다는 의견으로 변경되었다. 그 이외의 문항에 대해서는 답변의 큰 차이는 없었다.
Go to :

고 찰
2017년과 2018년 혈액종양 NGS 검사 시행 기관의 현재 상황을 본 설문을 통해 확인하였다. AML과 같은 혈액종양에서 획득된 체세포 돌연변이는 병인에 필수적인 역할을 함이 알려져 현재 질환의 진단 및 예후 표지자로 잘 확립되어 있다[
4]. 기존에 발표된 연구에 따르면 AML 환자에서 평균 13개의 유전자 변이가 관찰되며 이 중 5개는 백혈병 환자에서 흔하게 발견되는 변이였다[
5]. MDS 및 MPN 환자군에서도 78%의 환자에서 1개 이상의 종양유발 유전자의 변이가 보고되었으며 그 변이의 수가 많을수록 불량한 예후와 연관되어 있다는 보고가 있다[
6]. 이에 혈액종양 환자에 있어 많은 수의 표적 유전자를 선별하는 것이 필요하며 이는 기존의 단일 유전자 변이 분석 방법을 통해서는 이와 같은 다양한 유전자 변이의 확인에 어려움이 있다. 이에 혈액종양 환자의 진료에 NGS의 도입과 적용은 질환의 진단, 예후 예측 및 치료 방침 설정에 도움이 될 것이라 예상된다[
7]. 이는 본 설문에 답변한 대부분의 기관에서도 유사하게 혈액종양의 진단에 NGS 패널이 필수적 혹은 선택적으로 필요하다고 답변한 것과 일치한다. 또한 이러한 임상적 필요성을 반영하여 2017년도와 비교하여 2018년도에 NGS를 시행하는 기관의 수가 증가하였으며 각 기관이 운용하는 패널의 수가 증가하고 있음을 본 연구에서 확인할 수 있었다. 하지만 이러한 임상적 필요성에도 불구하고 초기 준비 비용, 각 혈액 종양과 연관된 유전자의 선정, 검사 시행 및 분석에 필요한 인력, 까다로운 검증 절차, 합리적인 TAT 유지 등의 어려움을 이유로 다수의 기관에서 혈액종양 NGS 검사의 도입이 쉽지 않음을 예상할 수 있었다.
혈액종양의 진단에 NGS 패널 적용이 확대되었음에도 불구하고 NGS 검사가 기존의 Sanger 시퀀싱 검사 방법을 대체 가능한가에 혈액종양의 진단에 NGS 패널 적용이 확대되었음에도 불구하고 NGS 검사가 기존의 Sanger 시퀀싱 검사 방법을 대체 가능한가에 대한 질문에는 2017년도 설문에서는 완전 혹은 일부 대체 가능하다는 답변이 과반 이상을 차지하였으나(66.7%) 2018년도 설문에는 일부 대체 가능하다는 답변이 과반을 차지하였다(57.1%). 흥미롭게도 2년 연속 응답한 2개의 기관에서도(NGS 검사 시행 중인 1개의 기관, 시행 예정인 1개의 기관) NGS 검사가 Sanger 시퀀싱 검사를 2017년도에서는 완전 혹은 일부 대체 가능하다고 의견을 주었으나 2018년도에는 일부 대체 가능하다는 의견으로 변경되었다. 이는 실제 패널 운용 시, 큰 크기의 염기의 삽입 혹은 결실이 있거나 GC 함량이 높은 CEBPA, FLT3 및 CALR 유전자와 같이 진단과 치료에 중요하나 NGS로 진단하기에 어려운 유전자들을 그 이유로 생각하여 볼 수 있을 것이다[8, 9].
본 조사 결과, NGS 검사를 시행하는 각 기관별 혈액종양 패널 운용의 상당한 차이가 확인되었다. 첫째, 각 기관별 시행 중 혹은 시행 예정인 패널의 차이가 존재하였으며 AML, MDS, MPN 패널의 운용은 활발한 반면 다발골수종 및 RNA 융합유전자 패널의 시행은 상대적으로 낮았다(
Fig. 1). 이는 다발골수종의 경우 유전자 변이가 질병의 발생 보다 진행에 관련되어 있다고 알려져 있고, 아직 많은 관련 유전 변이가 보고되지 않았기 때문으로 해석된다[
10-
12]. 또한 RNA 융합유전자 패널의 경우 기존의 다중(multiplex) PCR이 여전히 널리 사용되고 있기 때문에 상대적으로 TAT가 지연될 수 밖에 없는 NGS 패널의 운영이 저조한 것으로 파악된다[
13]. 이러한 이유로 각 기관마다 해당 종양의 NGS 패널 사용 여부의 차이가 있었음을 예상할 수 있었다. 둘째, 각 종양의 NGS 패널의 구성 유전자 및 그 수가 기관마다 차이가 있었다. 현재 각 기관에서 진행하는 패널에는 보건복지부 고시 필수 유전자는 모두 포함되어 있었으나 NGS의 대량 병렬 시퀀싱의 특성을 고려하였을 때 각 기관 역량 및 임상의 요청에 따라 추가된 검사 유전자가 다양하였다. 많은 기관들에서 현재 고시된 필수 유전자 이외에 AML의
ASXL1, DNMT3A, TET2 등과 같은 구동 돌연변이(driver mutation) 유전자[
14] 및
ANKRD26, GATA2, PAX5, ETV6, DDX41과 같은 생식세포 소인 유전자[
15] 등을 패널에 추가해야 할 것으로 생각된다는 의견이 있어(
Table 2), 추후 학회에서는 이러한 의견을 종합하여 패널 별 포함 유전자의 합의를 이루도록 하는 것이 필요하겠다.
임상 검사에서는 작은 오류가 환자 진단이나 치료에 영향을 줄 수 있으며 혈액종양 환자의 진료에 있어서 NGS 검사의 중요성을 감안한다면 검사실에서는 검출된 변이에 대해 정확하고 일관된 해석을 제공해야 한다. 해석에 참고한 데이터베이스나 분석을 시행한 시점 등에 따라 변이의 해석이 기관별로 다를 수 있기 때문에[
16] 학회에서 변이 해석에 적용하는 데이터베이스를 구축하고 주기적으로 이를 업데이트하여 그 결과를 공유한다면 정확하고 일관된 보고에 큰 도움이 될 것으로 판단된다.
이번 설문조사를 통하여 기관 간 혈액종양 NGS의 현황을 파악하고 그 차이를 확인하였다. 최근 식품의약품안전처에서 체세포 변이의 NGS 유전정보분석 세부 지침을 발표하였으며[
17], 혈액종양 NGS 패널의 검증 지침[
18]이 새롭게 제정되어 검사 표준화를 위한 노력이 진행되고 있으나, 혈액종양 NGS 패널에 통용되는 표준화된 보고 양식을 포함한 지침은 현재 존재하지 않는다. 이에 학회 차원의 표준화 지침 제정과 혈액종양 NGS 검사 시행기관의 정기적인 현황 조사 및 의견 취합이 필요할 것으로 생각된다.
Go to :

감사의 글
본 연구는 진단혈액학회 표준화위원회의 지원으로 이루어졌습니다.
Go to :

Notes
Go to :

REFERENCES
1. Merker JD, Valouev A, Gotlib J. 2012; Next-generation sequencing in hematologic malignancies: what will be the dividends? Ther Adv Hematol. 3:333–9. DOI:
10.1177/2040620712458948. PMID:
23606936. PMCID:
PMC3627325.


2. Endrullat C, Glökler J, Franke P, Frohme M. 2016; Standardization and quality management in next-generation sequencing. Appl Transl Genom. 10:2–9. DOI:
10.1016/j.atg.2016.06.001. PMID:
27668169. PMCID:
PMC5025460.


3. Kim J, Park WY, Kim NKD, Jang SJ, Chun SM, Sung CO, et al. 2017; Good laboratory standards for clinical next-generation sequencing cancer panel tests. J Pathol Transl Med. 51:191–204. DOI:
10.4132/jptm.2017.03.14. PMID:
28535585. PMCID:
PMC5445206.


5. Ley TJ, Miller C, Ding L, Raphael BJ, Mungall AJ, Robertson A, et al. 2013; Genomic and epigenomic landscapes of adult de novo acute myeloid leukemia. N Engl J Med. 368:2059–74. DOI:
10.1056/NEJMoa1301689. PMID:
23634996. PMCID:
PMC3767041.


6. Papaemmanuil E, Gerstung M, Malcovati L, Tauro S, Gundem G, Van Loo P, et al. 2013; Clinical and biological implications of driver mutations in myelodysplastic syndromes. Blood. 122:3616–27. DOI:
10.1182/blood-2013-08-518886. PMID:
24030381. PMCID:
PMC3837510.


7. Bacher U, Shumilov E, Flach J, Porret N, Joncourt R, Wiedemann G, et al. 2018; Challenges in the introduction of next-generation sequencing (NGS) for diagnostics of myeloid malignancies into clinical routine use. Blood Cancer J. 8:113. DOI:
10.1038/s41408-018-0148-6. PMID:
30420667. PMCID:
PMC6232163.


8. Aguilera-Diaz A, Vazquez I, Ariceta B, Mañú A, Blasco-Iturri Z, Palomino-Echeverría S, et al. 2020; Assessment of the clinical utility of four NGS panels in myeloid malignancies. Suggestions for NGS panel choice or design. PLoS One. 15:e0227986. DOI:
10.1371/journal.pone.0227986. PMID:
31978184. PMCID:
PMC6980571.
9. Thomas M, Sukhai MA, Zhang T, Dolatshahi R, Harbi D, Garg S, et al. 2017; Integration of technical, bioinformatic, and variant assessment appro-aches in the validation of a targeted next-generation sequencing panel for myeloid malignancies. Arch Pathol Lab Med. 141:759–75. DOI:
10.5858/arpa.2016-0547-RA. PMID:
28557600.

10. Morgan GJ, Walker BA, Davies FE. 2012; The genetic architecture of multiple myeloma. Nat Rev Cancer. 12:335–48. DOI:
10.1038/nrc3257. PMID:
22495321.

11. Palumbo A, Avet-Loiseau H, Oliva S, Lokhorst HM, Goldschmidt H, Rosinol L, et al. 2015; Revised International Staging System for Multiple Myeloma: A report from International Myeloma Working Group. J Clin Oncol. 33:2863–9. DOI:
10.1200/JCO.2015.61.2267. PMID:
26240224. PMCID:
PMC4846284.


12. Walker BA, Wardell CP, Melchor L, Brioli A, Johnson DC, Kaiser MF, et al. 2014; Intraclonal heterogeneity is a critical early event in the development of myeloma and precedes the development of clinical symptoms. Leukemia. 28:384–90. DOI:
10.1038/leu.2013.199. PMID:
23817176. PMCID:
PMC3916874.

13. Kim B, Lee H, Shin S, Lee ST, Choi JR. 2019; Clinical evaluation of massively parallel RNA sequencing for detecting recurrent gene fusions in hematologic malignancies. J Mol Diagn. 21:163–70. DOI:
10.1016/j.jmoldx.2018.09.002. PMID:
30347268.

14. Papaemmanuil E, Gerstung M, Bullinger L, Gaidzik VI, Paschka P, Roberts ND, et al. 2016; Genomic classification and prognosis in acute myeloid leukemia. N Engl J Med. 374:2209–21. DOI:
10.1056/NEJMoa1516192. PMID:
27276561. PMCID:
PMC4979995.


16. Gao P, Zhang R, Li Z, Ding J, Xie J, Li J. 2019; Challenges of providing concordant interpretation of somatic variants in non-small cell lung cancer: A multicenter study. J Cancer. 10:1814–24. DOI:
10.7150/jca.29535. PMID:
31205538. PMCID:
PMC6547979.


17. National Institute of Food and Drug Safety Evaluation. 2018. 차세대염기서열분석(Next Generation Sequencing) 임상검사실 인증 검사분야별 가이드라인체세포(Somatic). National Institute of Food and Drug Safety Evaluation.
18. Kim H, Yun JW, Lee ST, Kim HJ, Kim SH, Kim JW, et al. 2019; Korean Society for Genetic Diagnostics Guidelines for Validation of Next-generation Sequencing-based Somatic Variant Detection in Hematologic Malignancies. Ann Lab Med. 39:515–23. DOI:
10.3343/alm.2019.39.6.515. PMID:
31240878. PMCID:
PMC6660343.


Go to :







 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download