초록
혈액종양 질환 관련 보고서는 검사실마다 다양한 형식으로 작성되고 있으나 최선의 환자 진료를 위하여서는 간결하고 명확한 정보를 줄 수 있는 보고서의 표준화가 필요하다. 이를 위하여 대한 진단혈액학회에서 구성한 진단혈액 표준화위원회는 급성백혈병 보고서를 중심으로 표준안을 제안하였다. 이 표준안을 통하여 임상 의사들과의 소통 및 환자 진료에 도움이 될 것으로 기대한다
Abstract
Reports on hematological neoplasms are produced in various formats in different laboratories. For best patient care, standardization of reports adopting a format that provides concise and clear information is necessary. To this end, the Diagnostic Hematology Standardization Committee, organized by the Korean Society for Laboratory Hematology, has proposed a standardized format for reporting diagnostic test results for acute leukemia patients. It is hoped that this standardization will broadly improve communication with clinicians and improve patient care.
혈액종양의 진단은 WHO Classification of Tumours of Haematopoietic and Lymphoid Tissues에 근거하여 상당 부분 진단적 동의가 이루어진 상황이다. 2017년도에는 4번째 개정판이 나왔고 전문가들의 의견과 자료를 바탕으로 꾸준히 수정 및 개선이 이루어지고 있다[1]. 그러나 혈액종양 질환 관련 보고서는 검사실마다 매우 다양한 형식으로 작성되고 있다. 보고서는 판독의사가 임상의사에게 전달하고자 하는 바가 명확하여야 하고 누락되는 정보가 없어야 한다. 최선의 환자 진료를 위해서는 판독의, 진료의 간에 정보의 전달을 원활히 할 수 있는 방안이 필요하며 이를 위하여 간결하고 명확한 정보를 줄 수 있는 보고서의 표준화가 필요하다[2]. 골수검사 보고서의 경우, 2008년도 국제혈액학표준화기구(International Council for Standardization in Hematology, ICSH)와 2016년도 미국병리학회(College of American Pathologists, CAP)에서 제안한 표준안이 있고[2, 3], 유세포검사 보고서의 경우 2006년도 Beth-esda Consensus Conference에서 제안된 유세포 분석 및 보고에 대한 표준안과 2007년도 임상검사표준연구소(Clinical Laboratory Standardization Institute, CLSI) 지침이 있으나[4, 5] 최근 실정을 반영하여 업데이트한 보고서는 없다. 세포/분자유전 보고서는 2014년도 유럽인간유전학회(European Society of Human Genetics, ESHG)의 Quality Committee와 2016년도 미국 의료유전학 및 유전체학회(American College of Medical Genetics and Genomics, ACMG)에서 제안한 지침 등이 있다[6, 7]. 그러나 국내에서는 2010년도 대한진단혈액학회에서 제안된 골수검체 및 보고서 표준화 지침과 2019년도 보고된 말초혈액 혈구 판독검사의 표준안[8] 이외에는 표준화된 작성 지침이 없으며 차세대염기서열 보고서의 경우는 국내외 모두 제안된 표준화 보고서는 없는 것이 현 실정이다.
이에 대한진단혈액학회에서 구성한 진단혈액 표준화위원회는 혈액종양 질환 중 대표적인 질환인 급성백혈병의 진단과 관련된 보고서를 중심으로 보고서의 표준안을 제안하고자 한다. 급성백혈병 결과보고서를 골수검사 보고서, 유세포검사 보고서, 세포유전검사 보고서, 분자유전검사 보고서로 나누고 분자유전검사 보고서를 차세대염기서열분석과 그 외 분자유전검사로 크게 분류하였다. 또한, 국문보고서를 기본으로 제시하였고 영문보고서는 Supplementary에 추가하였다. 국내외의 지침 및 연구들을 참고한 이 표준안을 통하여 검사 보고서의 표준화뿐 아니라 임상 의사들과의 소통 및 환자 진료에도 도움이 될 것으로 기대한다.
2008년 골수검체 및 골수검사보고서의 표준화를 위한 ICSH의 지침[3], 2010년도 대한진단혈액학회에서 제안된 골수검체 및 골수검사 보고서 표준화 지침 및 2016년 CAP에서 발간된 골수검사 공통 보고서[2]를 기반으로 보고서에 포함되어야 할 필수 내용을 포함하여 작성하였다.
해당 표준화 보고서를 완전성의 관점에서 필요한 것으로는 첫째, 일차적으로 골수형태를 잘 기술할 수 있도록, 둘째, 골수검체에 추가로 시행한 검사 결과를 넣을 수 있게, 셋째, 결과보고순서를 정하고, 넷째, 정확한 자료 구성을 위해 필요한 구성요소를 포함하고, 다섯째, 표준보고서에 포함될 임상정보와 검사실 정보를 선별하여 표준화 보고서를 작성하는 데 주안점을 두었다.
급성백혈병의 진단을 위한 골수검사 결과보고서에는 1) 임상정보, 2) 말초혈액도말, 3) 골수세포 감별 계수, 4) 골수흡인소견, 5) 골수생검소견, 6) 골수응고절편소견, 7) 그 외 특수검사, 8) 이전 골수소견 및 9) 진단명 및 추가설명(comments)이 포함되어야 한다. 진단명의 경우는 각 병원의 정책에 따라 가장 먼저 기술될 수도 있고, 가장 나중에 결론으로 기술될 수도 있다.
각 해당 내용을 상세히 살펴보면 1) 임상정보에는 임상소견(진단 및 치료력), 검체의 구성, 골수 검체 채취부위 및 단측 및 양측 시행 여부를 기술하고, 2) 말초혈액소견으로 적혈구, 백혈구, 혈소판의 수와 분포 및 이상세포를 기술한다. 3) 골수세포 감별 계수에서 유핵세포 감별에 포함되는 세포로는 모세포, 전골수구, 골수구, 후골수구, 대호중구, 분엽핵호중구, 호산구, 호염기구, 비만세포, 전단구, 단구, 림프구, 형질세포, 조직구, 적혈모구가 있으며, 거대핵세포, 깨진세포(smudge cell) 및 골모세포, 파골세포, 기질세포, 전이암세포 등의 비조혈세포는 유핵세포 감별 계수에 포함시키지 않는다. 4) 골수흡인소견은 검체의 적절성을 기술한 후, 적혈구계, 골수계, 혈소판계 등의 조혈세포의 수, 분포, 이형성을 우선 기록하고, 림프구, 형질세포 및 이상세포에 대해 기술한다. 5) 골수생검소견 역시 검체의 질에 대해 평가한 후, 세포 충실도 및 각 구성하는 세포의 수, 분포 등을 기술한다. 가능하면 골수절편에 대해 기술하고, 그 외 특수검사의 경우, 세포화학(cytochemistry), 면역조직화학(immunohistochemistry), 면역표현형(immunophenotyping), 세포유전학(cytogenetics), 분자유전학(molecular genetics) 등의 검사결과에 대해 기술한다. 이전 골수소견이 있다면 이전 결과를 포함한다. 그 외 진단과 관련된 검사 및 진단에 도움이 될 수 있는 참고사항이 있다면 추가설명으로 기술한다(Fig. 1, Supplementary Fig. 1).
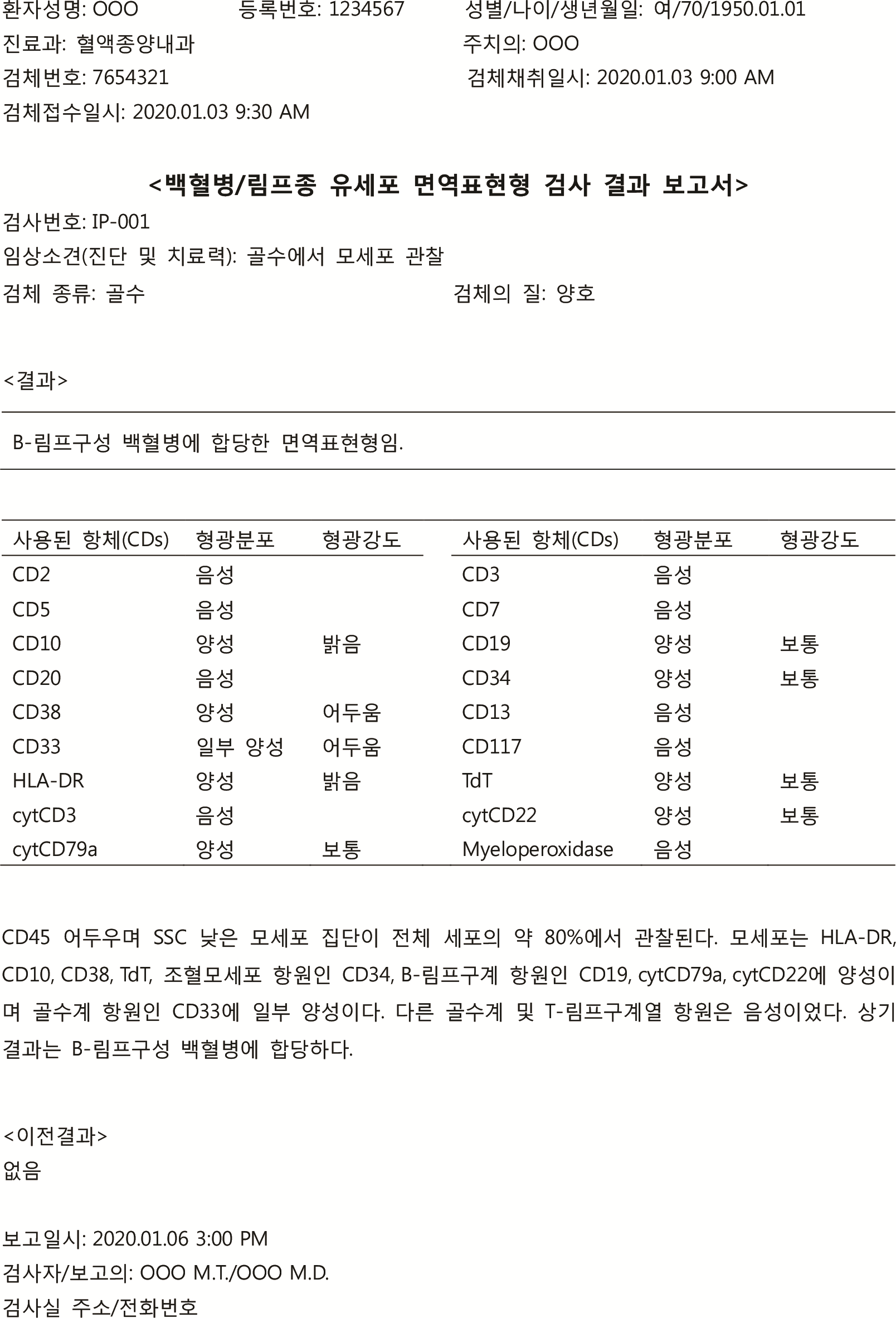
급성백혈병 면역표현형 유세포검사 보고서에 대한 표준안은 CLSI H43-A2 (종양성 림프조혈 세포의 임상적 유세포분석) [5] 및 2006 림프조혈종양의 면역표현형 분석의 Bethesda 국제 표준 권장안[4]을 바탕으로 보고서에 포함되어야 할 필수 내용을 포함하여 작성하였다.
급성백혈병의 진단을 위해서 임상정보, 형태학, 면역표현형 및 유전 정보 등 다양한 정보가 필요하며 이를 종합하여 판정하게 된다[1, 9]. 유세포검사 결과는 세포 계열을 판정하고, 분화의 정도 및 모세포 비율 등을 측정하는데 중요하다[10]. 또한, 진단 당시에 비정상 발현 표현형 등을 검출하여 이후 추적 검사 시 미세잔존질환 검출에도 유용하다[11-13]. 따라서 모세포 영역을 정확히 선정하는 것이 중요하며 여러 튜브로 검사를 시행한 경우, 동일한 집단에 대한 분석을 통해, 면역 표현형 결과를 제공하는 것이 중요하다.
급성백혈병의 진단을 위한 유세포검사 결과보고서에는 환자 정보, 검체 정보 및 검사와 관련된 정보, 그리고 결과가 포함되어야 한다. 환자 정보에는 이름, 등록번호, 생년월일, 나이, 성별, 처방한 의사, 임상소견(진단 및 치료력) 및 이전 유세포검사 결과, 다른 검사 결과 등을 포함할 수 있다. 검체 정보에는 검체 번호, 검체 채취일, 검체 접수일, 검체 종류(골수, 말초혈액), 검체의 질(상태, 세포생존율) 등을 기록하며, 검사 관련 정보에는 검사일, 검사자, 사용된 항체 등이 포함될 수 있다. 검사결과에는 모세포의 산란광(light scatter) 및 면역표현형을 기술한다. 특정 집단에 대비해 모세포가 차지하는 비율, 모세포의 형광분포(음성, 양성, 일부 발현), 정상 조혈 세포와 비교하여 모세포 집단의 형광 강도(어두움, 보통, 밝음, 다양함)를 기술해 준다. 면역표현형 결과에 대한 해석이 필요하며, 비정상 모세포 집단이 검출되지 않은 경우, 정상 세포 집단의 면역표현형 및 특성에 대해 기술한다. 비정상 모세포 분획이 검출된 경우, 면역표현형 및 감별진단을 포함해야 한다. 가능하면 WHO 분류에 근간한 해석을 해 주는 것이 좋으며[1], 관련검사 및 임상정보를 얻을 수 있는 경우에는 구체적인 진단명을 포함할 수 있다. 비고에는 검사의 제한점, 해석에 있어서의 제한점 및 판독 날짜, 판독의, 판독의의 연락처 등을 기입한다.
결과를 기술하는 경우 각 항체별 양성률을 기입하는 것은 어떤 것을 모집단으로 양성률이 기술된 것인지 불분명할 수 있어 권장되지 않고 양성이었거나 음성인 항원을 기술해주는 것이 적절하다. 예시로 작성한 급성백혈병 진단 시 유세포검사 결과보고서는 Fig. 2, Supplementary Fig. 2와 같다. 미세잔존질환 확인을 위한 유세포검사의 경우, 결과보고서에 포함되어야 할 사항이 조금 상이하여 Fig. 3, Supplementary Fig. 3에 제시되어 있다.
세포유전검사 보고서는 염색체검사 결과와 FISH 결과보고서에 필수적으로 포함되어야 할 내용을 바탕으로 작성하였다[6, 7, 14, 15]. 세포유전검사는 검사가 의뢰된 목적을 파악하고, 이에 대한 유용한 정보를 제공할 수 있도록 보고하는 것이 중요하다.
염색체검사 결과보고서에는 환자인적사항 정보, 염색체가 의뢰된 검체 정보, 염색체 검사방법, 염색체 결과, 염색체 결과 해석, 추천검사, 검사의 제한점 등의 내용이 포함되어야 한다. 염색체 결과는 국제표준명명법(International System for Human Cytogenomic Nomenclature)에 근거하여 염색체 핵형을 표기하여야 한다. 이러한 표기법은 세포유전학을 전공하지 않은 임상의가 의미를 이해하기에 어려우므로 결과 해석부분에서 염색체 이상 결과를 이해하기 쉽게 설명해야 한다. 또한 이러한 염색체 결과가 분자유전학적으로 어떤 의미인지, 진단, 예후판정, 치료방침 결정에 어떤 영향을 미칠 수 있는지도 기술하는 것이 필요하다. 또한 과거 염색체 결과도 현 결과보고서에 같이 포함시켜서 현재의 환자 상태를 분명히 전달할 수 있도록 하는 것이 유용한 보고가 될 수 있다. 추천검사는 염색체 결과를 근거로 하여 표적이 되는 FISH 검사 또는 RT-PCR, DNA 유전자 변이 검사 등을 제시하는 것이 필요하다. 핵형 분석 이미지(karyogram)는 적어도 2개 이상 첨부할 것을 추천하는데 1개의 부클론(subclone)마다 1개의 핵형 분석이미지를 추가한다(Fig. 4, Supplementary Fig. 4).
FISH 결과보고서에는 염색체 결과서에 포함되었던 내용과 같이 환자인적사항 정보, FISH가 의뢰된 검체 정보, FISH 검사방법, FISH 결과, FISH 결과 해석, 추천검사, FISH 검사의 제한점 등의 내용이 포함되어야 한다. FISH 검사방법에는 사용한 FISH 탐색자(probe)의 종류(break apart, dual color dual fusion, centromere probe 등)를 기록하고, 상품화된 탐색자를 사용한 경우는 제조회사 이름을 기록할 수 있고, 자가제조한 탐색자를 사용한 경우는 bacterial artificial chromosome (BAC) 이름 등을 기록할 수 있다. 분석한 세포 종류(간기세포 또는 중기세포)와 세포 수도 기록한다. 또한 FISH를 분석한 세포가 배양하지 않은 검체에 포함된 세포인지, 또는 배양 후 검체에 포함된 세포를 대상으로 분석했는지 여부도 기록하는 것이 필요하다. 형질세포종 환자를 대상으로 한 FISH 보고서 방법에는 형질세포 농축 방법으로 CD138 자기구슬(magnetic bead), FICTION (fluorescence immunophenotyping and interphase cytogenetics as a tool for investigation of neoplasia) 등을 사용하였는지 여부를 기술하고, 농축 후 형질세포의 순도(purity)가 얼마였는지도 기록하는 것이 필요하다.
FISH 결과도 염색체 핵형표기법과 마찬가지로 국제표준명명법에 근거하여 표기하여야 한다. 이러한 FISH 결과를 누구나 이해하기 쉽게 설명하고 요약식으로 보고하는 것이 필요하다. 또한 각 검사실에서 설정한 기준치 값도 결과지에 같이 제시하는 것이 환자의 결과 판정을 이해하는데 도움이 될 수 있다. FISH 결과는 “양성” 또는 “음성”으로 표기하는 것 보다는 “비정상” 또는 “정상”으로 표기하는 것이 더 바람직하다. 또한 비정상 결과가 나온 FISH 결과를 근거로 추적관찰 시 유용하게 이용될 수 있는 FISH 탐색자를 추천 검사에 제시하는 것이 필요하다. 또한 FISH 이미지를 첨부할 수 있다(Fig. 5, Supplementary Fig. 5).
유전자 돌연변이 결과 보고 양식에는 검체의 종류, 분석 대상 유전자명, 검사방법, 검사 대상 유전자의 위치 및 염기서열의 미국 국립생물공학정보센터(National Center for Biotechnology Information, NCBI) 참조번호(reference number), 분석된 유전자 이상의 상세 정보, 발견된 유전자 이상의 해석 및 그 의의, 검사방법의 제한점, 참고문헌 및 보고자명 등을 순차적으로 기술하는 것을 추천한다. 예시로, CEBPA 유전자의 직접염기서열 분석법에 의한 돌연변이 분석에 대한 결과 보고안을 그림에 나타내었다(Fig. 6, Supplementary Fig. 6).
실시간 정량 PCR법에 의한 유전자 이상의 정량 분석의 결과 보고 양식에는 검체의 종류, 검사법, 검사에 사용된 시발체 및 탐색자의 종류, 참고치, 결과 보고 방법의 정확한 명칭 및 사용된 계산법, 현재 결과, 해당 환자의 동일 검사에 대한 이전 결과, 보고자명 등을 순차적으로 기술하는 것을 추천하며, 예시로 BCR-ABL1 전좌에 대한 정량 PCR 법에 대한 결과 보고안을 그림에 나타내었다(Fig. 7, Supplementary Fig. 7) [16].
급성백혈병의 유전자 분석에 널리 사용되는 역전사-다중 PCR 법인 Hemavision (DNA Technology, Aarhus, Denmark) 결과 보고 양식에는 검체의 종류, 검사법, 선별 및 분할(split-out) 결과, Hemavision 검사로 검출 가능한 유전자 이상의 종류, 검사의 의의 및 제한점 및 보고자명 등을 순차적으로 기술하는 것을 추천하며, 예시를 그림에 나타내었다(Fig. 8, Supplementary Fig. 8) [17].
NGS 보고서 구성 시 가장 중요한 요소 중 하나는 간결성이다. 보고서는 짧고 간단하며 요점을 한눈에 파악할 수 있도록 구성하여야 한다. 임상적으로 중요한 정보는 보고서의 시작 부분에 위치하는 것이 좋고, 두 번째 페이지 이후의 정보는 주치의에게 전달되지 않을 가능성이 높다는 것을 염두에 두어야 한다. 중요한 정보는 눈에 잘 들어오도록 구성하여야 하며, 가능하다면 그래프, 차트, 표로 정보를 제시함으로써 보고서의 전체적인 명확성을 높일 수 있다(Fig. 9, Supplementary Fig. 9).
검출된 모든 유전자 변이는 HUGO 명명법(http://www.genenames.org), Human Genome Variation Society (HGVS) 명명법(http://www.hgvs.org), NCBI 표준 시퀀스(RefSeq, https://www.ncbi.nlm.nih.gov/refseq/), 표준 유전체(Standard genome build version)가 포함되어야 한다[18]. 이외에 일반적으로 잘 알려진 명칭을 추가로 포함할 수 있다. 예를 들어 TERT 유전자의 프로모터 부위 변이의 경우, c.1-124C>T (HGVS 명명법)로 표기하고 괄호 안에 일반적으로 알려진 명칭(TERT C228T)을 같이 보고하는 것이 의미 전달에 도움이 될 수 있다.
검출된 모든 유전적 변이는 미국임상종양학회(American Society of Clinical Oncology, ASCO)/분자병리학회(Association for Molecular Pathology, AMP) 지침에 따라 Tier I, 임상적 의의가 강한 변이(strong clinical significance); Tier II, 임상적 의의가 있을 수 있는 변이(potential clinical significance), Tier III, 임상적 의의가 불분명한 변이(unknown clinical significance), Tier IV, 양성 혹은 양성 가능성이 높은 변이(benign or likely benign)의 4 Tier 시스템으로 분류하여 보고한다[19]. 변이는 임상적으로 중요한 내림차순으로 보고되어야 한다. 보고서에 Tier IV, 양성 혹은 양성 가능성이 높은 변이는 포함하지 않는 것이 좋다(Table 1).
ASCO/AMP 치침의 단계(tier) 체계
변이는 표의 형태로 보고하는 것이 좋으며, 변이의 분류, 유전자명, 표준 시퀀스, 뉴클레오타이드 변이, 아미노산 변이, 대립유전자 빈도(variant allele frequency, VAF)를 포함하며(Table 2), 이에 추가로 깊이(depth), 정상인 빈도 등을 보고할 수 있다. 주치의의 의사결정에 도움을 주기 위해 발견된 유전자 변이에 대한 임상적, 병리학적인 맥락에서 해석적 의견을 제공하는 것이 유용하다. Tier I과 Tier II의 변이에 대해서는 해석적 의견을 제시하는 것이 좋으며, Tier III에 대한 상세한 해석은 보고서의 간결성과 균형 있게 이루어져야 한다. 해석적 의견은 특정 암에 대한 변이의 빈도, 기능의 영향, 진단 및 예후와의 관련성을 포함할 수 있다.
방법론적 세부 사항 및 검사의 한계점에 대해 보고서 하단에 제시하여야 하며, 검사방법 및 성능, 특히 커버리지 누락 부위(coverage gaps), 검출 한계(limit of detection), 최소 깊이(minimal coverage depth) 등을 포함한다(Table 3). 보고서에는 실제로 검사한 대상에 대한 세부 사항이 포함되어야 한다. 모든 유전자의 전체 염기서열을 분석하지 않는다면, 검사한 유전자의 특정 위치를 포함하여야 한다. 시퀀싱 플랫폼, 데이터 분석 파이프라인, 참조 유전체 버전, 변이를 필터링하고 우선 순위를 지정하는 데 사용하는 시스템 및 논리도 포함되어야 한다. 또한 깊은 인트론 부위(deep intron)나 유전자 복제수 변이(copy number variants)와 같은 구조적 이상(structural variant) 등 시퀀싱 방법에 의해 검출하지 못할 가능성이 있는 유전자 이상에 대한 기술을 포함한다. 정도관리 지표들을 자세하게 보고서에 포함시키는 것을 권장하지 않으나, 주요 지표를 포함하여 주치의가 품질 지표가 충족되었음을 확인 가능하도록 포함할 수 있다.
방법 및 한계점 포함 요소 및 예시
REFERENCES
1. Swerdlow SH, Campo E, editors. 2017. WHO classification of tumours of haematopoietic and lymphoid tissues. Revised 4th ed. International Agency for Research on Cancer;Lyon, France:
2. Sever C, Abbott CL, de Baca ME, Khoury JD, Perkins SL, Reichard KK, et al. 2016; Bone marrow synoptic reporting for hematologic neoplasms: Guideline from the College of American Pathologists Pathology and Laboratory Quality Center. Arch Pathol Lab Med. 140:932–49. DOI: 10.5858/arpa.2015-0450-SA. PMID: 26905483.

3. Lee SH, Erber WN, Porwit A, Tomonaga M, Peterson LC. 2008; ICSH guidelines for the standardization of bone marrow specimens and reports. Int J Lab Hematol. 30:349–64. DOI: 10.1111/j.1751-553X.2008.01100.x. PMID: 18822060.

4. Wood BL, Arroz M, Barnett D, DiGiuseppe J, Greig B, Kussick SJ, et al. 2007; 2006 Bethesda International Consensus recommendations on the immunophenotypic analysis of hematolymphoid neoplasia by flow cytometry: optimal reagents and reporting for the flow cytometric diagnosis of hematopoietic neoplasia. Cytometry B Clin Cytom. 72:S14–22. DOI: 10.1002/cyto.b.20363. PMID: 17803189.
5. Clinical and Laboratory Standards Institute. 2007. Clinical flow cytometric analysis of neoplastic hematolymphoid cells;Approved guideline-Second edition. CLSI document H43-A2. Clinical and Laboratory Standards Institute;Wayne, PA:
6. Claustres M, Kožich V, Dequeker E, Fowler B, Hehir-Kwa JY, Miller K, et al. 2014; Recommendations for reporting results of diagnostic genetic testing (biochemical, cytogenetic and molecular genetic). Eur J Hum Genet. 22:160–70. DOI: 10.1038/ejhg.2013.125. PMID: 23942201. PMCID: PMC3895644.

7. Mikhail FM, Heerema NA, Rao KW, Burnside RD, Cherry AM, Cooley LD. 2016; Section E6.1-6.4 of the ACMG technical standards and guidelines: chromosome studies of neoplastic blood and bone marrow-acquired chromosomal abnormalities. Genet Med. 18:635–42. DOI: 10.1038/gim.2016.50. PMID: 27124785.
8. Kwon JA, Kim YG, Park G, Kim JM, Cho YU, Huh J, et al. 2019; Recommendation for the peripheral blood cell morphology report. Lab Med Online. 9:115–25. DOI: 10.3343/lmo.2019.9.3.115.
9. Arber DA, Orazi A, Hasserjian R, Thiele J, Borowitz MJ, Le Beau MM, et al. 2016; The 2016 revision to the World Health Organization classification of myeloid neoplasms and acute leukemia. Blood. 127:2391–405. DOI: 10.1182/blood-2016-03-643544. PMID: 27069254.

10. Béné M, Nebe T, Bettelheim P, Buldini B, Bumbea H, Kern W, et al. 2011; Immunophenotyping of acute leukemia and lymphoproliferative disorders: a consensus proposal of the European LeukemiaNet Work Package 10. Leukemia. 25:567–74. DOI: 10.1038/leu.2010.312. PMID: 21252983.

11. Theunissen P, Mejstrikova E, Sedek L, van der Sluijs-Gelling AJ, Gaipa G, Bartels M, et al. 2017; Standardized flow cytometry for highly sensitive MRD measurements in B-cell acute lymphoblastic leukemia. Blood. 129:347–57. DOI: 10.1182/blood-2016-07-726307. PMID: 27903527. PMCID: PMC5291958.


12. Wood BL. 2016; Principles of minimal residual disease detection for hematopoietic neoplasms by flow cytometry. Cytometry B Clin Cytom. 90:47–53. DOI: 10.1002/cyto.b.21239. PMID: 25906832.

13. Schuurhuis GJ, Heuser M, Freeman S, Béné M-C, Buccisano F, Cloos J, et al. 2018; Minimal/measurable residual disease in AML: a consensus document from the European LeukemiaNet MRD Working Party. Blood. 131:1275–91. DOI: 10.1182/blood-2017-09-801498. PMID: 29330221. PMCID: PMC5865231.


14. Rack KA, van den Berg E, Haferlach C, Beverloo HB, Costa D, Espinet B, et al. 2019; European recommendations and quality assurance for cytogenomic analysis of haematological neoplasms. Leukemia. 33:1851–67. DOI: 10.1038/s41375-019-0378-z. PMID: 30696948. PMCID: PMC6756035.


15. McGowan-Jordan J, Simons A, Schmid M. 2016; ISCN: an international system for human cytogenomic nomenclature (2016). Cytogenetic and Genome Research. 149:Basel, Switzerland;1–2.
16. Cross NCP, White HE, Evans PAS, Hancock J, Copland M, Milojkovic D, et al. 2018; Consensus on BCR-ABL1 reporting in chronic myeloid leukaemia in the UK. Br J Haematol. 182:777–88. DOI: 10.1111/bjh.15542. PMID: 30125955. PMCID: PMC6175193.


17. Ohgami RS, Arber DA. 2013; Challenges in consolidated reporting of hematopoietic neoplasms. Surg Pathol Clin. 6:795–806. DOI: 10.1016/j.path.2013.08.001. PMID: 26839198.

18. Richards S, Aziz N, Bale S, Bick D, Das S, Gastier-Foster J, et al. 2015; Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med. 17:405–24. DOI: 10.1038/gim.2015.30. PMID: 25741868. PMCID: PMC4544753.


19. Li MM, Datto M, Duncavage EJ, Kulkarni S, Lindeman NI, Roy S, et al. 2017; Standards and guidelines for the interpretation and reporting of sequence variants in cancer: a joint consensus recommendation of the Association for Molecular Pathology, American Society of Clinical Oncology, and College of American Pathologists. J Mol Diagn. 19:4–23. DOI: 10.1016/j.jmoldx.2016.10.002. PMID: 27993330. PMCID: PMC5707196.






 PDF
PDF Citation
Citation Print
Print











 XML Download
XML Download