INTRODUCTION
수두-대상포진 바이러스(varicella zoster virus, VZV)는 수두(varicella, chicken pox)와 대상포진(herpes zoster, shingles)을 발병시킨다. 수두는 VZV 바이러스가 처음 감염했을 때 나타나는 질병으로 주로 소아에게 발생하며 미열, 두통, 피부 발진 등의 증상이 유발된다 (1). 초기감염 후 바이러스는 배근 신경절로 이동하여 휴지기에 들어가는 잠복감염을 하게 되며, 사람의 면역력이 저하되었을 때 잠복기의 바이러스가 다시 활성화되면 대상포진이 발생하며 국부적인 통증과 수포성 발진을 일으킨다 (2) (3). VZV는 유일하게 사람만을 숙주로 삼아 숙주 특이성이 높고, 자연 상태에서는 사람 이외의 동물에게 병원성이 나타나지 않는 것으로 알려져 있다 (4).
수두와 대상포진은 모두 약독화 생백신으로 예방할 수 있다. 1974년 일본에서 수두에 걸린 Oka라는 3세 남아로부터 분리한 야생주인 pOka주를 계대배양으로 약독화하여 vOka 백신주가 개발되었다. 약독화 방법에는 이종세포인 기니피그 배아 섬유아세포에서의 계대배양을 포함하였다 (5), (6). 이렇게 개발된 수두백신은 1988년부터 일본에서 상품화되었다. 이후 미국에서 vOka를 기반으로 하여 Merck사의 Varivax와 GSK사의 Varilrix가 개발되었다(7). 국내에서는 한국인 수두환자에서 분리한 바이러스를 약독화한 바이러스인 MAV/06주를 사용하여 Suduvax가 개발되었다 (8). Suduvax는 한국에서는 1994년부터 상용화되었고 국제적으로는 1998년부터 판매되었다.
VZV 약독화 백신의 작용 기전은 아직까지 명확하게 규명되어 있지 않다. 그러나 pOka주의 염기서열 비교 (9), (10), 그리고 여러 백신주와 임상주들간의 게놈 서열비교(11)에 의해 백신주에만 나타나는 특이 염기서열이 제시된 바 있다. 한편 일부의 바이러스에 변이가 축적되면 바이러스 집단에서 게놈의 변이를 유도하고 그 결과 다양한 변종이 발생하게 되는데 이렇게 발생한 바이러스 개체군은 약독화와 같은 생물학적 특성을 결정할 수 있다 (12). 변종 바이러스 개체군을 quasispecies라 하며 이는 바이러스의 유전적 다형성을 가장 잘 표현할 수 있는 단어이다 (13). 유전적 다형성에 의해 신경독성이 변할 수 있다는 것이 Mumps 백신에서도 보고되었다 (14).
본 연구에서는 Next generation sequencing (NGS) 기법을 사용하여 대표적인 수두 백신주(Suduvax, Varilrix, Varivax)의 SNP 분석과 유전적 다형성 분석을 통해 약독화에 따른 각 백신주가 어떠한 유전학적 특징과 다양성을 가지고 있는지 분석하고자 하였다.
Go to : 
재료 및 방법
DNA 추출 및 q-PCR
백신주는 ㈜GC녹십자(Yongin, Gyeonggi, Korea)로부터 제공 받은 Suduvax (Lot No. MG1111-FF-012), Varilrix (Lot No. A70CC807B), Varivax (Lot. No M015462) 제품을 사용하였다. 분말 상태의 제품 1개당 PBS 0.5 ㎖에 녹여 1.5 ㎖ E-tube에 담아 준비하였다.
준비한 백신주 시료를 ExgeneTM Cell SV mini kit (GeneAll Biotechnology, Seoul, Korea)를 이용하여 DNA를 추출하였다. 바이러스성 DNA와 세포성 DNA의 양을 확인하기 위해 IQ SYBR Green SuperMix (BIO-RAD, Hercules, CA, USA)로 q-PCR을 진행하였다. 기존에 알려진 참고문헌을 활용하여 세포성 DNA, VZV DNA를 각각 검출할 수 있는 2쌍의 primer를 이용하였다. 세포성 DNA양을 확인하기 위해 glyceraldehyde-3-phosphate dehydrogenase (GAPDH) 부위를 증폭하는 primer를 사용하였고, VZV의 바이러스성 DNA양을 확인하기 위해 ORF4 부위를 증폭하는 primer를 사용하였다 (15), (16). IQ SYBER Green SuperMix 10 ㎕, DNA template 1 ㎕, 10 pmol 농도의 forward primer (5'-ATCTCCGTACAACATCAACT-3')와 reverse primer (5'-CGATTTTCCAAGAGAGACGC-3') 각각 0.6 ㎕를 혼합하고 전체 혼합액의 양이 20 ㎕가 되도록 증류수를 넣어주었다. q-PCR은 initial denaturation 94℃ 240초 1회 이후 denaturation 94℃ 30초, annealing 48.9℃ 30초, extension 72℃ 30초 조건으로 45회 반복하였다. 바이러스 DNA에 대하여 시행한 q-PCR의 Ct값를 이용해 바이러스 시료 각각의 DNA양을 정량하였다. 이를 토대로 바이러스 간의 DNA 양의 균등화를 통해 객관적인 비교를 할 수 있도록 하였다.
차세대 시퀀싱(Next generation sequencing: NGS)
추출한 바이러스 DNA의 전체 게놈 시퀀싱은 Illumina사(San Diego, CA, USA)의 Hi-Seq 2500 rapid 시스템으로 실시하였고, 이 과정은 ㈜테라젠이텍스(Suwon, Gyeonggi, Korea)에 의뢰하였다. DNA library 제작은 Illumina TruSeq DNA Sample Preparation kit v2를 사용하였고, 제작된 library를 Hi-Seq 방식으로 시퀀싱하고 Illumina version 1.8을 사용하여 품질이 낮게 나온 read들을 제거하였다. 일정한 품질이 보장된 read들은 CLC Geneomics Workbench version 5.0 프로그램에 기본 파라미터 값을 적용하고 NCBI에 등록되어 있는 MAV/06 (JF306641), Varilrix (DQ008354), Varivax (DQ008355)주를 참고주로 하여 mapping assemble을 수행하였다. Mapping assemble의 결과 하나의 완성된 염기서열을 얻을 수 있었고 이를 NCBI GenBank에 등록하였다: Suduvax MT370825, Varilrix MT370826, Varivax MT370827.
ORF 위치 정보
게놈 전체의 염기서열을 얻은 백신주의 ORF는 각각 mapping에 사용한 서열을 기준으로 local blast를 진행하여 특정하였다. 그리고 CLC Sequence Viewer (ver.6.1, http://www.clcbio.com/index.php)의 ORF 확인 기능, NCBI에서 제공하는 ORF Finder 프로그램과 Bioedit Sequence Alignment Editor (version 7.0.9.0, http://www.mbio.ncsu.edu/BioEdit/bioedit.html)를 이용하여 온전한 형태의 시작코돈과 종결코돈을 갖는 ORF인지 확인하였다.
단일 염기 다형성 분석
DNA 대표 염기서열에서 차이를 보이는 유전적 변화 또는 변이를 단일 염기 다형성(Single nucleotide polymorphism; SNP) 이라고 한다. 백신주의 NGS 분석을 통해 얻어진 대표염기서열(Consensus sequence)을 Bioedit으로 읽고, ClustalW2 (version 2.1)를 이용하여 VZV의 참고주인 Dumas주의 게놈 염기 서열(NC001348)과 함께 정렬시켜 동일한 위치에서의 SNP 분석을 진행하였다.
유전적 다형성 분석
유전적 다형성은 개체군에 존재하는 임의의 동일한 위치 염기에서 2개 이상의 염기를 함께 가지고 있는 것을 말한다. 백신주의 NGS 분석으로 얻은 전체 염기서열 결과 파일(Counts 파일)로 염기서열의 각 위치별 A, C, G, T의 mapping 개수에 관한 정보를 얻을 수 있었다. Counts file에서 각 위치별로 mapping된 염기들 중, 가장 많은 비율을 차지하는 염기가 다수 염기(Major base)가 되고, 해당위치에서의 대표염기서열로 결정된다. 두 번째로 많은 비율을 차지하는 염기가 소수 염기(Minor base)로 결정된다. 백신주의 NGS 분석으로 얻은 Counts file을 이용하여 다수 염기와 소수 염기의 백분율을 구하였다. 이 때, 특정 위치에서 mapping 된 read의 개수가 35개 이상인 경우에만 유전적 다형성 분석에 유의하다고 판단하였다. 그리고 소수 염기의 비율이 5% 이상인 위치를 유전적 다형성 부위(Genetically polymorphic site; GPS)라고 명명하고, 이 위치들만 유전적 다형성 분석에 사용하였다 (17). Counts file은 우리 실험실에서 개발한 GPA (Genetic Polymorphism Analyzer) 프로그램(https://github.com/lis123kr/GPA)의 input 파일로 사용하여 분석하였다. 이 프로그램은 서열간의 소수염기비율을 비교하고, 유전적다형성을 찾아 다양한 형태로 분석한 결과를 보여준다.
Go to : 
결과
백신주 간의 SNP 분석
수두 백신주 간의 대표 염기서열이 다른 SNP 분석을 통해 어떠한 분자유전학적 차이가 있는지 알아보고자 하였다. VZV의 게놈 구조상 terminal repeat long (TRL)과 internal repeat long (IRL), internal repeat short (IRS)와 terminal repeat short (TRS)는 각각 역상보적인 관계로, 중복으로 분석되는 것을 피하고자 TRL과 TRS를 제외하고 분석하였다. TRL과 TRS를 제외한 부위에서 세 백신주 사이에 염기가 서로 다른 SNP는 모두 38 개로 분석되었다 (Table 1). Open reading frame (ORF)에서 29 개, non-coding region (NCR)에서 9 개로 나타났다. 치환으로 인한 SNP는 모두 29 개이었으며, transition (22개)이 transversion (7개) 보다 많이 발생하였다. ORF 지역에서의 염기 치환으로 인해 아미노산이 변하지 않는 동의적 변이는 8개로 모두 transition으로 인해 발생하였고, 아미노산 서열이 변하는 비동의적 변이는 모두 17 개로 10 개의 transition과 7 개의 transversion으로 인하였다. NCR에서의 치환 4 곳은 모두 transition이었다. 특이하게도 transversion 7 군데는 R1 지역에서 2 군데, R2 지역에서 4 군데, 그리고 R3지역에서 1 군데 등, 모두 R지역에서 관찰되었다(Data not shown).
삽입/결실은 ORF에서 4 군데, NCR에서 5 군데 관찰되었다. ORF에서의 삽입/결실은 ORF11의 R1에서 3 군데와 ORF22의 R3에서 한 군데에서 관찰되었다. NCR의 삽입/결실 중 두 개는 NCR62/63의 R4와 ORI 지역에서 관찰되었다. R지역에서의 삽입/결실에는 여러 개의 뉴클레오티드가 관여하였으며, 그 숫자는 R1에서 세 군데에서 15 개씩, R3에서 27 개, 그리고 R4에서 54 개였다. 이들 숫자는 무작위적인 것이 아니라 각 R지역에서의 반복 단위 길이와 일치하기 때문에 실제로 이 R지역에서의 삽입/결실이 반복 단위로 이루어졌는지 알아 보았다. Fig. 1에 나타낸 바와 같이 R1에서는 15 bp로 구성된 B반복단위가, R3에서는 9 bp로 구성된 A반복단위 3 개가, 그리고 R4에는 27 bp로 구성된 A반복단위 2 개가 삽입/결실에 관여한 것을 알 수 있다.
SNP 중 세 백신주 모두 다른 경우는 없었다. 치환에 의한 29 개의 SNP에서 Suduvax만 다른 경우는 10 군데, Varilrix만 다른 경우는 6 군데, 그리고 Varivax만 다른 경우는 13 군데로 Varivax의 염기서열이 다름으로써 생성된 SNP가 가장 많았다. R지역에서의 삽입/결실에 의한 SNP의 경우 Suduvax에서만 다르고, Varilrix와 Varivax에서는 동일하게 관찰되었다.
백신주의 유전적 다형성 분석
소수 염기의 비율이 5% 이상인 부위를 GPS라 한다. Suduvax에서 89 개, Varilrix에서 68 개, Varivax에서 116 개로, GPS의 수는 Varivax에서 가장 많았고, Varilrix에서 가장 적었다(Table 2). 그러나 GPS에서의 소수 대립유전자형 빈도(Minor allele frequency, MAF)의 평균은 Varilrix에서 제일 높았고, Varivax에서 제일 낮았다. GPS는 VZV 백신주 게놈 전체에 골고루 분포하지 않고 특정 부위에 비교적 많이 관찰되는 것을 알 수 있다(Fig. 2). GPS가 상대적으로 많이 관찰되는 ORF는 ORF11, 14, 22, 31, 55, 그리고 62이다. 이 중 ORF11, 14, 22 지역은 각각 R1, R2, R3 지역을 포함하므로 이들 ORF에서의 GPS 형성에 R 지역의 역할을 알아보고자 하였다. Table 2에서 볼 수 있는 바와 같이 세 백신주 모두에서 ORF11, 14, 22의 GPS는 대부분 이들 ORF에 포함되어 있는 R1, R2, R3에 기인한 것임을 알 수 있었다. 특히 세 백신주 모두에서 ORF11의 GPS는 100% R1에 의한 것이며, ORF14의 GPS는 각 백신주에서 하나를 제외한 나머지가 R2에 기인한 것임을 알 수 있다. 특이하게도 ORF14의 GPS 중 R2에 속하지 않는 하나의 GPS는 세 백신주 모두에서 같은 위치였다(Dumas 기준 19431 위치). ORF14/R2나 ORF22/R3에서 세 백신주 사이에 GPS의 개수나 평균 MAF 값에서 큰 차이는 없는 것으로 보인다(Table 2). 그러나 ORF11/R1의 경우, Oka-유래 두 백신주에 비해 Suduvax에서 훨씬 많은 15 개의 GPS가 관찰되었다. Varilrix와 Varivax에서는 각각 2 개의 GPS가 관찰되었으며, 이 두 GPS의 게놈상 위치는 동일하였다. ORF31과 55에서는 모든 백신주에서 3 개 이상의 GPS가 관찰되었으며, R 지역을 포함하는 ORF를 제외하면 ORF62에서 가장 많은 GPS가 관찰되었다(Fig. 2). 세 백신주의 ORF별 GPS의 수와 평균 MAF 값은 Supplemented Table 1에 나타나 있다.
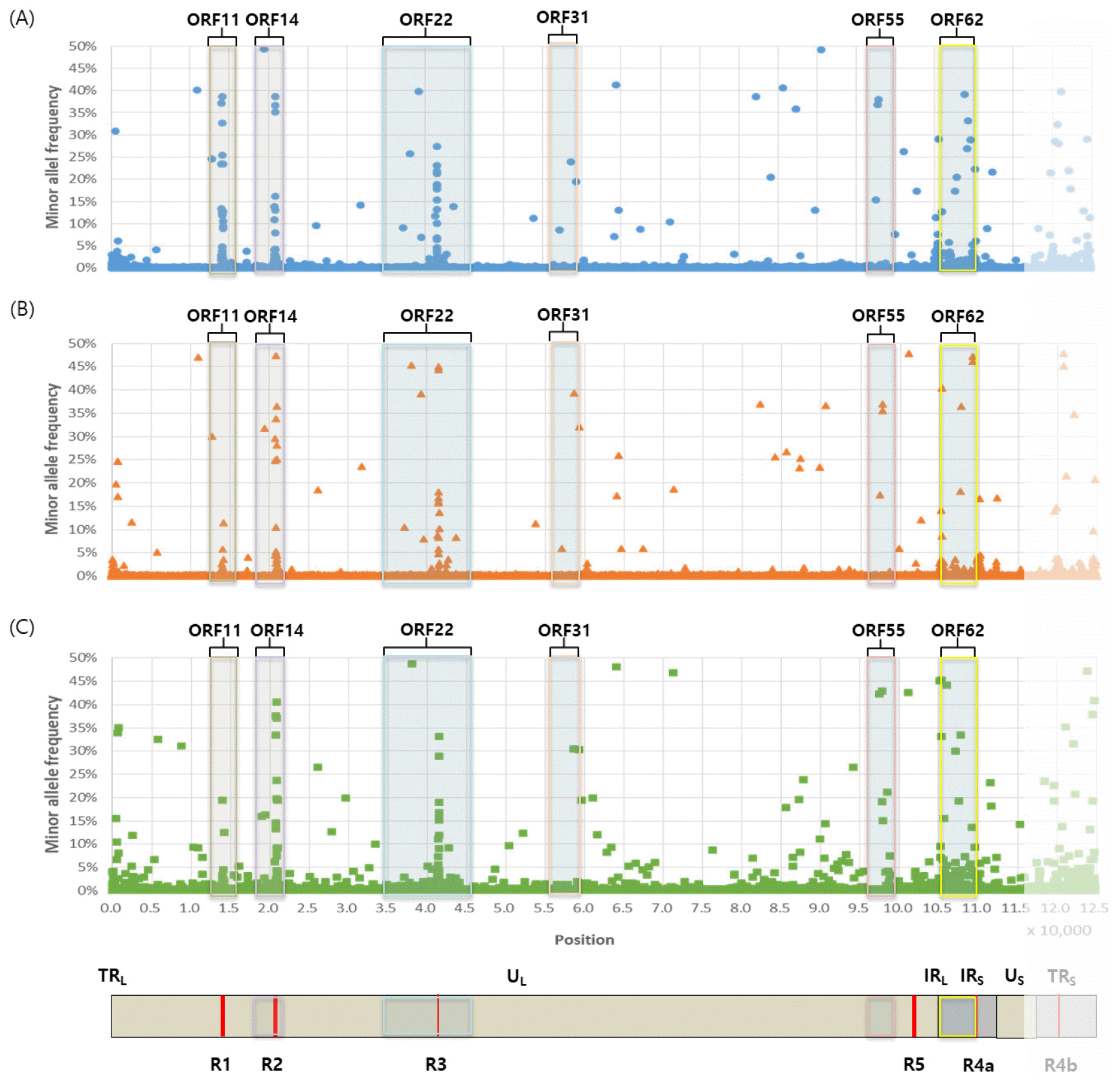 | Fig. 2Distribution of GPS on the genome of VZV vaccine strains. Nucleotide positions are shown in X axis, and the frequency of minor alleles at GPS is shown in Y axis. Genome structure of VZV is shown at the bottom of the Figure. (A), Suduvax; (B), Varilrix; (C), Varivax. |
Table 2.
The number of GPS and mean MAF at GPS found in the three VZV vaccine strains
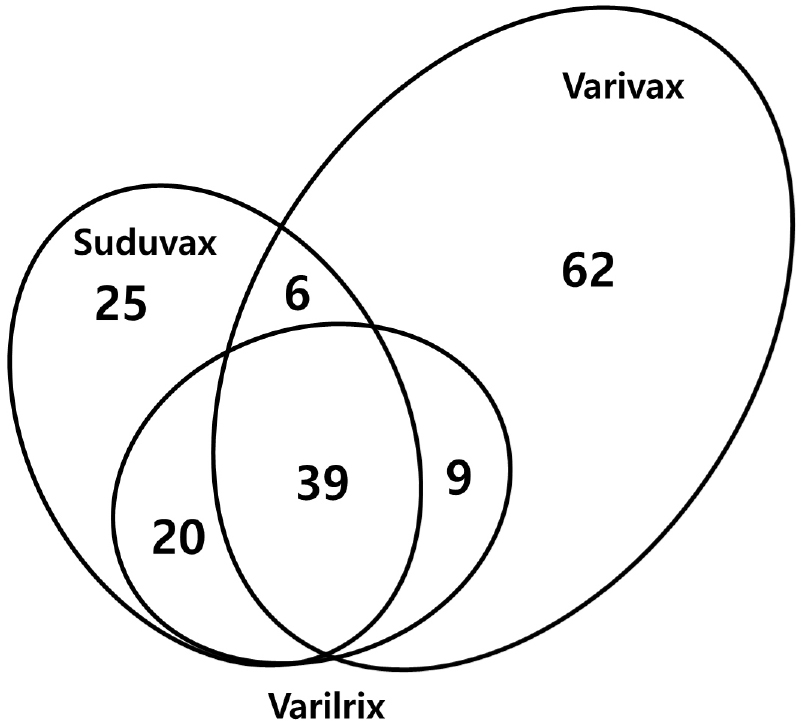
세 백신주간의 GPS 분포를 살펴 보았다. 세 백신주 중 어느 하나에서라도 관찰되는 GPS의 수는 모두 161 개였다(Supplemented Table 2). 이 중 세 백신주 모두에서 관찰되는 GPS는 39 개였고, 두 백신주에서는 공통으로 관찰되지만 세번째 백신주에서는 관찰되지 않는 GPS의 수는 Fig. 3에 나타낸 바와 같다. 또한 Suduvax에서만 관찰되는 GPS는 25 개, Varivax에서만 관찰되는 GPS는 62 개인 반면, Varilrix에서만 관찰되는 GPS는 없는 것으로 나타났다(Fig. 3). 이 중 세 백신주에서 공통으로 나타나는 GPS 39 개에 대해서 좀 더 자세히 살펴 보았다. 세 백신주 모두에서 적어도 하나 이상의 GPS가 관찰되는 ORF는 모두 20 개인데(Supplemented Table 2), 이 중 16개의 ORF와 하나의 NCR에서 세 백신주 공통인 GPS가 관찰되었다. 특히 ORF22에서 9개, ORF14에서 8개로 많은 공통 GPS가 관찰되었다. 공통 GPS에서의 평균 MAF는 세 백신주에서 23.1% (Suduvax) ~ 26.5% (Varilrix)로 전체 GPS의 평균 MAF보다 높았다. 세 백신주 공통인 39 개 GPS 중 17 개가 SNP에도 해당되었다.
Go to : 
고찰
현재 수두를 예방하기 위해서 전 세계적으로 사용되는 백신은 모두 약독화 생백신이다. 수두백신은 여러 계대배양을 거쳐 약독화 되었으며 (5), (8), 약독화 과정에 대한 명확한 메커니즘은 아직 확인되지 않은 상태이다. VZV는 서열 간의 변이가 적어 유전학적 보존성이 크기 때문에, 변이의 분석이 용이하다. 조금씩 발생하는 변이들은 선택압에 의해 축적되어 유전적 다양성이 증가하게 된다. 본 연구에서는 NGS 기법을 사용하여 대표적인 수두 백신주들을 SNP 분석과 유전적 다형성 분석을 통해 약독화에 따른 각 백신주가 어떠한 유전적 특징과 다양성을 가지는지 분석하고자 하였다.
세 백신주에서 공통적으로 SNP와 GPS가 많이 관찰되어 유전적으로 다양성이 높은 지역은 ORF 11, 14, 그리고 22로 나타났다. ORF11은 RNA에 결합하는 단백질을 만들며, glycoprotein E와 IE62 단백질의 발현에 어느 정도 기여하기 때문에, in vivo상에서 병원성에 영향을 미치게 된다 (18). ORF14는 glycoprotein C를 만들며, 이 단백질은 숙주세포 안으로 바이러스가 진입하는데 필요한 transmembrane protein을 만든다. 이 유전자는 바이러스 증식에 꼭 필요한 유전자는 아니지만, 계대가 진행된 바이러스 주에서는 야생형 바이러스 보다 glycoprotein C의 발현율이 낮다고 알려져 있다 (19), (20). ORF22는 바이러스 증식에 필수적인 유전자로 허피스바이러스에 잘 보존되어 있으며, VZV 단백질 중 가장 큰 inner tegument protein을 암호화한다. 이 단백질은 숙주세포 내의 CRLs (Cullin-RING ubiquitin ligases)을 분해시켜 숙주세포의 S기 진입을 막아 바이러스의 증식이 효과적으로 이루어지게 만든다 (21). 이들 유전자의 SNP와 GPS에 의한 유전적 다양성은 백신주들의 약독화와 어느 정도 관계가 있는 것으로 생각되지만, 아직 직접적인 연관성에 관한 연구 결과는 없으므로 향후 연구가 필요한 부분이라고 본다.
ORF 11, 14, 22의 유전적 다양성은 대부분이 각 ORF에 존재하는 반복 서열인 R 지역(R1, R2, R3)에 의해 나타나고 있다. R 지역은 특정한 길이를 가지는 서열들이 반복되어 있고, 그 반복 개수는 VZV주에 따라 다양하다 (22). R1은 여러 개의 18 bp 단위와 15 bp 단위의 조합에 의해 이루어져 있다. Suduvax는 Oka-유래 백신주들에 비해 15 bp 단위 하나를 더 가지고 있다. R2는 42 bp 단위 여러 개와 42 bp의 일부분인 32 bp 단위 하나로 구성되어 있다. R3는 ORF22의 3’ 말단에 존재하며, 여러 개의 9 bp 단위로 구성되어 있다.
NCR 중에서는 ORF 62와 63 사이에 위치하는 NCR62/63이 유전적으로 가장 다양한 것으로 나타났다. NCR62/63에는 반복 서열인 R4지역과 ORI 지역이 있다. R4 지역은 여러 개의 27 bp 단위와 그 일부분이 11 bp 단위로 구성되어 있다. Suduvax는 Oka-유래 백신주들에 비해 54 bp의 결실이 있는데 이는 27 bp 단위 두 개가 결실된 것이다. ORI 지역은 DNA 복제가 시작되는 지역으로, TA 서열이 여러 개 반복되어 있다. ORI 지역의 TA 반복 횟수를 임상주인 pOka와 비교해 보았을 때, vOka 유래 백신주(Varilrix, Varivax)에서 더 적게 나타났다. 이러한 TA 반복 지역이 짧으면 게놈 복제 효율이 낮아진다는 연구 결과를 감안하면 (23), ORI 지역에서의 염기 TA의 반복성이 백신주간 달라 vOka 유래 백신주의 게놈 복제 효율이 낮을 것으로 추측한다.
R 지역을 포함하는 ORF의 유전적 다양성이 높은 것은 앞에서 설명한 바 있다. 이들 ORF11, 14, 22 이외에도 ORF31, ORF55와 ORF62 지역에서 비교적 높은 유전적 다양성이 관찰되었다. ORF31은 VZV가 증식하기 위한 필수적인 유전자로, glycoprotein B를 암호화한다. Glycoprotein B는 virion envelope 표면에 형성되며, 숙주 세포의 receptor와 결합하여 세포 내로 진입을 위해 세포막과 융합에 관여한다 (24), (25). ORF55는 VZV가 증식하기 위한 필수적인 유전자로 알려져 있다. Helicase/primase 복합체를 만들며, replication fork에서 DNA를 풀어 단일 가닥 DNA를 만들고, leading strand와 lagging strand에서 DNA 합성이 일어나게 한다. ORF62는 VZV가 증식하기 위한 필수적인 유전자로 알려져 있으며, 초기 유전자의 trans-activating factor에 관여한다. ORF62 지역은 많은 돌연변이가 발생하며, 바이러스의 증식을 억제시켜 VZV의 약독화에 가장 중요한 ORF로 알려져 있다 (3), (11), (26).
세 백신주 간의 SNP 중 동의적 치환이 일어난 8 군데는 해당 유전자의 기능에 영향을 주지 않을 것으로 생각된다. 비동의적 치환 17 군데 중에서 9 군데는 같은 그룹의 아미노산으로 바뀌기 때문에 단백질의 구조와 기능에는 큰 영향을 미치지 않을 것으로 생각된다. 그러나 나머지 8 군데에서는 다른 그룹의 아미노산으로 치환되므로 단백질의 구조와 기능에 영향을 줄 것으로 생각된다. 예를 들어 Asn/Lys 치환이 모두 세 군데(Dumas 위치 20745, 20787, 20871)에서 관찰되었는데, 이 세 군데는 모두 ORF14에 존재하므로 세 백신주 간의 유전적 다양성에 크게 관여할 것으로 생각된다. ORF55에서는 Ala/Thr과 Arg/Cys의 두 치환이 관찰되었으며, ORF59에서 Arg/Gln 치환이, ORF62에서 Met/Thr 치환이, 그리고 ORF64에서는 Arg/Gln 치환이 관찰되었다. 이와 같은 다른 그룹 아미노산으로의 치환이 백신주에 어떤 영향을 미칠지에 대해서는 아직 알려진 바가 없으나, 세 백신주의 효율성과 돌파감염의 차이에 어느정도 기여할 것으로 생각된다 (10), (11), (27).
Go to : 




 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download