INTRODUCTION
2019년 12월 중국 우한에서 시작된 원인미상의 중증 폐렴이 SARS-CoV-2 (severe acute respiratory syndrome coronavirus 2)에 의한 것이라고 밝혀진 이후, 3월 초에 세계보건기구가 판데믹을 선언하였고 6월까지 전세계 200여개 국가에서 800만명 이상의 감염자와 45만여명에 이르는 사망자가 발생되었다 (1). 우리나라에서도 6월까지 약 12,000여명의 감염사례가 확인되었고 이들 중280여명이 사망(치사율 2.31%) 하였으며, 최근에는 지역사회 감염으로 추정되는 감염사례가 지속적으로 발생되고 있다 (2). 새롭게 나타난 감염병을 제어하는 최선의 방법은 질병 발생 이전에 적절한 예방이지만 아직 인류는 SARS-CoV-2 감염 억제를 위한 몇 가지 의미 있는 치료제나 백신 후보물질의 안전성 및 효과를 검토하는 기초단계에 머물러 있는 실정이다 (3), (4). 따라서 지금까지의 SARS-CoV-2의 감염확산 억제 방법은 정확한 병원체 진단으로 감염자를 신속하게 파악한 후 의료적 처치 및 격리 등의 방역정책과 추가 감염을 막기위한 사회적 거리두기가 활용되고 있다.
SARS-CoV-2를 포함하여 일반적으로 신종 감염병을 대응하기 위한 전략은 크게 세가지로 나눌 수 있다. 첫째, 감염병 원인체를 파악하는 인지 전략인데 새로운 병원체는 대부분 과학지식이 축적되어 있지 않은 상태에서 유행이 시작된다. 따라서 해당 신종병원체를 신속하게 확보하고 유전적, 생화학적 특성을 분석하여 이를 임상적으로 연결한 기술이 진단 기법이며, 초기 임상특성에 부합하는 민감도, 특이도가 확보된 진단법의 적용 여부가 공중보건학적 대응 전략 수행에 중요하게 작용한다 (5), (6), (7). 둘째는 해당 감염병의 원인병원체를 직접 중화하거나 숙주의 임상증상을 완화시키는 약물중재방법, 즉 치료기술이다 (8), (9). 신종감염병 발생 초기에는 바이러스성 병원체의 유입으로 진행되는 일련의 병리학적 증상을 완화하는 대증요법을 사용할 수밖에 없으나, 병원체의 특성 분석을 통한 지식정보가 확보된다면, 특이적으로 작용하는 승인약물 또는 승인이 필요한 신약 치료기술을 적용함으로써 질병중재를 시도할 수 있다. 마지막으로 세번째는 해당병원체에 노출된 환자들의 임상경과, 병원체-숙주의 면역학적 반응 특성 등을 종합하여 동일한 병원체의 노출로부터 인간을 보호하기 위한 예방 기술이며, 대표적으로는 백신을 들 수 있다 (10), (11). 인류집단에 처음 등장하는 신종 감염병은 숙주의 면역방어체계가 미처 구축되기 전에 감염을 통하여 체내로 침입하게 된다. 만일 이용가능 하다면, 이미 알려진 감염병 백신과 마찬가지로 숙주에게 면역기능을 부여하는 예방적 백신투여가 새로운 감염병을 방어하는 전략 중에서 가장 효과적인 방법이다.
본 논고에서는 신종감염병 발생 초기의 대응전략으로 매우 중요한 원인 병원체 인지역량 강화 전략에 대한 최신의 과학기술 개발 현황을 고찰하고, 보다 진보된 수단으로써 감염병 발생을 탐지할 수 있는 과학지식의 활용 의견을 제시하고자 한다.
Go to : 
세포배양을 통한 병원체 확보
최근 인류집단에서 발생되는 대유행 또는 지역적인 대발생의 원인 병원체는 02-03년 SARS-CoV (severe acute respiratory syndrome coronavirus), 09년 신종인플루엔자, 12년 MERS-CoV (middle east respiratory syndrome coronavirus) 및 19년 SARS-CoV-2 (severe acute respiratory syndrome coronavirus 2) 등 지금까지 알려지지 않았던 새로운 바이러스에 의한 현상이 대부분이다 (12), (13), (14). 이러한 신종 바이러스가 인류집단에 처음 도입되었을 때 가장 중요한 것은 이 병원체가 무엇인지를 파악하는 인지 역량이다. 병원체의 실체를 파악하기 위해서는 무엇보다도 이 신종 병원체가 가진 생물학적 특성을 신속하게 규명하는 것이 가장 중요하다. 병원체가 사람의 체내에 침투하여 기관이나 장기에 감염된 이후에는 발열, 호흡기 증상, 소화기 증상 등 임상적으로 여러가지 증상이 나타날 수 있는데 이렇게 신종 바이러스에 감염된 환자의 임상검체를 대량으로 확보하고 바이러스를 직접 분리해서 이의 특성을 분석하기에는 과학적, 윤리적 문제가 뒤따를 수 있다. 그러므로 적절한 실험실내 복제기구, 예를 들어 세포배양이나 실험 동물 감염 system을 통한 병원체의 확보가 선행되어야 해당 병원체의 생물학적, 유전학적, 그리고 면역학적 특성분석을 통한 인지 역량을 획득할 수 있다 (15).
세포배양 기술의 발달로 다양한 바이러스가 실험실 내에서 복제될 수 있는 기술적 진보가 최근에 많이 이루어지고 있지만 여전히 신종 바이러스를 세포배양법을 통하여 성공적으로 증폭한다는 것은 그리 쉬운 일만은 아니다. 신종바이러스의 숙주 침투경로에 따라 호흡기계 세포조직, 소화기계 세포조직 등 다양한 기원의 세포들이 바이러스를 분리하는 세포배양법에 사용되고 있다. 그러나 실험실내 세포배양법은 대부분 2차원적인 단층배양세포를 활용하게 되며, 사용되는 세포의 종류들을 보면 숙주에서 직접 분리한 초대 배양세포(primary culture cell)가 일부 있기는 하나 대부분은 불멸화된 세포주(immortalized cell line)을 사용하게 된다 (15), (16). 이들 세포주가 신종 바이러스 복제에 성공적으로 활용될 수 있는 결정적인 기여는 대부분 세포주가 가지고 있는 수용체를 신종바이러스가 어떻게 인식하고 세포내로의 침투에 활용하는가에 달려있다 (Fig. 1). 안타깝게도 실험실내 세포주가 가지는 수용체의 생물학적 특성과 3차원 구조로 이루어진 숙주 내 감수성 세포조직의 특성이 다를 수 있기 때문에 신종 바이러스의 분리에 선택된 세포주가 적절한 수용체를 가지고 있을지라도 결과적인 감염 효율은 커다란 차이를 보이기도 한다. 이러한 상황에서 보다 숙주내의 조직 특이성에 가깝게 유지될 수 있도록 특이 수용체, 또는 바이러스의 세포 침입 과정에 관련된 다양한 보조 단백질이 부가적으로 발현되도록 유전학적으로 조작된 세포주를 제작하여 신종바이러스 분리에 활용하기도 한다 (17), (18), (19).
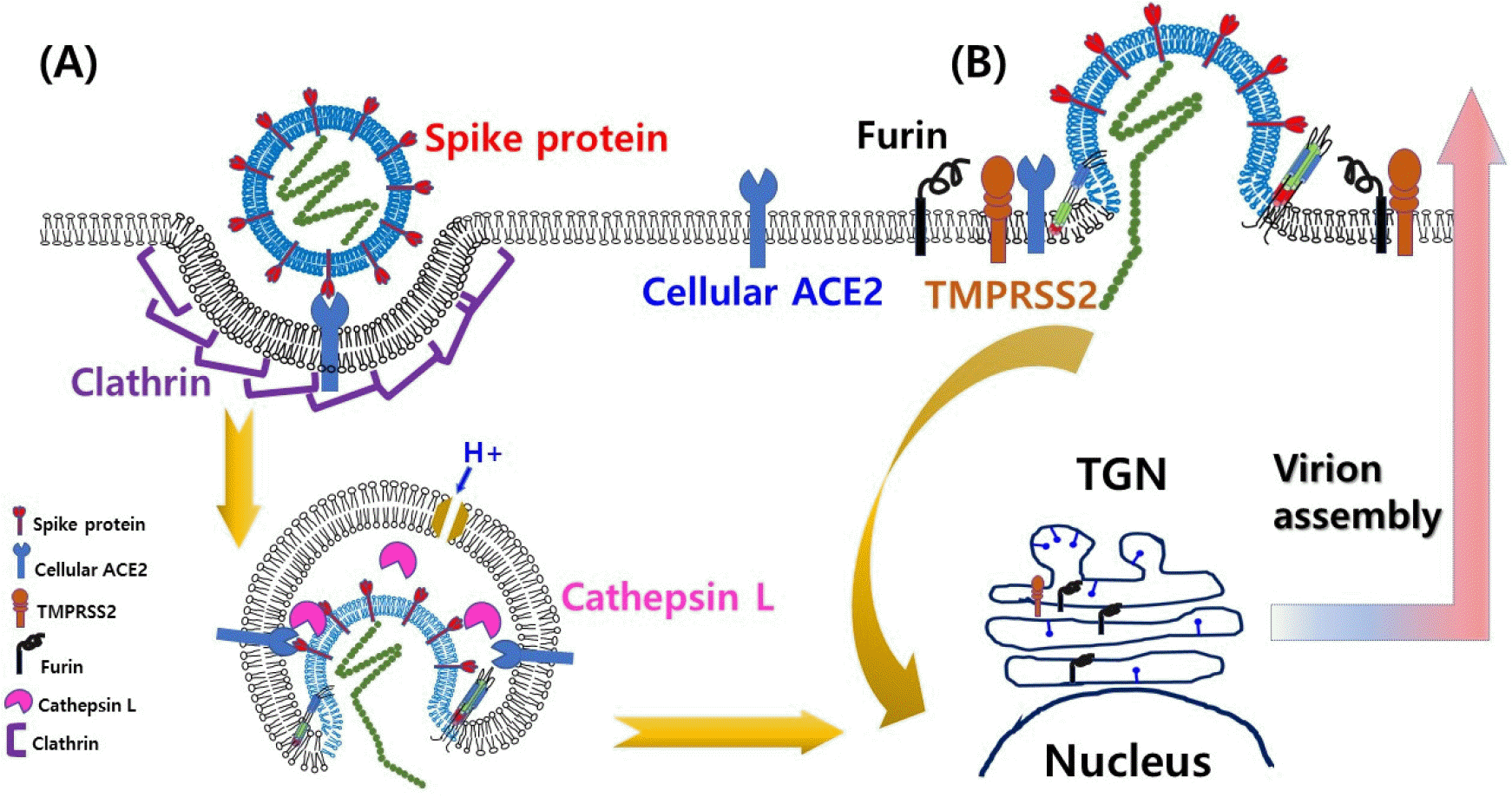 | Fig. 1Schematic diagram of postulated SARS-CoV-2 cell entry through endocytosis and direct membrane fusion. The SARS-CoV-2 preferentially utilizes ACE2 (angiotensin converting enzyme 2) as a cellular receptor for recognition of susceptible cells. Once cellular receptor interacts with trimerized viral spike proteins, multifactorial entry machineries including conformational alteration of spike proteins at the cell membrane involving endocytic pathways (A) and/or unique trans-membranous protease activities (B) are processed. |
병원체 유전자 정보의 확보
세포배양에 의한 바이러스복제 성공률이 그다지 높지 않은 것과는 대조적으로 신종바이러스의 유전자를 분석하고 그 특성을 분석하는 것은 상대적으로 빠르게 진행될 수 있다 (20). 최근에 각광받고 있는 deep-sequencing기법 등 차세대 염기서열 분석법(NGS; next generation sequencing)을 이용하면 세포배양 기법보다 상대적으로 신속하게 바이러스 유전자의 특성정보를 확보할 수 있다 (21), (22). 그러한 이유는 아무리 다양한 기원에서 출발한 신종 바이러스일지라도 그 병원체가 가지는 유전물질이 DNA 또는 RNA(혹은 혼합된 경우) 이기 때문에 이들을 확인하는 최신의 분자생물학적 기법을 적용하면, 의외로 빠른 시간에 신종 바이러스의 유전자 특성이 분석될 수 있는 것이다. 또한 생명과학을 연구하는 학자들은 이미 바이러스가 가지고 있는 유전물질 보호막(외피 단백질, 또는 바이러스 외막 등)을 매우 효과적으로 제거할 수 있는 기술이 있다 (23), (24). 과거에는 페놀 등 독성 유기용매로 단백질과 인지질 성분이 대부분인 바이러스 외피(외막)를 제거했지만 최근에는 아주 간단한 kit(예를 들면 viral RNA/DNA mini kit 등)를 사용하여 안전하게 바이러스가 많이 존재하리라고 여겨지는 적절한 감염된 환자의 검체로부터 바이러스 유전자를 획득할 수 있다.
SARS-CoV-2의 경우 2019년 중국 우한지방에서 원인을 알 수 없는 폐렴 발생으로 시작되어 2020년 1월 9일 중국 보건당국과 세계보건기구가 신종 코로나 바이러스의 존재를 알렸고 이틀 후 해당 병원체 감염에 의한 공식적인 세계 최초의 환자로부터 직접 확보된 전장유전체 정보가 국제사회에 공개되었다 (25). 새로운 병원체의 출현과 관계된 일련의 과정에서 신속하게 신종 바이러스의 전장 유전체 정보가 이처럼 신속하게 공개되었다는 것은 해당 감염원을 인지하는 분자진단 역량 확보에 매우 중요한 일로 평가될 수 있다. 그 이후 세계 각국에서 확인된 환자, 또는 환자 검체를 감수성 세포에 접종하여 얻어진 바이러스 배양체로부터 속속 전장유전체 정보가 공개되었고, WHO에서 운영하는 GISAID (Global Initiative on Sharing All Influenza Data)를 통하여 공유되기 시작하였다 (26), (27). 우리나라에서도 2월 초 아프리카 녹색원숭이 신장세포(Vero)에서 배양된 바이러스의 전장유전체 정보의 공유가 시작되었으며, 그 이후 세포배양 바이러스가 아닌 환자 검체로부터 직접 전장유전체 정보를 획득하여 공유함으로써 2020년 5월까지 전세계적으로 25,000여건이 넘는 바이러스 전장유전체가 전문가 집단에 공유되고 있다 (28).
바이러스 전장유전체가 이렇게 신속하게 공유된다는 것은 분자 진단기술 확보에 매우 중요한 의미를 가진다. 감염병의 원인 바이러스를 진단하는 방법 중에 가장 확실한 방법은 두말할 나위없이 증상을 보이는 환자로부터 채취된 검체 병원체의 존재를 직접 확인 하는 것이다. 일반적으로 세균이나 기생충 같은 비교적 크기가 큰 병원체는 광학 현미경 또는 고배율 현미경 관찰을 통하여 환자의 검체나 조직 내에서 병원체를 형태학적으로 관찰할 수 있다. 그러나 바이러스는 그 크기가 나노메터 수준으로 광학현미경으로는 형태 분석이 불가능하며, 중금속 염색 등 복잡한 전처리 과정이 필요한 전자현미경으로 확대배율을 10,000배에서 100,000배 이상으로 설정하여야 관찰이 가능하다 (29). 따라서 가장 확실하지만 효율과 기술적 측면에서 전자현미경 관찰 기법을 바이러스 진단에 상시적으로 활용하기는 어렵다. 신종 바이러스에 의한 감염병의 대발생이나 대유행 단계에서는 실험실 진단의 정확성과 아울러 효율성이 강조된다. 그러므로 평상시 숙주세포(환자)가 가지고 있지 않은 바이러스 유전자를 증폭하여(RNA 바이러스의 경우 cDNA를 합성한 후 증폭) 이의 염기서열 정보를 확보하여 분석하는 분자진단 기법이 실질적으로 활용되는 것이다 (30), (31), (32).
Go to : 
유전자 증폭을 통한 병원체 탐지기술
1980년대 말 인위적인 유전자 증폭방법으로 중합효소 연쇄반응법(PCR; polymerase chain reaction)이 개발된 이후 PCR 기법은 다양한 생명과학 연구에 활용되었다 (33), (34) (35). 특정 병원체의 유전자를 증폭하기 위하여 PCR 기법을 적용할 때 가장 중요한 것이 알려진 염기서열 정보로부터 제작된 primer의 선택이다. 고전적인 PCR에서 사용되는 DNA 중합효소(일반적으로 Thermophilus aquaticus 라는 고온적응 미생물에서 분리된 중합효소, Taq polymerase)는 DNA 중합 반응의 시작을 위하여 목적 유전자 이중가닥 DNA에 각각 상보적으로 결합하는 primer 두가지(forward 및 reverse)가 필요하다. 따라서 증폭하고자 하는 유전자의 정보를 사전에 확보하고 있어야 하며, 이들 두개의 primer가 결합하는 부위는 가급적 유전자 변이가 없는 염기서열이 잘 보존된 영역(highly conserved)이어야 한다는 제한점이 있다. 그러므로 신종바이러스 감염사례에서 환자의 검체로부터 해당 병원체의 유전자 검출을 위해서는 유전정보의 확보 및 분석이 선행되어야 하며, PCR 법의 이러한 유전정보 의존적인 특징이 높은 민감도를 가진 PCR진단 기능의 한계점으로 종종 작용하기도 한다 (36), (37). PCR 진단결과의 판독은 전기영동을 통하여 증폭된 유전자의 크기와 양을 표준물질과 비교하는데 이때 적절한 발색 시약을 사용하여 증폭 유무를 시각적으로 확인하는 정성판독을 하거나 발색의 강도를 수치화 하는 검출기기를 통해서 간접적인 정량분석도 가능하다. 두가지 경우 모두 유사한 크기의 전혀 다른 유전자(주로 숙주유전자 또는 정상세균총에서 유래된 유전자)의 비특이적 증폭이나, 강력한 유전자 증폭기능을 가진 PCR과정에서의 오염으로 인한 위양성 결과가 나타날 수 있음을 항상 염두에 두어야 한다. 직전 PCR 과정에서 이미 증폭된 유전자에 의한 오염을 방지하기 위하여 dTTP 대신 dUTP와 uracil N-glycosylase (UNG)를 이용한 pre-hydrolysis 과정으로 carry-over 오염을 최소화하는 방법도 활용되고 있다 (38), (39).
분류학적으로 동일한 그룹에 속하는 여러 종, 또는 속의 바이러스들이 가지는 유전자 특성은 공통적인 경우가 있다. Primer 선택 과정에서 동일 계열에 속하는 유전자의 변이가 가장 적은 부위, 즉 가장 잘 보존된 유전자 영역을 찾을 수만 있다면 그룹내 여러 바이러스들의 유전자를 한가지 PCR 기법을 적용함으로써 증폭할 수도 있다. 이때 활용되는 primer를 특별히 범용 primer라고 한다 (40). 실제 primer를 고안함에 있어 동일 그룹내 바이러스유전자의 동질성이 100%가 되지 않고 일부 염기에 변이가 관찰되더라도 inosine 등을 활용한 degenerated primer를 고안하고 이를 활용하면 유전적으로 유사한 그룹 내의 여러가지 바이러스 유전자를 한번의 반응으로 증폭하고 확인할 수 있다. 그렇지만 최종적으로 바이러스를 특정하기 위해서는 증폭산물의 염기서열 분석이 필요한 경우가 대부분이다. 이번 SARS-CoV-2 발생 초기에는 기존의 사람코로나 바이러스, 즉 HCoV (human coronavirus)-229E, OC43, NL63, HKU1 및 MERS-CoV와 SARS-CoV 유전자를 모두 증폭할 수 있는 pan-coronavirus PCR법으로 초기 진단을 실시하였으며, 이때에도 최종 증폭 산물을 대상으로 추가적인 염기서열 분석을 실시하여 실제 어떤 바이러스 유전자인지를 확인하는 과정을 거쳤다 (41).
비교적 최근에는 PCR 및 염기서열 분석에 소요되는 반응시간 단축을 위하여 실시간으로 유전자를 증폭하고 이를 정량적으로 검증할 수 있는 실시간 유전자 정량 증폭법(quantitative real-time PCR; qRT-PCR)이 개발되어 다양한 병원체의 분자진단에 사용되고 있다 (42), (43). qRT-PCR 기술 개발 초기에는 분자 비이콘, 사이버그린 등 다양한 방법의 증폭산물 signaling 기법이 소개되었으나 최근에는 probe 및 primer 고안이 수월하여 바이러스 유전자 변이 또는 새로운 바이러스 검출에 쉽게 적용할 수 있는 Taqman based qRT-PCR법이 임상적으로 적용할 수 있는 바이러스 유전자 진단에 널리 사용되고 있다 (44), (45), (46). qRT-PCR 기술은 증폭을 목표로 하는 유전자에 상보적인 primer외에 목표 유전자(대부분 100-150개의 염기)내에 또다른 상보적 염기서열을 가진 probe가 추가된다. 이 probe의 3’ 및 5’ 말단에는 각각 발광물질과 그 물질의 발광상태를 억제하는 물질(quencher)이 연결된다. 임상 검체내에 증폭하고자 하는 바이러스 유전자가 있을 경우 primer 부착시 형광물질을 가진 probe도 동일하게 부착되고 대상 유전자가 중합효소에 의해 새로운 DNA가 한번씩 합성될 때 마다 DNA 중합효소가 가진 exonuclease 기능에 의하여 probe가 분해됨으로써 quencher에 의해 억제되어 있던 형광물질이 발광된다. qRT-PCR 장비는 DNA 합성 주기마다 probe에서 발광되는 형광의 증가폭을 측정하여 일정한 수치 및 그래프로 나타내 주며 이에 따라 대상 유전자의 존재유무는 물론 증폭되는 DNA의 양을 동시에 확인할 수 있다. 현재 우리나라 질병관리본부 및 여러 의료기관에서 SARS-CoV-2의 진단을 위해 사용하고 있는 qRT-PCR 기술은 이를 바탕으로 제작되고 검증된 방법으로 제조사 및 제조방법에 따라 약간 차이는 있지만 전반적으로 qRT-PCR 진단 체계의 민감도는 대략 10-100개 이상의 바이러스 유전자가 있을 경우 이를 인식할 수 있는 정도로 민감도가 높다 (42), (43). SARS-CoV-2의 경우 바이러스 유전물질이 RNA이므로 qRT-PCR 기술을 적용하기 위한 전단계로 바이러스 RNA로 부터 cDNA를 합성하는 역전사과정이 추가로 필요하며, 이는 qRT-PCR 반응 초기에 역전사 효소를 추가로 사용하여 진행된다.
지금까지는 qRT-PCR 기술의 기본적인 사항을 살펴보았는데 실제 SARS-CoV-2 와 같은 신종 감염병 유행 초기에 임상역학 전략인 봉쇄(containment)를 수행하기 위해서는 위음성의 확률을 최소화하는 진단기법의 도입이 필요하다. 따라서 qRT-PCR 기술 개발시 바이러스 전체 유전자의 한 부분만을 대상으로 하는 것이 아니라 2-3개 흩어진 영역의 유전자를 대상으로 다중 qRT-PCR 기술을 적용하기도 한다. 이번 SARS-CoV-2의 qRT-PCR 기술에도 이러한 개념이 적용되어 RNA 중합효소 유전자(RdRP) 영역 및/또는 하위 유전자 영역으로 E 유전자, N 유전자 등 두가지 이상 유전자 영역을 증폭하는 다중 분자진단 기술이 이용되고 있다(Table 1) (47). 이와 같이 qRT-PCR 기술은 유전자 정보가 확보된다는 전제가 필요하지만 SARS-CoV-2 와 같이 새롭게 출현한 바이러스의 봉쇄정책을 위한 위음성을 최소화할 수 있도록 고안되어 신종 바이러스 감염에 의한 환자의 실험실적 진단에 활용된다.
Table 1.
Currently licensed molecular diagnostic kits for the COVID-19 in Korea (47)
Go to : 
qRT-PCR 진단 기술의 보완점
cDNA를 합성하는 역전사 과정과 qRT-PCR 기술은 현재까지 개발된 분자생물학적 진단법 중에서 최상의 민감도와 특이도를 확보할 수 있는 유일한 방법이다. 그러나 분자진단을 목적으로 하는 qRT-PCR 기술이 가지는 한계점도 분명히 존재한다. 첫째로, cDNA 합성을 포함한 qRT-PCR을 수행하기 위해서는 비교적 순수한 상태의 RNA 또는 DNA가 필요하다. 이는 객담, 인후가검물 등 다양한 호흡기 검체나 분변 같은 소화기 검체로부터 얼마나 순수하게 바이러스 유전자를 추출해 내는가에 좌우된다. 이 과정에는 객담 같은 점성의 검체를 적절한 시약으로 처리하여 바이러스 유전자를 가급적 안정적으로 추출할 수 있는 기술의 개발이 동시에 요구되며, 분변검체와 같은 특별한 경우 qRT-PCR 기술에 사용되는 중합효소들의 기능을 억제하는 방해물질이 존재할 가능성을 검체 처리 단계에서 제거하기 위한 추가 고려사항이 필요하다. 즉, 순수한 바이러스 유전자를 얻기 위한 검체의 전처리 단계가 qRT-PCR 결과를 크게 좌우한다고 할 수 있다.
두번째로, 경우에 따라서는 가급적 많은 유전자 영역을 증폭하는 다중 qRT-PCR 기술이 필요한데 현재까지 개발된 qRT-PCR에 사용되는 발광화학은 probe에 표지하는 형광물질의 파장을 달리하여 이론적으로는 최대 6개의 서로 다른 probe가 사용 가능하다. 실제로는 서로 다른 형광 probe 를 동시에 사용할 경우 형광물질들이 가지는 고유의 파장 간섭현상으로 이상적인 민감도를 확보하기가 불가능하다 (48), (49). 따라서 파장간섭이 없는 다양한 발광 스펙트럼의 표식 및 탐지 기작을 적용한 고집적 qRT-PCR (HD-qRT-PCR; highly divergent qRT-PCR) 기술이 개발된다면, 유전자 증폭을 통한 다중 분자진단에 획기적 기여가 가능할 것으로 생각된다.
세번째, qRT-PCR법은 보편성, 민감도 및 특이도 등 여러가지 장점이 있음에도 불구하고 주요한 선결조건이 있다. 즉, qRT-PCR 개발은 인지하고자 하는 대상 바이러스의 유전정보에 전적으로 의존하여야 한다는 것이다. 이 조건은 때로는 신종 바이러스성 질환이 등장할 때나 기존의 바이러스가 변이를 일으켰을 경우 qRT-PCR 활용에 걸림돌로 작용할 가능성이 있어 GISAID에서는 주기적으로 SARS-CoV-2 유전자 증폭에 사용되는 primer 및 probe 변이 분석 정보를 제공하고 있다(Table 2).
Table 2.
Mutation ratio of primers and/or probe sites in target genes for qRT-PCR kits (GISAID)
새로이 등장한 바이러스의 유전자 정보가 충분치 않거나 심지어 아예 없는 경우에는 유전정보 비의존적 분자생물학 기법인 전장유전체 분석(WGS; whole genome sequencing)이 사용되기도 한다 (50). 기술적 측면 이외에 qRT-PCR법이 가지는 또다른 한계점으로 증폭결과 분석을 위해서 첨단 검출장비가 필요하다는 것이다. 따라서 qRT-PCR법은 분석을 위한 연구장비가 잘 갖춰진 전문 실험실에서만 가능하다는 단점이 있다. 최근에 qRT-PCR법의 현장 적용을 위한 간편한 POC (point of care) 장비들이 개발되고 있지만 아직 전문 분석기기에 견주어 동등 또는 그 이상의 민감도와 특이도가 확보되면서, 현장에서 널리 사용될 수 있는 소형 장비는 없다.
고가 장비 접근성의 문제점을 해결하면서 qRT-PCR법을 사용할 경우와 유사한 효능을 나타낼 수 있는 유전자 분석방법이 개발되었는데 이를 등온 중합효소연쇄 반응법(LAMP; loop-mediated isothermal amplification)이라고 한다 (51), (52), (53). qRT-PCR법과 비교하여 민감도 및 특이도가 유사하다는 가정하에 LAMP가 가지는 장점은 짧은 시간내(주로1시간 이내)에 한가지의 특정온도 만으로 유전자를 증폭할 수 있고, 여섯개의 priming 유전자를 사용하므로 상대적으로 높은 특이도를 기대할 수 있으며, qRT-PCR과는 달리 고가의 장비가 필요하지 않아 현장 사용이 가능하다. 그러나 LAMP가 가지는 단점-극복 가능하리라 생각되지만-도 있는데 첫째, 6가지 primer의 고안이 qRT-PCR법에 사용되는 2 primers, 1 probe 보다 복잡하다는 것이다. 또한 qRT-PCR법에서 3가지 정도의 유전자를 다중으로 증폭할 수 있지만 LAMP 에서는 이론적으로 힘들다. 마지막으로 qRT-PCR법에 사용되는 주요 reagent의 지적 재산권은 이미 공개되어 있어서 누구나 자유롭게 활용가능 하지만, LAMP법을 응용한 최신 기술에는 지적재산권이 남아 있는 경우가 많아 자유로운 활용에 장애물이 되고 있다. 그럼에도 불구하고 LAMP법이 가지는 신속성, 간편성은 현장 신속진단법으로 활용되기에 충분한 매력을 가지고 있다. 이러한 LAMP법의 편리성과 유전자 가위로 알려진 CRISPR-Cas12의 유전자 편집 기술을 접목하여 바이러스 유전자를 검출하는 정성분석 기법이 소개되기도 하였다 (54), (55). 이 방법은 기존의 qRT-PCR법 보다 민감도면에서 약간 성능이 낮지만 lateral flow 기술을 응용하여 고가의 장비 없이 현장에서 직접 유전자 분석결과를 확보하는 것이 가능하다 (56).
Go to : 
전장유전체 분석기법의 활용
최근의 차세대 염기서열 분석 기법(NGS; next generation sequencing)의 발달은 유전체 정보가 불명확한 신종 바이러스질환의 출현을 인지하기 위한 다양한 기술적 선택이 가능하게 해주고 있다 (50). DNA 신규합성법(de novo synthesis)을 이용한 deep-sequencing이나 검체 내의 RNA를 cDNA 로 전환하지 않고 직접 분석하는 RNA sequencing 법이 활용될 수 있다 (57). 이러한 NGS 기술을 응용한 WGS기법은 새로이 출현한-유전정보가 없어 적절한 primer/probe 적용이 곤란한- 병원체의 인지에 유용할 뿐 아니라 일정규모 이상의 전장 유전체 정보가 확보되면 임상역학적 자료와 병합하여 새로운 바이러스의 분자역학적 진화방향과 전파력에 대한 예측, 돌연변이 hot spot의 분석과 이의 분자병리학적 연관성 규명 등 신종 바이러스의 유용한 인지기술에 사용될 수 있다 (58), (59). 뿐만 아니라 정제된 대량의 WGS 정보는 숙주 수용체 또는 바이러스 항원과 숙주 항체의 결합특성 분석을 통한 백신 개발 전략 및 치료제 후보물질과의 구조적 상호작용 규명 등 바이러스 특성분석에도 확대 적용될 수 있다 (60), (61).
그러나 이 기술이 신종 바이러스성 감염병이 발생되었을 때 실질적으로 널리 활용되기 위해서는 몇 가지 해결해야 할 문제가 있다. 첫째, NGS를 위해서는 고품질의 cDNA 또는 RNA 가 필요하다. 특히 RNA 염기서열을 직접 분석하는 RNA sequencing의 경우에는 RNA 순도가 가장 중요하며, deep-sequencing을 위한 cDNA도 양과 질에 있어서 qRT-PCR의 경우보다 높은 품질이 요구된다. 게다가 RNA-sequencing의 경우 목표 유전자가 RNA이기 때문에 신종 바이러스의 유전물질이 DNA인 경우에는 감염 후 복제기작이 어느정도 진행되어 숙주세포내에 mRNA가 생성된 이후 이를 대상으로 할 수밖에 없어, real-world 유전체 정보 확보가 용이하지 않다. 또한 RNA 바이러스일지라도 감염 후 잠복기가 긴 경우, 즉 숙주에 감염된 이후 활발한 RNA 복제가 일정기간 지연된 후 진행된다면 RNA 유전정보 확보를 통한 병원체 인지 기능의 민감도가 낮아질 수 있다.
둘째, WGS 과정에서 생성되는 숙주 유전정보를 제거하고 원하는 바이러스 유전체 정보를 획득하기 위한 고차원의 생물정보학적인 분석 기법이 요구된다 (62). 종종 WGS 기초자료는 용량이 매우 방대하여 데스크톱 컴퓨터 정도의 사양으로 분석하기 곤란하며, 분석시간도 24시간 이상 수일이 소요되는 경우가 많다. 또한 고가의 WGS 기기 및 시약이 소요된다는 단점도 해결해야 할 과제이다. 이러한 단점, 즉 처리시간과 고가의 장비 등을 해결하기 위한 시도로 MinION (Oxford Nanopore)과 같이 작고 가벼우며 현장으로 쉽게 운반할 수 있는 장치가 개발되어 일부 사용되고 있지만 qRT-PCR이 가지는 신속성과 편리성, 그리고 범용성을 모두 해결하지는 못한다 (63), (64). 다만 고품질 RNA 확보를 위한 virome capture 기법과 같은 분자생물학적 기술진보와 MinION 등 NGS 장비의 소형화를 포함한 기계공학적 기술의 발달과 더불어 가까운 시일내에 신속성을 가진 간편하고 유용한 NGS 분석기법이 활용될 날이 머지않아 다가온다면 신종바이러스에 의한 감염병 대응에 WGS정보가 임상적으로 유용하게 활용될 수 있을 것이다.
Go to : 
CONCLUSION
대유행의 원인 병원체인 바이러스를 분리하거나 바이러스가 가지는 유전자 특성을 탐색하는 방법은 모두 증폭이라는 공통점이 있다. 이러한 증폭 과정을 거치지 않고 바이러스가 가지는 항원을 직접 탐지하거나 바이러스가 숙주(사람)에 감염된 이후 숙주의 면역반응에 의하여 나타나는 항체를 측정하는 방법도 병원체 인지 분야에 중요하게 활용되는 기술이다. 그러나 항원탐지는 주로 바이러스 단백질을 표적으로 하므로 증폭이 불가능 하고 숙주 면역반응의 변화를 탐지하기 위해서는 감염 후 일정시간이 필요하다. 결론적으로 조작의 간편성과 범용성에도 불구하고 항원 항체반응을 응용한 인지기술은 높은 민감도와 특이도가 필요한 대유행 초기의 환자 발견을 위한 신속한 진단방법으로는 적절하지 않다고 할 수 있다. 다만 일정기간이 경과된 후 집단면역도 분석에는 항체 분석법이 필수적으로 요구되며, 다양한 조직의 바이러스 분포 등 연구목적의 항원 탐색은 반드시 필요한 기술이다. 하지만 현재 환자의 확진에 사용되고 있는 유전자 진단기술로는 임상적 증상 소실 이후에도 감염성 바이러스의 존재가 확인되지 않은 상태로 바이러스 유전자의 일부가 계속 검출되는 재양성 사례가 발생되기도 하는데 이에 대한 평가가 시급한 상황이다. 또한 젊은 성인의 경우 SARS-CoV-2에 감염되었더라도 비교적 임상증상이 미약하거나 자각하지 못한 채로 진단 대상에서 제외되는 경우도 지속적으로 보고되고 있어 이러한 사례에 대한 새로운 진단 체계의 도입 및 적용이 요구되고 있다.
새롭게 출현한 신종 바이러스성 질병의 발생초기에 원인 병원체를 신속하고 정확하게 인지하는 역량은 공중보건학적 대응 방향 설정을 위한 과학 근거 제시에 매우 중요하다. 따라서 COVID-19 대응에 다양하게 활용되고 있는 현재 진단법들이 가지는 한계는 가능한한 개선되어야 하고, 이를 위한 미래 진단기술 개발 연구분야(Box 1)를 제시하면서 논고를 마친다.
Box 1. 미래 진단기술 개발 연구분야
1) 미지의 바이러스를 쉽게 복제할 수 있는 세포배양 플랫폼 전략 개발; 진단대상 병원체를 확보하는 기술은 질병의 원인규명에 매우 중요하다.
2) 다양한 임상 검체로부터 고품질의 유전자를 안정적으로 분리할 수 있는 정제방법 개발; 안정적인 임상 검체 처리기술은 병원체 유전자 진단의 민감도를 높이기 위한 중요한 수단이다.
3) 기존의 발광 화학기술 한계를 뛰어넘는 감별진단용 다중 발광탐지기술 개발; 증폭된 유전자의 분별 기술을 확대함으로써 다중진단으로 임상양상과 병원체를 보다 면밀하게 연관지을 수 있다.
4) 대용량 유전체 정보를 쉽게 분석할 수 있는 간편한 알고리즘 개발; 다양한 병원체 고유특성이 암호화 되어있는 전장 유전체 정보가 쉽게 분석된다면, 질병중재에 보다 많은 지식을 단시간내에 확보할 수 있게 된다.
5) 유전자 증폭기술의 시간적 공간적 한계를 극복할 수 있는 대용량 현장 신속진단법(POC) 및 기기의 개발; 민감도와 특이도가 qRT-PCR과 동등 또는 그 이상의 성능을 가진 대용량의 신속, 간편한 현장 진단법은 광범위한 집단 감염으로 나타나는 판데믹의 특성상 의료인 및 의료자원의 고갈을 방지하고 의료적 판단에 신속한 접근성이 확보될 수 있다.
6) 의미있는 분자표식(molecular signature) 탐색과 기능 규명; 유전자 탐지 이외의 새로운 생물학적 지표를 찾아냄으로써 진단, 치료 예방 전략 수립에 방향성을 제시할 수 있다.
Go to : 




 PDF
PDF Citation
Citation Print
Print


 XML Download
XML Download