초록
연구 재료 및 방법
평활한 티타늄시편(NE0), 산처리만 시행한 티타늄시편(E0), 마이크로그루브 및 산처리된 티타늄시편(E60/10), 파이브로넥틴을 고정시킨 평활한 티타늄시편(NE0FN), 산처리 및 파이브로넥틴을 고정시킨 티타늄시편(E0FN), 그리고 마이크로그루브 및 산처리 후 파이브로넥틴을 고정시킨 티타늄시편(E60/10FN) 실험군 제작 후 인간치은섬유아세포의 세포행동 관련 44개 유전자에 대한 실시간 중합효소연쇄반응 실험을 진행하였다.
결과
인간치은섬유아세포 부착과 증식 등에 관여하는 4종류 신호전달 경로가 활성화되었다. Focal adhesion 경로에 속하는 Integrin α5, Integrin β1, Integrin β3, Talin-2 유전자들, PI3K-AKT 신호 전달 경로에 속하는 AKT1, AKT2, NF-κB 유전자들, MAPK 신호전달 경로에 속하는 MEK2, ERK1, ERK2 유전자들, 세포주기 신호 전달 경로에 속하는 cyclin D1, CDK4, CDK6 유전자들이 마이크로그루브-파이브로넥틴 복합 티타늄표면(E60/10FN)에서 상향조절되었다.
Abstract
Purpose
To determine the effects of the microgroove-fibronectin complex surface on the expression of various genes related to cellular activity in human gingival fibroblasts.
Materials and Methods
Smooth titanium specimens (NE0), acid-treated titanium specimens (E0), microgroove and acid-treated titanium specimens (E60/10), fibronectin-fixed smooth titanium specimens (NE0FN), acid-treated and fibronectin-immobilized titanium specimens (E0FN), and microgroove and acid-treated titanium specimens immobilized with fibronectin (E60/10FN) were prepared. Real-time polymerase chain reaction experiments were conducted on 44 genes related to cell behavior of human gingival fibroblasts.
Results
Adhesion and proliferation of human gingival fibroblast on microgroove-fibronectin complex titanium were activated through four types of signaling pathway. Integrin α5, Integrin β1, Integrin β3, Talin-2, which belong to the focal adhesion pathway, AKT1, AKT2, NF-κB, which belong to the PI3K-AKT signaling pathway, MEK2, ERK1, ERK2, which belong to the MAPK signaling pathway, and Cyclin D1, CDK4, CDK6 genes belonging to the cell cycle signaling pathway were upregulated on the microgroove-fibronectin complex titanium surface (E60/10FN).
무치악 부위의 보철적 수복을 위해 임플란트 치료가 도입된 이후로 임플란트 주위 조직의 단기적 및 장기적 예후를 개선하기 위한 많은 시도들이 이루어져 왔다. 이러한 목표를 달성하기 위한 방법에는 비가동성 각화치은을 보존하거나 증대하는 수술적 방법과 임플란트 주위 연조직의 생물학적 폭경을 유지하고 과풍융되지 않은 임플란트 치관을 제작하여 예후 개선에 기여하는 보철적 방법 등이 있다.1 또다른 방법으로 임플란트-연조직계면의 안정성을 개선하여 임플란트의 단기적 및 장기적 예후를 개선하는 방법이 보고되고 있다.1 예시로 기존의 절삭형 티타늄 지대주를 지르코니아 지대주 등으로 교체하여 지대주 자체의 재료를 변화시키는 방법, 폴리싱이나 샌드블라스팅 등을 통해 지대주 표면 성상을 변화시키는 방법 그리고 복합레진 코팅, 콜라겐 코팅, 비타민E 코팅 등의 다양한 코팅을 통한 표면처리를 시행하는 방법 등이 있다.2 이 중 마이크로그루브(microgroove) 티타늄표면에 산처리를 시행한 후 파이브로넥틴(Fibronectin, FN) 처리를 시행하여 임플란트 주위 연조직반응을 개선시키는 방법이 주목받고 있다.
기존 연구에서 티타늄표면상의 마이크로그루브는 인간 치은섬유아세포의 contact guidance activity를 증진시키는 것으로 보고되고 있으며, 구체적으로는 작은 크기의 마이크로그루브들보다 폭 60 μm, 깊이 10 μm의 마이크로그루브에서 contact guidance activity가 보다 더 증진되었다고 보고하였다.3 또한, 마이크로그루브 티타늄 표면은 인간 치은섬유아세포의 형태를 변화시키고, 파이브로넥틴 mRNA수준 및 파이브로넥틴 분비량을 증가시키는 것으로 알려졌다.4 마이크로그루브 티타늄표면에 부가적인 산처리를 실시한 연구에서는 마이크로그루브 티티늄표면 및 산처리가 인간 치은섬유아세포의 증식과 유전자 및 단백질발현의 유의미한 증진을 이끌어내는 것으로 보고되었다.5 이후에는, 마이크로그루브 티타늄 표면에 산처리를 시행한 후 파이브로넥틴 코팅을 실시한 표면이 인간 치은섬유아세포의 반응을 더욱 증진시킬 수 있는가에 대한 의문이 제기되었고 이에 대한 검증의 필요성이 대두되었다. 이에 대해 다른 연구에서는 파이브로넥틴이 다른 세포외기질 단백질의 assembly를 조직하고 세포부착, 세포이주, 세포신호전달을 촉진시킨다고 하였다.6 이는 인간 섬유아세포의 이주에 파이브로넥틴이 필요하기 때문에 세포성 파이브로넥틴(cellular FN)의 부재시 세포 운동성이 감소한다고 설명한 다른 논문의 내용과 일치한다.7 이러한 현상은 파이브로넥틴이 세포외기질 중 핵심 구성요소로서 세포표면의 Integrin 수용체와 상호작용을 하면서 다양한 세포활동을 조절하는 세포외 기질 단백질이기 때문에 나타나는 것으로 규명되었다.8
선행 연구에서 마이크로그루브-파이브로넥틴 복합 티타늄표면에서 세포증식 측정, 역전사 중합효소연쇄반응 및 단백질발현분석을 통해 마이크로그루브-파이브로넥틴 복합 티타늄표면이 인간 섬유아세포의 증식을 크게 증가시킨다는 사실을 밝히고 인간 치은섬유아세포의 부착과 증식에 관여하는 76개의 유전자 중 가장 중요한 10개의 유전자를 선정함으로써 파이브로넥틴이 인간 치은섬유아세포에 미치는 영향에 대해 평가한 바 있으나,9 실험대상이 되는 유전자들에 대한 정량적 실시간 중합효소연쇄반응 실험의 부재 때문에 파이브로넥틴에 의해 발현되는 유전자들에 대한 정량적 분석 및 유전자 발현량에 대한 비교분석은 이루어지지 않은 바 있다. 본 연구의 목적은 마이크로그루브-파이브로넥틴 복합 티타늄표면이 인간 치은섬유아세포의 세포-기질접착(focal adhesion), 세포증식, 세포주기진행 등과 연관된 44개 유전자발현에 미치는 영향을 실시간 정량적 중합효소연쇄반응을 통해 분석하고, 그 이유에 대해 선행연구보다 더 깊이 있고 폭 넓게 고찰하는데 있다. 이를 위해 티타늄시편을 제작하고 파이브로넥틴을 최적 고정 농도로 고정 후 인간 치은섬유아세포 배양을 진행하였다. 이후 실시간 정량적 중합효소연쇄반응 실험을 통해 세포부착, 세포-기질접착과 세포증식에 관련된 다양한 유전자발현을 평가하였다.
티타늄시편 제작과 마이크로그루브, 산처리, 파이브로넥틴처리는 선행연구에서 사용한 방법과 동일한 방법으로 시행하였다.9 파이브로넥틴을 고정시킨 평활한 티타늄시편(NE0FN), 산처리 및 파이브로넥틴을 고정시킨 티타늄시편(E0FN), 마이크로그루브 및 산처리 후 파이브로넥틴을 고정시킨 티타늄시편(E60/10FN)이 완성되었고, 평활한 티타늄시편(NE0), 산처리만 시행한 티타늄시편(E0), 마이크로그루브 및 산처리된 티타늄시편(E60/10) 등 총 6개의 실험군을 제작하였다.
실험을 위해 ATCC (ATCC® PCS-201-018™, Manassas, USA)에서 인간 치은섬유아세포를 구입하였다. 치은섬유아세포는 37°C에서 95% 공기와 5% CO2로 채워진 T-75 flask (Sigma Aldrich, St. Louis, USA)내에서 계대배양하였다. 본 연구에서는 계대배양 후 3 - 5세대에 해당되는 세포를 이용하였다. 세포부착과 세포증식에 관여하는 44개 유전자를 선정하여 mRNA의 상대적 발현량을 반정량적 실시간 중합효소연쇄반응(semi quantitative real-time RT-PCR)을 통해 분석하였다. cDNA 증폭은 PCR thermal cycler (Bio-Rad, Hercules, USA)를 이용하여 94°C에서 30초, 58°C에서 45초, 72°C에서 30초간 실시하였다. Table 1에 모든 유전자의 primer와 predesigned probe가 제시되어 있다. 세포밀도가 well당 1 × 104개가 되도록 24-well tissue culture plate (Sigma Aldrich)의 NE0, E0, E60/10, NE0FN, E0FN, E60/10FN에 인간 치은섬유아세포를 접종하여 37°C, 5% CO2 하에서 48, 72시간 동안 배양하였다. Total RNA는 Trizol (Invitrogen, San Jose, USA)로 추출하였고 RNA의 농도는 NanoDrop 1000 (NanoDrop Technologies, Wilmington, USA)을 이용하여 결정하였다. Total RNA의 1 μg을 iScript cDNA Synthesis Kit (Bio-Rad)를 이용하여 cDNA를 합성하였다. 내부 대조군인 GAPDH (Glyceraldehyde 3-phosphate dehydrogenase)의 발현량을 기준으로 하여 differential display PCR을 통해 인간치은섬유아세포의 mRNA을 최종선정하였고 이의 유전자발현은 TaqMan® Gene Expression Assay Kit (Applied Biosystems, Waltham, USA)을 이용하여 결정하였다. Table 1에는 본 연구의 Semi quantitative real-time RT-PCR에 사용한 유전자의 이름이 알파벳 순으로 나와있다. IQ Supermix (Bio-Rad)을 이용하여 Chromo-4 Reverse Transcription-Polymerase Chain Reactions (Bio-Rad Laboratories, Hemel Hempstead, UK)를 시행 후 MJ Opticon Monitor Analysis Software (Bio-Rad)를 이용하여 유전자발현을 정량화하였다. 측정값들은 GAPDH (Glyceraldehyde 3-phosphate dehydrogenase) 발현량으로 보정하여 E0, E60/10, NE0FN, E0FN, E60/10FN의 발현량을 대조군인 NE0에 상대적인 배수로 표시하였다. ELISA를 이용한 실시간 중합효소연쇄반응을 동시에 그리고 독립적으로 5번씩 시행하였으며, 평균값과 표준편차를 계산하였다. NE0, E0, E60/10, NE0FN, E0FN, E60/10FN간의 평균값을 비교하기 위해 일요인 분산분석(one-way ANOVA)을 사용하였다. 실험결과 간 상관관계를 분석하기 위하여 피어슨상관관계분석을 시행하였다. 모든 통계분석에는 SPSS 18.0 (SPSS Inc., Chicago, USA)을 사용하였다.
본 연구에서는 인간 치은섬유아세포 부착 및 증식에 관련된 44개 유전자가 선택되었고, mRNA 발현은 반정량적 실시간 역전사 중합효소연쇄반응을 통해 분석하였다(Table 1). 실험 48, 72시간 후 실시간 중합효소연쇄반응을 통해 유전자발현량을 확인하였다. 실험대상이 되는 44개 유전자들은 각 유전자가 속한 신호전달 졍로에 따라 focal adhesion, PI3K-AKT, MAPK, 및 cell cycle 신호전달 경로의 4개 군으로 분류한 뒤 반정량적 실시간 역전사 중합효소 연쇄반응 실험결과를 도출하였다. 각 군간 중복되는 유전자들은 보다 더 핵심적인 역할을 하는 신호전달 경로군에 포함시켜 결과로 제시하였다.
먼저 focal adhesion 신호전달 경로군의 결과는 다음과 같다. FN은 실험 48, 72시간 후 E60/10, E60/10FN군이 다른 모든 군들과 비교 시 유의미한 상향조절이 관찰되었다(P < 0.001). ON은 실험 48시간 후 E60/10FN, 실험 72시간 후 E0FN, E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). TIMP1은 실험 48, 72시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). ILK는 실험 48시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었고(P < 0.001), 실험 72시간 후 NE0FN이 NE0, E0, E0FN과 비교시 상향조절되었다(P < 0.01). Integrin α2는 실험 48시간 후 유의미한 상향조절이 관찰되지 않았고, 실험 72시간 후에는 E0, E60/10가 E0FN과 비교시 상향조절되었다(P < 0.001). Integrin α5는 실험 48, 72시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). Integrin αν는 실험 48시간 후 E60/10, E0FN, E60/10FN이 다른 모든 군들과 비교 시 하향조절되었고(P < 0.001), 실험 72시간 후에는 NE0, E0에서 상향조절되었다(P < 0.001). Integrin β1은 실험 48시간 후 E60/10FN을 NE0, E0, E60/10와 비교시 상향조절이 이루어졌고(P < 0.01), 실험 72시간 후에는 E60/10FN을 NE0와 비교시 상향조절되었다(P < 0.05). Integrin β3는 실험 48시간 후 E60/10FN이 NE0, E0, E60/10, NE0FN와 비교 시 상향조절되었고(P < 0.001), 실험 72시간 후 NE0FN, E0FN, E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001), Syndecan-4는 실험 48시간 후 E60/10FN(P < 0.001), 실험 72시간 후 E60/10, E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). Talin-2는 실험 48, 72시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절이 관찰되었다(P < 0.001). Paxillin은 실험 48시간 후 E60/10FN (P < 0.001), 실험 72시간 후 E0FN, E60/10FN이 다른 모든 군들과 비교 시 상향조절이 관찰되었다(P < 0.001). p130CAS는 실험 48시간 후 E60/10FN이 NE0, E0, E60/10, NE0FN과 비교 시 상향조절이 이루어졌고(P < 0.001), 실험 72시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). c-Src는 실험 48, 72시간 모두 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). c-Crk는 실험 48, 72시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). c-Jun은 실험 48시간 후 E60/10, E0FN, E60/10FN (P < 0.001), 실험 72시간 후 E60/10, NE0FN, E0FN, E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.01). RAC1은 실험 48시간 후 E60/10FN이 E0와 비교시 상향조절되었고(P < 0.05), 실험 72시간 후에는 E60/10, E60/10FN이 NE0, E0, E0FN과 비교시 상향조절되었다(P < 0.01)(Fig. 1).
PI3K-AKT 신호전달 경로군의 결과는 다음과 같다. AKT1은 실험 48시간 후 E60/10 (P < 0.001), E60/10FN이 NE0, E0, NE0FN, E0FN과 비교 시 상향조절되었다(P < 0.001). AKT2는 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). PI3KCG는 실험 48시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었고(P < 0.001), 실험 72시간 후에는 E60/10FN이 NE0, E0, E60/10, NE0FN과 비교시 상향조절되었다(P < 0.01). PRKAR1A는 실험 48시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.01). NF-kB는 실험 48, 72시간 모두에서 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). CREB1은 실험 48, 72시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). CREB2는 실험 48시간 후 NE0FN, E0FN, E60/10FN이 NE0, E0, E60/10과 비교 시 상향조절되었고(P < 0.001), 실험 72시간 후에는 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001)(Fig. 2).
MAPK 신호전달 경로군의 결과는 다음과 같다. EGFR은 실험 48시간 후 E60/10FN(P < 0.001), 실험 72시간 후 E60/10, NE0FN, E0FN, E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). CTGFR은 실험 48시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절이 관찰되었고(P < 0.001), 실험 72시간 후에는 E60/10, NE0FN, E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.05). TGFBR1은 실험 48시간 후 E60/10FN (P < 0.001), 실험 72시간 후 E60/10, E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). TGFBR2는 유의미한 상향조절이 관찰되지 않았다. MEK2는 실험 48시간 후 E60/10FN을 E0와 비교시 상향조절되었고(P < 0.05), 실험 72시간 후에는 E60/10, E0FN, E60/10FN을 NE0, E0와 비교시 상향조절되었다(P < 0.001). ERK1은 실험 48시간 후 E60/10FN (P < 0.001), 실험 72시간 후 E0FN, E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). ERK2는 실험 48시간 후 E60/10FN (P < 0.001), 실험 72시간 후 E60/10이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). c-Fos는 실험 48시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었고(P < 0.001), 실험 72시간 후 E60/10FN을 NE0과 비교 시 상향조절되었다(P < 0.05). JNK1은 실험 48시간 후 E0FN, E60/10FN이 다른 모든 군들과 비교 시 상향조절이 이루어졌고(P < 0.001), 실험 72시간 후 E60/10이 NE0, NE0FN과 비교 시 상향조절되었다(P < 0.01). JNK2는 실험 48시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었고(P < 0.001), 실험 72시간 후 E60/10을 NE0와 비교 시 상향조절되었다(P < 0.05). PAK1은 실험 48시간 후 E60을 NE0, E0, NE0FN과 비교 시 상향조절되었고(P < 0.01), 실험 72시간 후에는 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). PEBP1은 실험 48시간 후 E60/10FN을 NE0, E0, E60/10, NE0FN과 비교 시 하향조절되었고(P < 0.001), 실험 72시간 후 E60/10FN을 NE0, E0, NE0FN, E0FN과 비교 시 하향조절되었다(P < 0.001)(Fig. 3).
Cell cycle 신호전달 경로군의 결과는 다음과 같다. CDK2는 실험 48시간 후 E0FN (P < 0.001), E60/10FN이 다른 모든 군들과 비교 시 상향조절되었고(P < 0.001), 실험 72시간 후 E60/10이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). CDK4는 실험 48시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었고(P < 0.001), 실험 72시간 후 E60/10을 NE0, NE0FN과 비교 시 상향조절되었다(P < 0.01). CDK6는 실험 48, 72시간 모두 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). Cyclin D1은 실험 48, 72시간 후 E60/10FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). Cyclin E는 실험 48시간 후 E60/10FN, 실험 72시간 후 E60/10이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). p21CIP1은 실험 48, 72시간 모두 E60/10이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001). p27KIP1은 실험 48시간 후 E60/10FN을 NE0와 비교 시 유의미한 상향조절이 관찰되었고(P < 0.01), 실험 72시간 후 E60/10, E60/10FN이 E0와 비교시 상향조절되었다(P < 0.05). c-Myc는 실험 48시간 후 E60/10FN (P < 0.001), 실험 72시간 후 E60/10, E0FN, NE0FN이 다른 모든 군들과 비교 시 상향조절되었다(P < 0.001)(Fig. 4).
파이브로넥틴은 세포부착, 세포성장, 세포이주, 세포분화에 있어서 중요한 역할을 하며, Integrin과 같은 수용체에 결합하는 세포외기질의 고분자 당단백질로서 콜라겐, 피브린, Syndecan과 같은 다른 세포외기질 단백질과 결합한다.10 파이브로넥틴은 인테그린 수용체와 상호작용을 하면서 focal adhesion, AKT-PI3K, MAPK, 및 cell cycle 신호전달 경로를 활성화시켜 세포행동에 관여한다.
Focal adhesion 신호전달 경로에 속하는 Integrin α5, Integrin β1, Integrin β3는 파이브로넥틴 처리에 대해 상향조절되는 경향을 보였다(Fig. 1). 이는 막관통수용체로서 세포-세포 및 세포-기질 간 부착을 촉진시키는 역할을 하는 Integrin 본연의 기능과 일치하는 것으로,11 세포증식이 발생하는 상황에서 그 활성이 더욱 강하게 발현된 것으로 풀이된다. Talin-2와 Paxillin은 E60/10FN이 상향조절되었는데, 이는 Talin-2와 Paxillin이 세포-기질간접촉 부위에서 세포골격단백질 역할로 세포부착 및 세포증식에 관여함에 따라 나타난 결과로 유추된다.12,13 Syndecan-4는 파이브로넥틴 처리시 발현량이 증가하는 경향을 보였는데(Fig. 1), 이는 Syndecan-4가 세포표면에서 co-receptor로서 파이브로넥틴 처리시 primary signaling receptor의 ligand-dependent activation을 조절하거나 액틴기반 세포골격과 상호작용하는 등 다양한 기능을 수행하여 세포증식 등에 관여하기 때문으로 추측된다.14
AKT1, AKT2는 PI3K-AKT 신호전달 경로의 핵심 유전자로서 세포생존과 세포대사에 중요한 역할을 한다.15 세포생존에 AKT가 관여하는 것은 AKT의 가장 중요한 역할 중에 하나인데, 세포자멸사(apoptosis) 및 세포생존에 관여하는 다양한 요소를 활성화시킴으로써 성장인자와 관련된 세포생존을 촉진하고 세포자멸사를 억제한다.15 본 연구에서 AKT1, AKT2에서 파이브로넥틴 처리시 전반적으로 AKT1, AKT2 발현량이 증가한 것은 세포생존에 관련된 활성이 증가와 연관된 것으로 사료된다. 또한 NF-κB는 AKT에 의해 활성화되면서 DNA전사, 세포생존, 사이토카인 생산 등을 조절하는 핵심적인 역할을 하는 유전자이다.16 NF-κB는 실시간 중합효소연쇄반응 실험에서 전반적으로 다른 유전자들과 비슷한 발현양상을 보이면서 72시간 후 E60/10FN에서 매우 상승된 발현량을 보였는데, 이는 NF-κB가 PI3K-AKT pathway의 다양한 상위 신호를 전달받는 AKT의 영향을 크게 받았을 것으로 사료된다(Fig. 2).
MAPK 신호전달 경로에 속하는 EGFR, CTGFR, TGFBR1와 같은 수용체는 E60/10FN에서 활성이 크게 증가하는 양상을 보였다. 이는 마이크로그루브, 산처리, 파이브로넥틴 등 다양한 요인에 의해 세포증식이 촉진된 상태에서 성장인자와 같은 세포외리간드 발현이 증가되고 수용체활성이 증가됨에 따라 MAPK 신호전달 경로가 활성화된 결과로 발생되었을 것이다.17 EGFR,CTGFR,TGFBR1와 같은 수용체에 의해 Ras가 활성화된 이후에는 일련의 kinase cascade를 거쳐18 MEK와 ERK가 인산화된다. 이러한 연속적인 과정을 통해 E60/10FN에서 유전자 발현이 크게 나타난 MEK2, ERK1, ERK2 실험결과를 EGFR, CTGFR, TGFBR1 실험결과와 연관지어 설명할 수 있다(Fig. 3). 또한 ERK는 c-Fos의 carboxylterminal transactivation domain (TAD)안의 multiple residues를 인산화하여 전사활성을 증가시키는 역할을 한다. 따라서 c-Fos의 발현양상 역시 ERK1, ERK2의 발현양상을 따랐다고 추측할 수 있다.19
Cyclin D1은 CDK4와 CDK6와 결합하여 세포주기 중 G1기의 진행과 G1/S 이행에 관여하는 E2F 전사인자발현을 자극한다. CDK6는 G1 중기에 활발히 활성화되어 Cyclin D1과 더불어 CDK억제제인 p21CIP1 및 p27KIP1을 강력하게 통제한다.9 세포주기 과정에서 Cyclin E는 CDK2와 함께, Cyclin D는 CDK4/6와 함께 복합체를 형성하게 된다. 이와 같은 복합체 형성과정을 통해 Cyclin E-CDK2간, Cyclin D-CDK4/6간의 실험 결과의 유사성을 설명할 수 있다(Fig. 4). 반대로 p21CIP1은 모든 종류의 Cyclin/CDK 복합체를 억제하는 역할을 함으로서 세포주기를 조절한다.20 p27KIP1역시 암호화된 단백질이 Cyclin E-CDK2 복합체 및 Cyclin D-CDK4복합체의 활성을 방해하는 역할을 한다. p21CIP1는 E60/10에서 더 강한 유전자발현이 나타났다(Fig. 4, E60/10). 이는 마이크로그루브 티타늄표면에서 p21의 강한 발현이 나타났다고 보고한 다른 선행연구 결과와 일치한다.21,22 p27KIP1은 E60/10, E60/10FN에서 상향조절되었는데(Fig. 4), 이는 p27KIP1이 마이크로그루브와 같은 미세환경을 감지하는 역할을 하기 때문에 나타난 결과이다(Fig. 4).21 c-Myc은 p21 전사억제 및 p27분해에 관련된 S-Phase Kinase Associated Protein 2 (Skp2)를 활성화하여 세포주기진행을 차단하는 기능을 가진 p21이나 p27과 같은 유전자발현을 억제한다.23 즉, c-Myc은 p27KIP1이 보인 발현 양상과 유사하게 E60/10FN이 상향조절됨으로써 활성화된 p27KIP1을 억제하려는 경향을 보였다고 할 수 있다.
본 연구에서 다룬 44개의 유전자들은 모두 세포증식, 세포부착, 세포이주 등에 직간접적으로 연관된 focal adhesion, AKT-PI3K, MAPK, 및 cell cycle 신호전달 경로에 속하는 유전자들이다. 일부 유전자들을 제외하고 대부분의 유전자들은 E60/10FN, E0FN, NE0FN, E60/10, E0, NE0 순으로 상향조절되는 경향을 보였다. 이는 마이크로그루브, 산처리에 의해 표면적이 증가하여 파이브로넥틴이 표면에 고정되는 양이 증가하기 때문에 세포증식이 더욱 활성화되었기 때문이다. 파이브로넥틴 처리를 하지 않았더라도 마이크로그루브 존재 유무나 산처리 여부에 따라서 활성화 양상이 달라졌는데, 이는 마이크로그루브의 존재가 contact guidance activity를 증가시켜 세포 증식에 안정된 환경을 제공하기 때문으로 추측된다. 다만, 일부 유전자들에서 실험 48, 72시간 후 실험결과가 일관되게 나타나지 않거나, 유전자 기능에 상반되는 발현결과가 나타난 경우가 있었기 때문에 이에 대한 추가적인 연구가 필요하다.
Acknowledgements
This research was supported by a grant of the Korea Health Technology R&D Project through the Korea Health Industry Development Institute (KHIDI), funded by the Ministry of Health and Welfare, Republic of Korea (Grant No. HI16C1838).
References
1. Rompen E, Domken O, Degidi M, Pontes AE, Piattelli A. 2006; The effect of material characteristics, of surface topography and of implant components and connections on soft tissue integration: a literature review. Clin Oral Implants Res. 17(Suppl 2):55–67. DOI: 10.1111/j.1600-0501.2006.01367.x. PMID: 16968382.

2. Corvino E, Pesce P, Mura R, Marcano E, Canullo L. 2020; Influence of modified titanium abutment surface on peri-implant soft tissue behavior: a systematic review of in vitro studies. Int J Oral Maxillofac Implants. 35:503–19. DOI: 10.11607/jomi.8110. PMID: 32406646.

3. Lai Y, Chen J. 2015; Comparative study on contact guidance activity of human gingival fibroblasts on microgroove surfaces. Zhonghua Kou Qiang Yi Xue Za Zhi. 50:33–7. PMID: 25779073.
4. Chou L, Firth JD, Uitto VJ, Brunette DM. 1995; Substratum surface topography alters cell shape and regulates fibronectin mRNA level, mRNA stability, secretion and assembly in human fibroblasts. J Cell Sci. 108:1563–73. DOI: 10.1242/jcs.108.4.1563. PMID: 7615675.

5. Kim SY, Oh N, Lee MH, Kim SE, Leesungbok R, Lee SW. 2009; Surface microgrooves and acid-etching on titanium substrata alter various cell behaviors of cultured human gingival fibroblasts. Clin Oral Implants Res. 20:262–72. DOI: 10.1111/j.1600-0501.2008.01652.x. PMID: 19397638.
6. Leiss M, Beckmann K, Girós A, Costell M, Fässler R. 2008; The role of integrin binding sites in fibronectin matrix assembly in vivo. Curr Opin Cell Biol. 20:502–7. DOI: 10.1016/j.ceb.2008.06.001. PMID: 18586094.

7. Missirlis D, Haraszti T, Kessler H, Spatz JP. 2017; Fibronectin promotes directional persistence in fibroblast migration through interactions with both its cell-binding and heparin-binding domains. Sci Rep. 7:3711. DOI: 10.1038/s41598-017-03701-0. PMID: 28623309. PMCID: PMC5473823. PMID: f05ac0fbbf9a498f9e9f5b297e6e308e.

8. Mao Y, Schwarzbauer JE. 2005; Fibronectin fibrillogenesis, a cell-mediated matrix assembly process. Matrix Biol. 24:389–99. DOI: 10.1016/j.matbio.2005.06.008. PMID: 16061370.

9. Kim EC, Lee DY, Lee MH, Lee HJ, Kim KH, Leesungbok R, Ahn SJ, Park SJ, Yoon JH, Jee YJ, Lee SC, Lee SW. 2018; The effect of fibronectin-immobilized microgrooved titanium substrata on cell proliferation and expression of genes and proteins in human gingival fibroblasts. Tissue Eng Regen Med. 15:615–27. DOI: 10.1007/s13770-018-0153-7. PMID: 30603583. PMCID: PMC6171696.

10. Pankov R, Yamada KM. 2002; Fibronectin at a glance. J Cell Sci. 115:3861–3. DOI: 10.1242/jcs.00059. PMID: 12244123.

11. Hynes RO. 2002; Integrins: bidirectional, allosteric signaling machines. Cell. 110:673–87. DOI: 10.1016/S0092-8674(02)00971-6. PMID: 12297042.
12. Burridge K, Connell L. 1983; A new protein of adhesion plaques and ruffling membranes. J Cell Biol. 97:359–67. DOI: 10.1083/jcb.97.2.359. PMID: 6684120. PMCID: PMC2112532.

13. Turner CE, Glenney JR Jr, Burridge K. 1990; Paxillin: a new vinculin-binding protein present in focal adhesions. J Cell Biol. 111:1059–68. DOI: 10.1083/jcb.111.3.1059. PMID: 2118142. PMCID: PMC2116264.

14. Carey DJ. 1997; Syndecans: multifunctional cell-surface co-receptors. Biochem J. 327:1–16. DOI: 10.1042/bj3270001. PMID: 9355727. PMCID: PMC1218755.

15. Song G, Ouyang G, Bao S. 2005; The activation of Akt/PKB signaling pathway and cell survival. J Cell Mol Med. 9:59–71. DOI: 10.1111/j.1582-4934.2005.tb00337.x. PMID: 15784165. PMCID: PMC6741304.

16. Vanhaesebroeck B, Alessi DR. 2000; The PI3K-PDK1 connection: more than just a road to PKB. Biochem J. 346:561–76. DOI: 10.1042/bj3460561. PMID: 10698680. PMCID: PMC1220886.

17. Orton RJ, Sturm OE, Vyshemirsky V, Calder M, Gilbert DR, Kolch W. 2005; Computational modelling of the receptor-tyrosine-kinase-activated MAPK pathway. Biochem J. 392:249–61. DOI: 10.1042/BJ20050908. PMID: 16293107. PMCID: PMC1316260.

18. Avruch J, Khokhlatchev A, Kyriakis JM, Luo Z, Tzivion G, Vavvas D, Zhang XF. 2001; Ras activation of the Raf kinase: tyrosine kinase recruitment of the MAP kinase cascade. Recent Prog Horm Res. 56:127–55. DOI: 10.1210/rp.56.1.127. PMID: 11237210.

19. Monje P, Hernández-Losa J, Lyons RJ, Castellone MD, Gutkind JS. 2005; Regulation of the transcriptional activity of c-Fos by ERK. A novel role for the prolyl isomerase PIN1. J Biol Chem. 280:35081–4. DOI: 10.1074/jbc.C500353200. PMID: 16123044.
20. Xiong Y, Hannon GJ, Zhang H, Casso D, Kobayashi R, Beach D. 1993; p21 is a universal inhibitor of cyclin kinases. Nature. 366:701–4. DOI: 10.1038/366701a0. PMID: 8259214.

21. Lee SW, Kim SY, Rhyu IC, Chung WY, Leesungbok R, Lee KW. 2009; Influence of microgroove dimension on cell behavior of human gingival fibroblasts cultured on titanium substrata. Clin Oral Implants Res. 20:56–66. DOI: 10.1111/j.1600-0501.2008.01597.x. PMID: 19133333.

22. Huang NF, Lee RJ, Li S. 2010; Engineering of aligned skeletal muscle by micropatterning. Am J Transl Res. 2:43–55. PMID: 20182581. PMCID: PMC2826821.
23. Bretones G, Delgado MD, León J. 2015; Myc and cell cycle control. Biochim Biophys Acta. 1849:506–16. DOI: 10.1016/j.bbagrm.2014.03.013. PMID: 24704206.

Fig. 1
Expression of FN, ON, TIMP1, ILK, Integrin a2, Integrin a5, Integrin av, Integrin b1, Integrin b3, Talin-2, Paxillin, Syndecan-4, p130CAS, c-Src, c-Crk, c-Jun, RAC1 gene of human gingival fibroblasts cultured on smooth (NE0), acid-etched (E0), microgrooved (E60/10), and fibronectin-immobilized (NE0FN, E0FN, and E60/10FN) titanium substrata after 48 and 72 h of culture using semi quantitative real-time RT-PCR. The relative expression levels were analyzed by normalizing with GAPDH, and are presented as fold changes relative to the control, NE0 at 48 h of culture. One-way ANOVA (n = 5). ***: significant difference (P < 0.001). **: significant difference (P < 0.01). *: significant difference (P < 0.05). Note that only significant differences between the highest gene expression levels and other gene expression levels are presented.
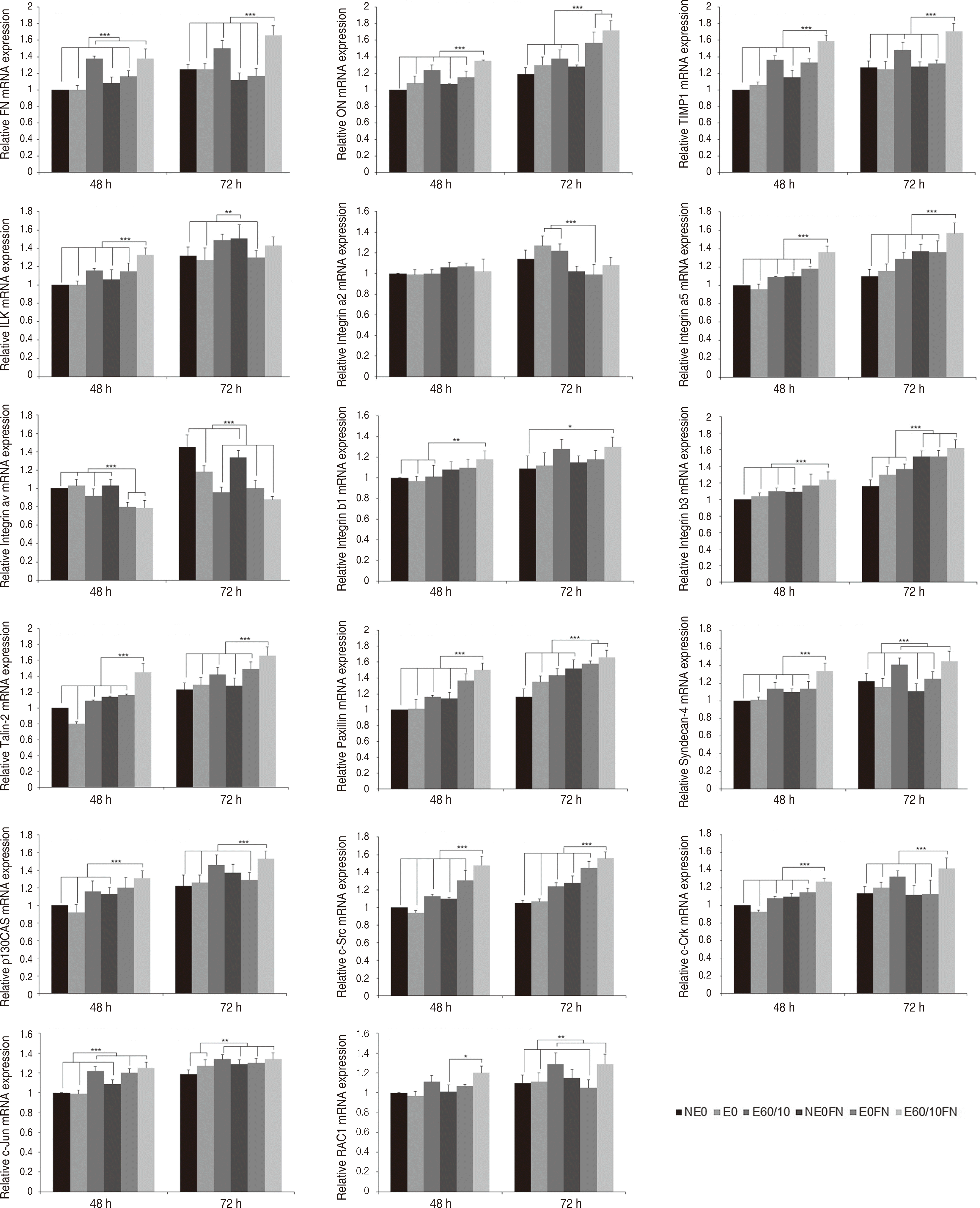
Fig. 2
Expression of AKT1, AKT2, PIK3CG, PRKAR1A, NF-κB, CREB1, and CREB2 gene of human gingival fibroblasts cultured on smooth (NE0), acid-etched (E0), microgrooved (E60/10), and fibronectin-immobilized (NE0FN, E0FN, and E60/10FN) titanium substrata after 48 and 72 h of culture using semi quantitative real-time RT-PCR. The relative expression levels were analyzed by normalizing with GAPDH, and are presented as fold changes relative to the control, NE0 at 48 h of culture. One-way ANOVA (n = 5). ***: significant difference (P < 0.001). **: significant difference (P < 0.01). Note that only significant differences between the highest gene expression levels and other gene expression levels are presented.
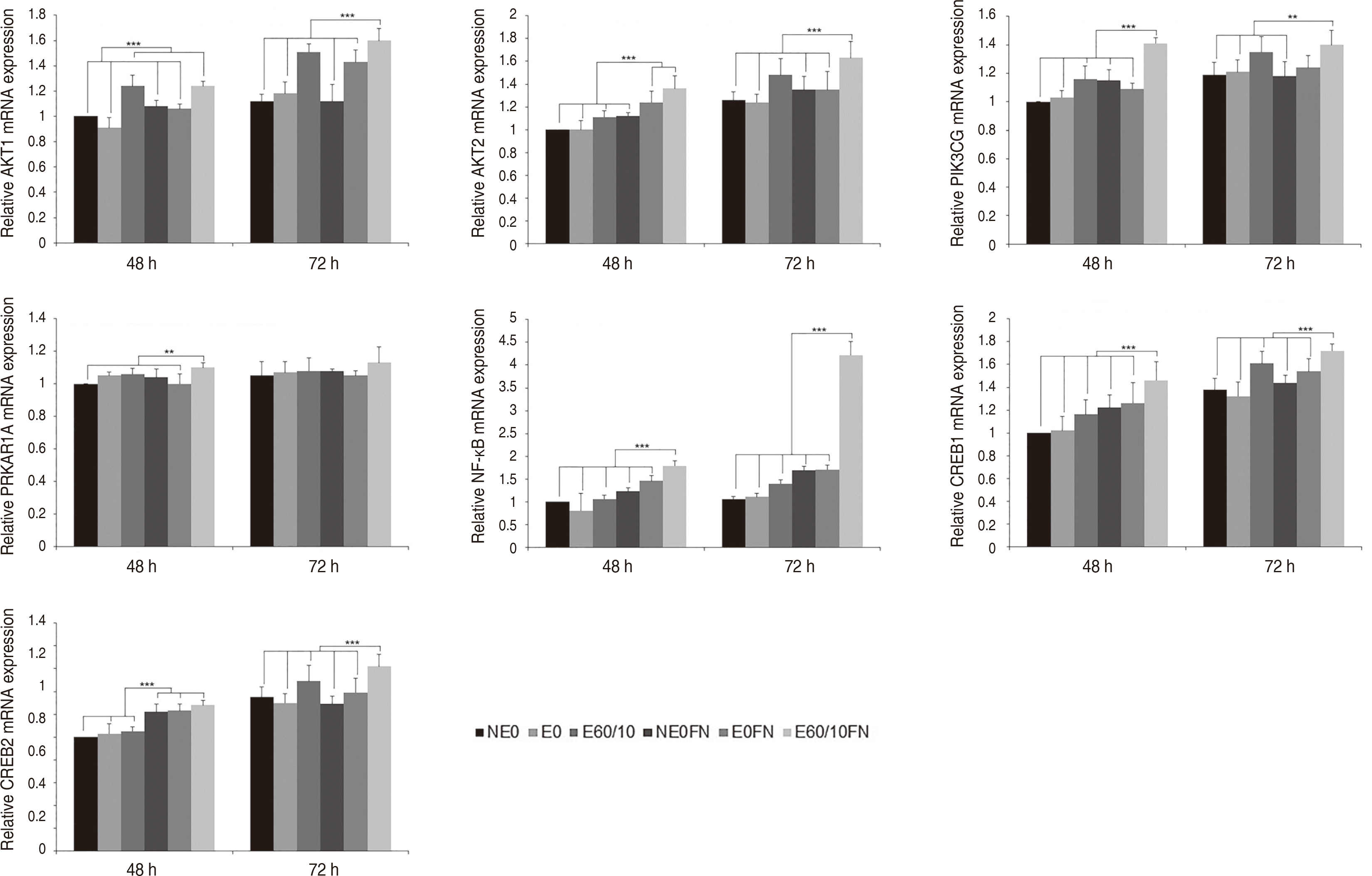
Fig. 3
Expression of EGFR, CTGFR, TGFBR1, TGFBR2, MEK2, ERK1, ERK2, c-Fos, JNK1, JNK2, PAK1, and PEBP1 gene of human gingival fibroblasts cultured on smooth (NE0), acid-etched (E0), microgrooved (E60/10), and fibronectin-immobilized (NE0FN, E0FN, and E60/10FN) titanium substrata after 48 and 72 h of culture using semi quantitative real-time RT-PCR. The relative expression levels were analyzed by normalizing with GAPDH, and are presented as fold changes relative to the control, NE0 at 48 h of culture. One-way ANOVA (n = 5). ***: significant difference (P < 0.001). **: significant difference (P < 0.01). *: significant difference (P < 0.05). Note that only significant differences between the highest gene expression levels and other gene expression levels are presented.
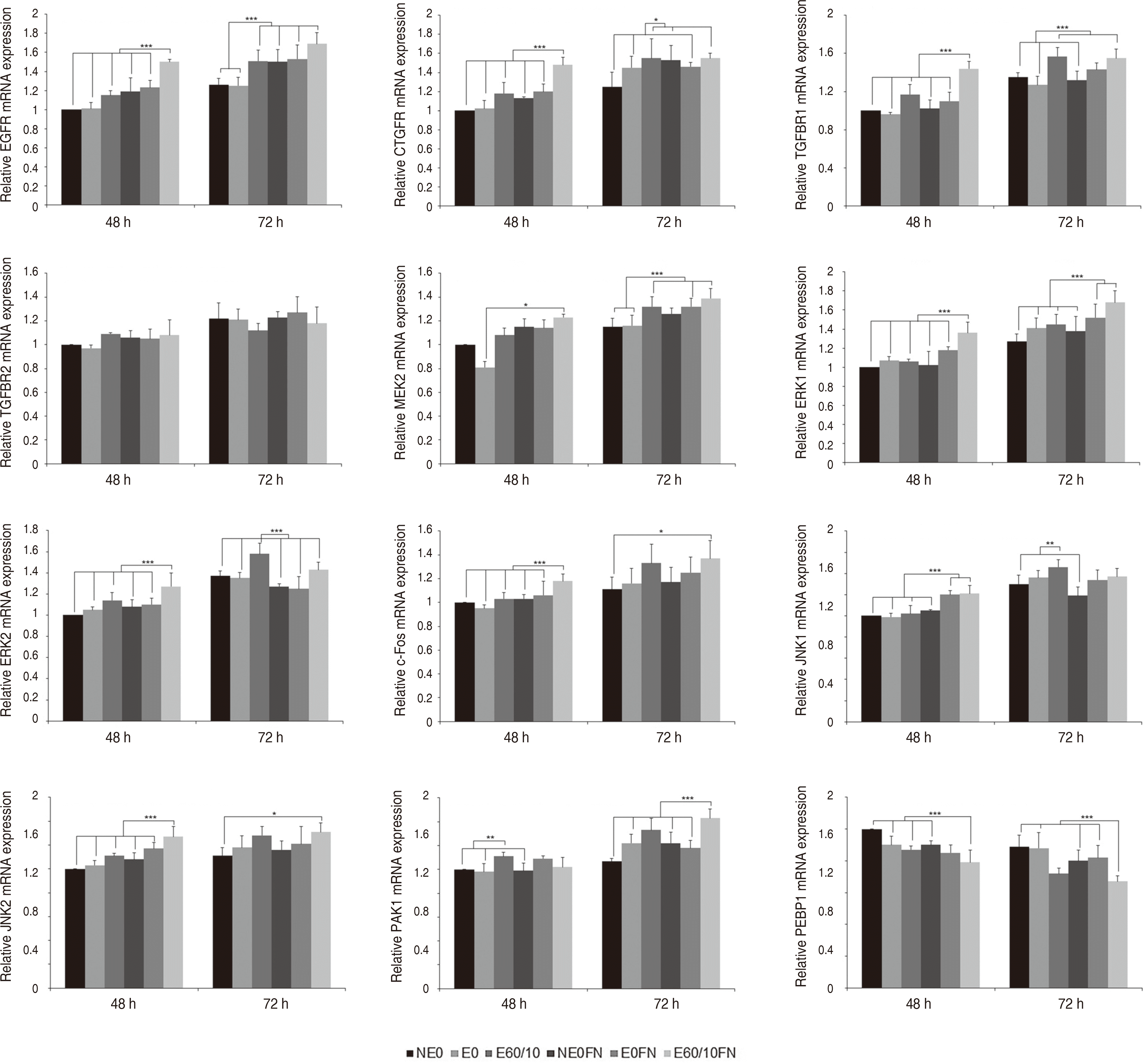
Fig. 4
Expression of CDK2, CDK4, CDK6, Cyclin D1, Cyclin E, p21CIP1, p27KIP1, and c-Myc gene of human gingival fibroblasts cultured on smooth (NE0), acid-etched (E0), microgrooved (E60/10), and fibronectin-immobilized (NE0FN, E0FN, and E60/10FN) titanium substrata after 48 and 72 h of culture using semi quantitative real-time RT-PCR. The relative expression levels were analyzed by normalizing with GAPDH, and are presented as fold changes relative to the control, NE0 at 48 h of culture. One-way ANOVA (n = 5). ***: significant difference (P < 0.001). **: significant difference (P < 0.01). *: significant difference (P < 0.05). Note that only significant differences between the highest gene expression levels and other gene expression levels are presented.
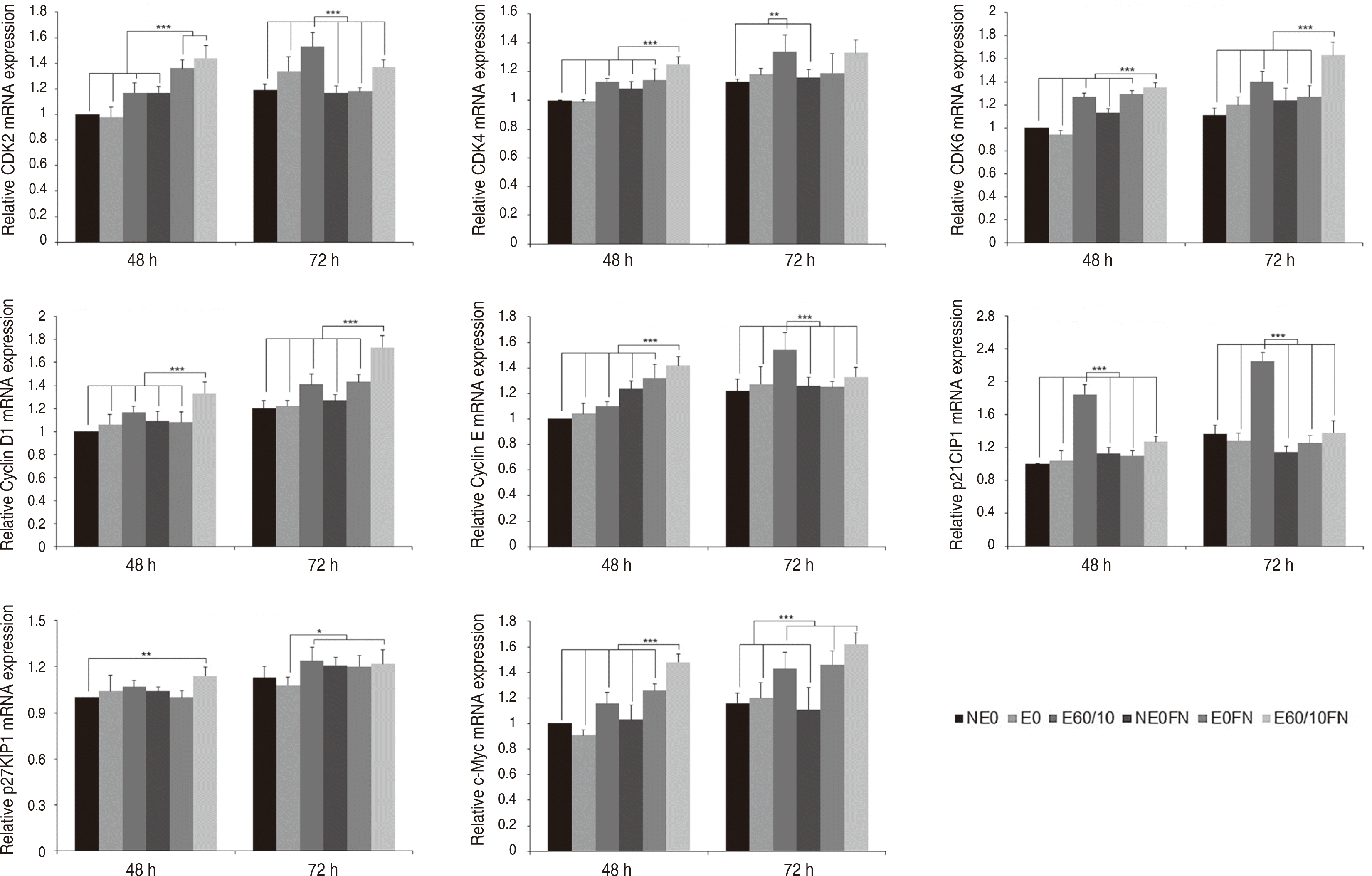
Table 1
Gene-specific primers (Taqman® gene expression assay) used in quantitative real-time PCR




 PDF
PDF Citation
Citation Print
Print



 XML Download
XML Download