INTRODUCTION
조류 인플루엔자 바이러스는 8개의 분절된 RNA 유전자를 보유하고 있으며, 그 중 hemagglutinin (HA) 및 neuraminidase (NA)를 코딩하고 있는 유전자에 따라 16개의 HA아형과 9개의 NA 아형으로 구분된다 (1). 특히 H5 및 H7 아형의 조류 인플루엔자 바이러스는 가금류에서 HA 단백질 분절부위의 염기서열 변이에 의해 닭에서 높은 폐사율을 일으키는 고병원성 조류 인플루엔자 바이러스로 변이될 수 있다 (2). 저병원성 조류 인플루엔자 바이러스가 고병원성 조류 인플루엔자 바이러스로 변이된 사례는 1959년 이후 총 42번이 확인되었으며, 그 중 H5 아형의 조류 인플루엔자 변이는 총 15번 확인되었다 (2). 대부분 발생국가 안에서의 방역 활동을 통해 제거되었으나, 1996년 중국 광동성 거위에서 최초로 발견된 고병원성 H5 바이러스는 현재까지 발생이 지속되고 있다 (3). 특히 이 바이러스는 가금류에서 야생조류로 바이러스가 다시 전파되면서, 야생조류를 통해 유럽, 북미, 아프리카 지역을 포함하여 다양한 국가에 발생을 야기하고 있다 (4, 5).
중국 광동성 거위 유래의 H5형 고병원성 조류 인플루엔자 바이러스는 오랜 기간 변이를 일으키며, 다양한 clade 및 subclade로 진화하고 있다 (3). 중국을 포함하여 고병원성 조류 인플루엔자 발생이 지속되는 나라에서는 H5형 조류인플루엔자 백신을 개발하여 사용하고 있으나, 새로 발생하는 clade는 대부분 기존의 H5 조류인플루엔자 바이러스 백신으로는 방어가 안되는 특징을 보이고 있다 (6, 7, 8). 이러한 바이러스 변이 가능성 및 바이러스 풍토화 예방을 위하여, 대부분의 나라에서는 백신을 사용하지 않고 살처분 정책을 적용하여 바이러스를 박멸하고 있다 (8).
이러한 고병원성 조류 인플루엔자의 변이 및 이에 따른 백신의 효능 감소를 확인하기 위하여, H5형 고병원성 조류 인플루엔자 바이러스의 항원성 분석을 위해 다양한 연구가 진행되고 있다 (6, 9).
특히 항원지도(Antigenic map) 분석을 통해 바이러스 간 항원성을 비교하는 연구는 사람의 계절독감 백신 주 선별에 사용되어 오다가, 최근 조류 인플루엔자 바이러스 분석에 활용되어 지고 있다 (6, 9, 10). 하지만 대부분 연구는 고병원성 조류 인플루엔자 바이러스 항원성 변이 분석에 한정되어져 있고, 저병원성 H5형 조류 인플루엔자 바이러스 항원 분석에는 활용되지 못하였다.
H5형 및 H7형 저병원성 조류 인플루엔자 바이러스는 고병원성 조류 인플루엔자 바이러스로의 변이 가능성 때문에, 고병원성 조류 인플루엔자와 마찬가지로 World Organisation for Animal Health (OIE) 보고 대상 질병에 해당되며 대부분 국가에서 살처분을 통한 박멸정책을 적용하고 있다 (8). 최근에도 멕시코를 포함하여 저병원성 조류 인플루엔자 바이러스가 고병원성 조류 인플루엔자 바이러스로 변이하고 있으며, 멕시코의 경우 저병원성 조류 인플루엔자 바이러스를 이용하여 백신을 개발하여 사용하고 있다 (8, 11). 뿐만 아니라, 2013년 중국에서 발생한 H7N9 처럼 야생조류 유래 저병원성 조류 인플루엔자 바이러스가 인체 감염을 일으키는 바이러스로 변이 할 수도 있으며, 고병원성 조류 인플루엔자 바이러스로 변이 후 인체감염을 일으킬 수도 있다 (12, 13).
따라서 야생조류의 저병원성 H5형 조류 인플루엔자 바이러스의 항원 변이는 미래 발생을 대비하여 지속적으로 감시되어야 한다. 또한, 저병원성 조류 인플루엔자 바이러스의 항원 변이에 대한 자료는 고병원성 조류 인플루엔자 바이러스의 항원 변이 속도 확인을 위한 비교자료로 활용될 수 있다. 본 연구에서는 항원지도 작성을 활용하여 한국에서 분리된 저병원성 및 고병원성 H5형 조류 인플루엔자의 항원성을 비교 분석하였다.
MATERIALS AND METHODS
바이러스 및 항체
한국의 야생조류 분변에서 분리된 10개의 저병원성 H5형 조류 인플루엔자를 본 연구에 활용하였다. 모든 바이러스는 10일령 유정란을 이용하여 3일간 배양하였으며, 저병원성 조류 인플루엔자 바이러스의 경우 최초 시료로부터 3번 종란에서 계대한 후 본 연구에 사용하였다. 본 연구에 사용된 바이러스의 세부 정보는 Table 1과 같다. 고병원성 조류 인플루엔자 바이러스의 경우 기존 reverse genetics 방법을 통해 병원성을 제거한 A/Vietnam/1194/2004 (VNM/04), A/Indonesia/5/2005 (IDN/05), A/chicken/Korea/ES/2003 (ES/03) 및 A/mandarin duck/Korea/K10-483/2010 (Kr/10) 바이러스를 이용하였다.
Table 1.
List of low pathogenic avian influenza viruses used in this study
10종의 저병원성 H5, ES/03 및 Kr/10 바이러스는 건국대학교 자체 예찰 조사에서 분리되었으며, 병원성을 제거한 ES/03 및 Kr/10 바이러스는 기존 백신개발 연구에서 개발되었다 (14). 또한 병원성을 제거한 VNM/04 및 IDN/05 바이러스는 연세대학교에서 건국대학교로 분양한 바이러스를 이용하였다 (14). 10종의 저병원성 H5형 조류 인플루엔자 바이러스에 대한 각각의 항혈청 및 모든 바이러스는 외부 반출 없이 건국대학교 내에서 실험을 진행하였으며, reverse genetics를 통해 병원성이 제거된 바이러스들은 완전한 병원성 제거가 확인되어 BSL-2 실험실에서 실험을 진행하였다.
유전적 상관관계 분석
기존에 보고된 primer를 이용하여 PCR을 통해 HA 유전자를 증폭하였다 (15). 증폭된 HA 유전자는 마크로젠 사(www.macrogen.co.kr)를 통하여 sanger방식으로 염기서열 분석에 활용되었다. 분석된 유전자는 NCBI genbank (https://www.ncbi.nlm.nih.gov/genbank/)에 보고된 다른 유전자와 함께 alignment후 같은 길이가 되도록 양 끝 염기서열을 제거하였다. MEGA 10 프로그램을 이용하여 Maximum-likelihood 방식을 통하 phylogenetic tree 분석을 실시하였으며, HKY 유전자 치환 모델 및 bootstrap 방법이 이용되었다 (16, 17).
RESULTS
유전적 상관관계 분석
Phylogenetic analysis 수행 결과 한국 야생조류에서 분리된 저병원성 H5형 조류 인플루엔자 바이러스는 유라시아 대륙에서 유행중인 다른 H5형 바이러스들과 유사성을 나타내는 것으로 확인되었다 (Fig. 1). 유라시아 대륙의 저병원성 H5형 조류 인플루엔자 바이러스는 중국 광동성 유래 고병원성 H5형 바이러스와는 phylogenetic tree에서 확연히 구분되는 것이 확인되었다. 또한 유라시아 대륙의 저병원성 h5형 바이러스는 bootstrap 값 90 이상으로 확인되는 두개의 그룹으로 나뉘어 지는 것이 확인되었다. 한국에서 분리된 10개의 저병원성 H5형 조류 인플루엔자 바이러스는 이 두개의 그룹에 각각 7개, 3개씩 해당되었다.
Fig. 1
Maximum likelihood phylogenetic tree of the HA gene of H5 subtype. The black circle (●) identifies the genes of low pathogenic avian influenza isolates, and the black triangle (▼) identifies the genes of highly pathogenic avian influenza strains used in this study. The percentages of replicate trees in which the associated taxa clustered together in the bootstrap test (1,000 replicates) are shown next to the branches. The scale bar and branch lengths indicate numbers of nucleotide substitutions per site.
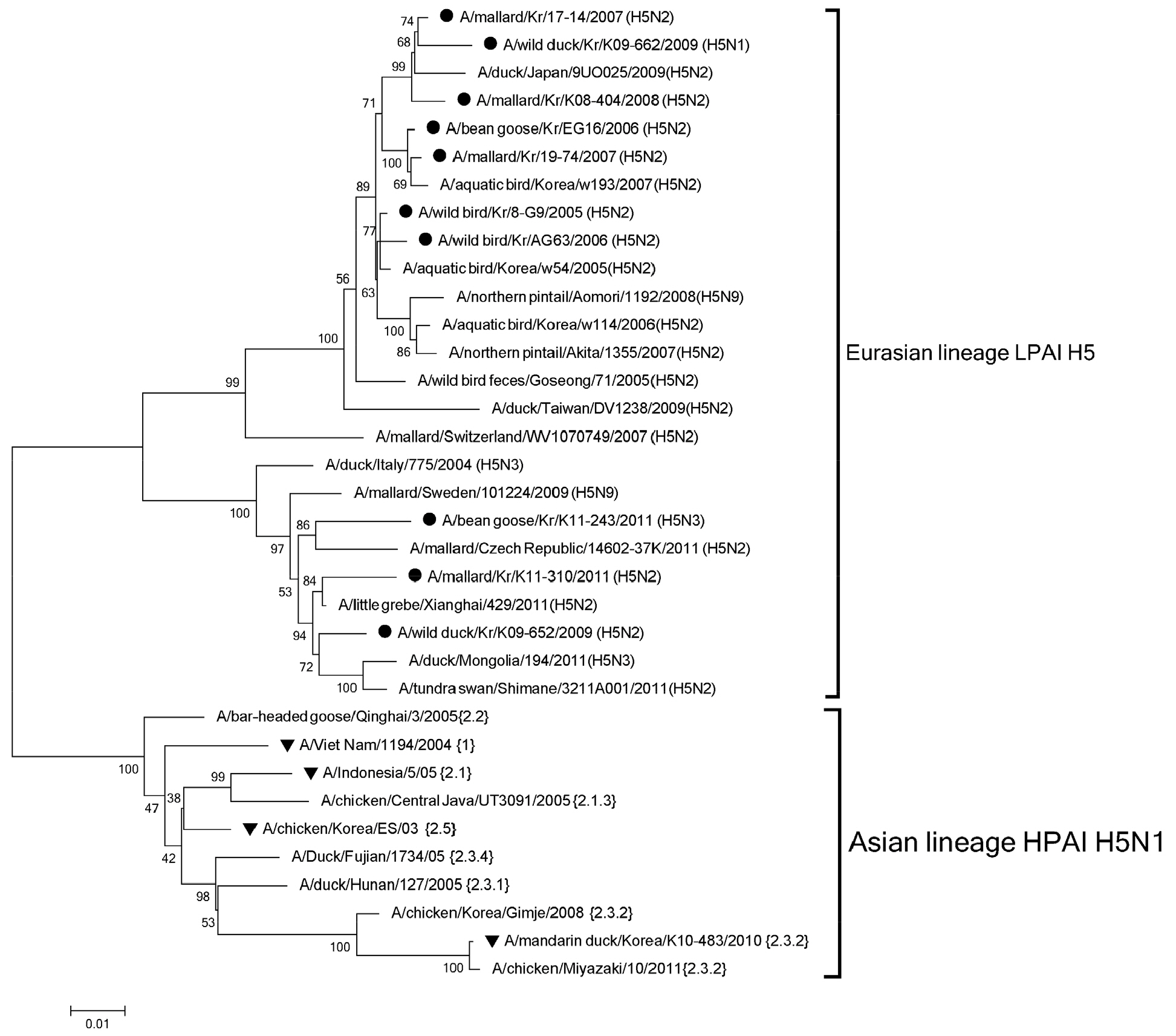
저병원성 H5 바이러스와 고병원성 H5 바이러스와의 유전적 상동성은 87.0%-91.0%로 확인되었으며, 아미노산의 상동성은 88.5%-93.1%로 확인되었다. 특히 2010년 국내에서 분리된 Kr/10 고병원성 조류 인플루엔자 바이러스의 경우 저병원성 H5 바이러스와 가장 낮은 유전자 및 아미노산 상동성을 나타내었다(87.0%-87.6%, 88.5%-89.0%).
항원성 분석
14개의 항원 및 10개의 항체를 활용한 상호간 HI test 및 MN test 결과 저병원성 조류 인플루엔자 바이러스 간에는 상대적으로 높은 항원-항체 반응성을 나타내었으나, 저병원성 조류 인플루엔자 바이러스에 대한 항체는 고병원성 조류인플루엔자 바이러스에 대해 반응성이 낮은 것으로 확인되었다 (Table 2). 특히 IDN/05 (clade 2.1) 및 Kr/10 (clade 2.3.2) 바이러스는 ES/03 (clade 2.5) 및 VNM/04 (clade 1)에 비하여 더 낮은 반응성을 나타내었다.
Table 2.
Hemagglutination inhibition tests and microneutralization assay results between 10 antisera and 14 antigens, including 10 low pathogenic viruses and 4 high pathogenic viruses
| Anti-serum | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 8G9 | AG63 | EG16 | 17-14 | 19-74 | K08-404 | K09-652 | K09-662 | K11-243 | K11-310 | ||
| Antigen | 8G9 | 64a 128b |
64 128 |
32 64 |
128 256 |
32 64 |
32 64 |
32 64 |
64 128 |
16 32 |
8 16 |
| AG63 |
128 64 |
128 64 |
64 32 |
128 128 |
64 64 |
64 64 |
64 64 |
128 128 |
32 32 |
16 16 |
|
| EG16 |
128 64 |
128 128 |
64 32 |
128 128 |
64 64 |
64 64 |
64 32 |
128 64 |
32 32 |
16 16 |
|
| 17-14 |
128 128 |
128 128 |
64 64 |
128 256 |
64 64 |
64 64 |
64 64 |
128 256 |
32 64 |
32 32 |
|
| 19-74 |
64 64 |
128 64 |
32 32 |
64 128 |
32 32 |
32 32 |
32 32 |
64 64 |
16 16 |
16 16 |
|
| K08-404 |
256 256 |
256 256 |
128 64 |
256 256 |
128 128 |
128 128 |
64 128 |
128 256 |
32 64 |
32 32 |
|
| K09-652 |
128 128 |
64 64 |
64 64 |
128 256 |
32 32 |
64 64 |
64 64 |
128 128 |
32 32 |
32 16 |
|
| K09-662 |
128 256 |
128 256 |
64 128 |
128 512 |
32 128 |
64 128 |
64 128 |
128 256 |
32 64 |
16 32 |
|
| K11-243 |
128 256 |
128 256 |
32 128 |
12 8 256 |
32 128 |
32 128 |
32 128 |
64 256 |
32 64 |
16 32 |
|
| K11-310 |
64 128 |
128 128 |
32 64 |
64
25 6 |
32 64 |
32 64 |
32 64 |
32 128 |
16 32 |
16 32 |
|
| ES/03 (2.5c) |
64 128 |
32 128 |
16 32 |
16 32 |
8 16 |
32 64 |
16 32 |
16 64 |
8 32 |
8 16 |
|
| VNM/04 (1) |
32 64 |
8 8 |
4 16 |
8 16 |
8 16 |
8 32 |
8 16 |
16 32 |
4 8 |
4 8 |
|
| IDN/05 (2.1) |
16 32 |
2 4 |
2 4 |
4 8 |
2 4 |
4 32 |
4 8 |
8 16 |
2 4 |
2 8 |
|
| Kr/10 (2.3.2) |
4 16 |
2 8 |
4 16 |
4 16 |
4 16 |
2 16 |
4 16 |
4 16 |
2 16 |
2 4 |
|
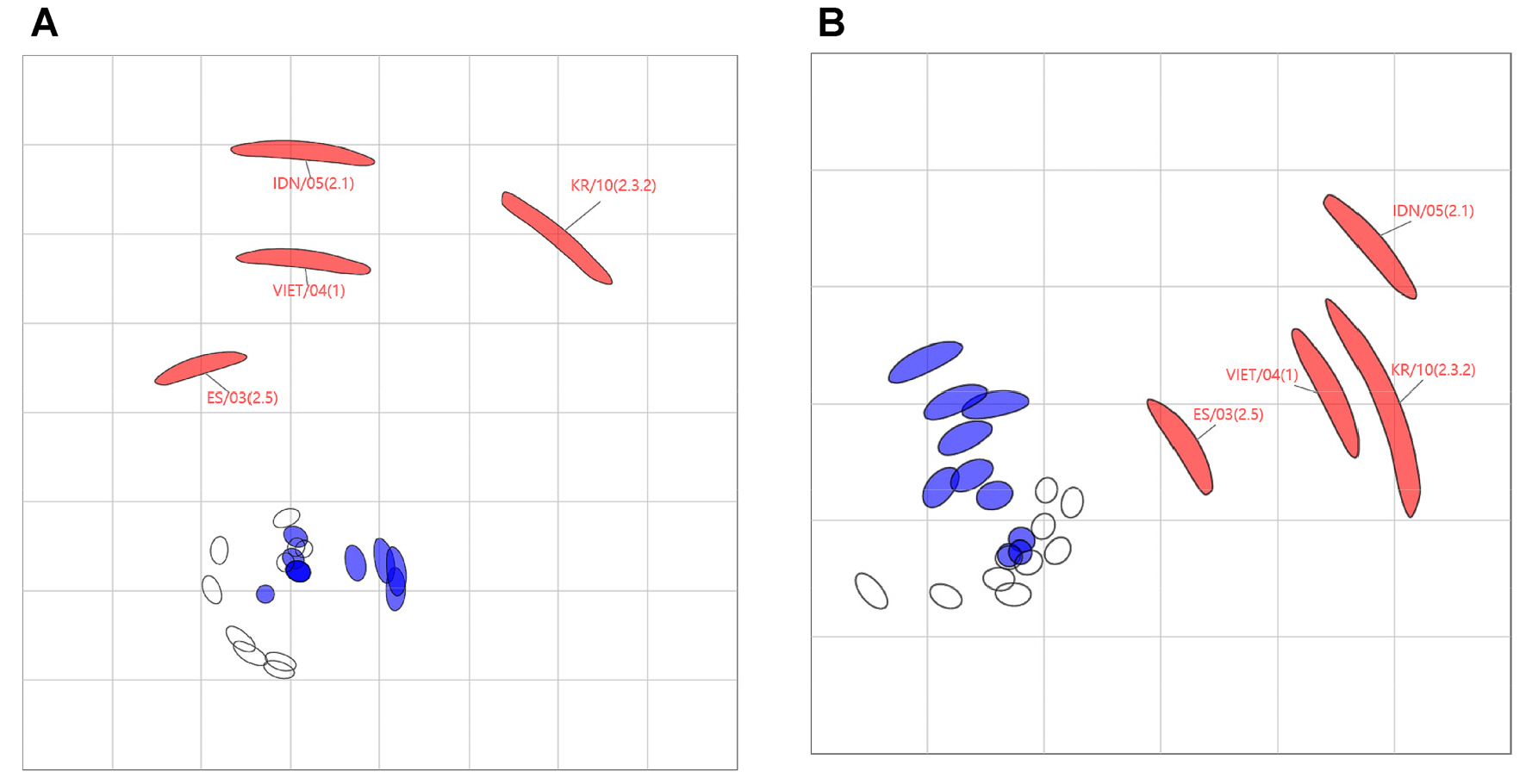
분석결과의 시각화 및 상호간 항원 관계 분석을 위해서 항원 지도를 제작하였다 (Fig. 2). 제작한 항원지도에서 저병원성 조류 인플루엔자 바이러스들은 최대 약 21.5배 이하의 항원성 차이를 나타내는 것으로 확인되었으나, 고병원성 조류 인플루엔자 바이러스는 최소 2배 이상의 항원-항체 반응성 차이를 나타내었다. ES/03 바이러스의 경우 상대적으로 저병원성 조류 인플루엔자 바이러스와 가까운 것으로 확인되었으며, Kr/10 및 IDN/05 바이러스의 경우 저병원성 바이러스와 항원 관계가 먼 것으로 확인되었다.
Fig. 2
Antigenic map of the H5 subtype viruses. The map is based on the results from (A) hemagglutination inhibition tests and (B) microneutralization assay between ten antisera (uncolored open shapes) and 14 antigens, including ten low pathogenic viruses (blue shapes) and four high pathogenic viruses (red shapes). The distance between the shapes represent antigenic distance where one unit (grid) represents a 2-fold change in the serum dilution by HI test. The size and shape represent the statistical confidence in the placement of the strain or antiserum. The antigenic map was constructed using ACMACS software available through https://acmacs-web.antigenic-cartography.org/.
DISCUSSION
바이러스의 항원성 변이는 향후 백신 개발 및 항체 치료제 개발에 큰 영향을 미칠 수 있다 (9, 10). 따라서 향후 동물 및 사람에서 심각한 질병을 일으킬 수 있는 병원체의 항원 변이에 대한 지속적인 모니터링이 필요하다. 특히 조류 인플루엔자 바이러스는 변이율이 높고, 항원성 변이가 빈번하게 발생하는 것으로 알려져 있다 (10). 본 연구에서 유전적으로 다양한 저병원성 및 고병원성 조류 인플루엔자 바이러스 간 항원성을 비교하였으며, 이는 향후 H5형 조류 인플루엔자 발생 시 대응을 위한 기초자료로 활용될 수 있을 것으로 기대된다.
본 연구결과는 저병원성 바이러스는 항원성 변이가 거의 발생하지 않았으나, 고병원성 조류 인플루엔자의 경우 항원 변이가 상대적으로 빠르게 일어난다는 것이 확인되었다. 본 연구에서는 2005년부터 2011년 사이에 분리된 저병원성 조류 인플루엔자 바이러스와, 2003년부터 2010년에 분리된 고병원성 조류 인플루엔자 바이러스가 사용되었다. 저병원성 바이러스는 약 6년간 항원변이가 거의 일어나지 않은 반면, 고병원성 조류 인플루엔자 바이러스는 유사한 기간 내에 더 많은 항원 변이가 발생한 것으로 확인되었다.
고병원성 바이러스의 항원 변이는 백신 사용으로 인한 양성 선택(positive selection)에 의한 것으로 추정된다. 실제 중국, 인도, 인도네시아, 베트남 및 이집트 등 대부분 상재국에서 오랜기간 백신을 사용해오고 있으며, 이들 지역에서 더 많은 바이러스 변이가 확인되고 있다 (8, 20, 21). 또한 저병원성 H7 바이러스가 고병원성 H7으로 변이한 멕시코의 경우 백신 사용 이후 바이러스 변이 속도가 급격하게 증가한 것이 확인되었다 (11). 백신사용으로 인해 생성된 항체가 바이러스의 감염을 완전히 예방하지 못한 경우 항체에 의해 면역 회피 변이 바이러스가 선별될 수 있다 (22). 이러한 양성 선택 및 선별은 바이러스의 변이를 가속화할 뿐 아니라, 대부분 변이가 항원부위에 집중되게 되며, 고병원성 조류 인플루엔자 바이러스가 다양한 항원성을 가지게 하는데 기여한 것으로 추정된다.
이에 반해, 저병원성 H5형 조류 인플루엔자 바이러스는 백신을 사용하고 있지 않고, 대부분 국가의 가금류에서 상재화 되어 있지 않다 (1, 8). 본 연구에서 사용된 저병원성 H5형 조류 인플루엔자 바이러스는 모두 자연숙주인 야생조류에서 분리된 것으로 선택압(selection pressure)를 거의 받지 않았을 것으로 추정된다. 야생조류에서도 자연감염으로 인한 항체 형성이 가능하나, 본 연구에서 이러한 항체가 항원 변이에는 거의 영향을 주지 않는다는 것이 확인되었다. 또한 phylogenetic tree 분석에서 다른 집단을 형성한 바이러스 간에도 항원성이 유사한 것이 확인되었다. 조류 인플루엔자 바이러스의 항원성을 결정하는 부위는 대부분 HA 단백질의 머리 부분에 해당하며 이는 또한 세포의 수용체와 결합하는 부위이다 (23). 본 연구결과 저병원성 바이러스 간 항원성이 유사한 것을 기반으로, 야생조류에서는 이러한 항원 부위 변이가 많이 발생하지 않았을 것으로 추정된다.
HI test에 기반한 항원 지도에 비하여, MN assay에 기반한 항원지도는 바이러스 간 항원성이 더 가까운 것으로 나타났다. HI test는 바이러스의 혈구 응집 억제능만을 확인하나, MN assay는 바이러스 중화능을 확인하는 것으로, HA 단백질의 머리 부분뿐 아니라 다양한 다른 항체에 의한 바이러스 감염 억제 효능이 확인될 수 있어 이러한 결과가 나온 것으로 추정된다. 전체적인 항원지도의 분포 양상은 두 분석 결과 거의 유사하게 확인되었다.
본 연구 결과, 향후 유라시아 대륙의 저병원성 H5형 조류 인플루엔자 바이러스가 상재화 되거나 고병원성으로 변이하는 경우, 다른 저병원성 H5형 조류 인플루엔자 바이러스를 이용하여 백신 및 치료제 개발에 활용할 수 있다는 것이 확인되었다. 하지만 백신은 고병원성 조류 인플루엔자 바이러스처럼 항원 변이를 가속화 시킬 수 있어 적용 여부에는 주의가 필요하다.
저병원성 및 고병원성 H5 바이러스의 상대적인 항원성 변이 분석을 통하여, 고병원성 조류 인플루엔자 바이러스에서 더 다양한 항원 변이가 확인되었다. 본 연구에서 확인하였다. H9N2 바이러스 등 좀 더 다양한 조류 인플루엔자 바이러스 분석을 통해 조류 인플루엔자 바이러스 항원성 변이에 대한 이해를 확장하고 이를 예방하기 위한 방법을 모색해야 할 것이다. 또한 지속적인 항원성 변이 모니터링 시스템 구축을 통해 장기적인 분석이 진행되어야 할 것이다.




 PDF
PDF Citation
Citation Print
Print


 XML Download
XML Download